細菌
| 細菌 | |||
|---|---|---|---|
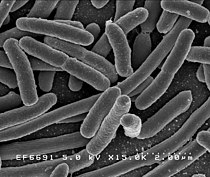 大腸菌(Escherichia coli)
| |||
| 地質時代 | |||
| 太古代(先カンブリア代) - 現代 | |||
| 分類 | |||
| |||
| 学名 | |||
| Bacteria Woese et al. 2024 | |||
| シノニム | |||
| |||
| 和名 | |||
| 細菌/真正細菌 | |||
| 下位分類(門)(2024年7月現在)[1] | |||
|
細菌(さいきん、真正細菌、ラテン語: bacterium、複数形 bacteria、バクテリア)とは、古細菌、真核生物とともに全生物界を三分する、生物の主要な系統(ドメイン)の一つである。語源はギリシャ語の「小さな杖」(βακτήριον)に由来する[2]。細菌は大腸菌、枯草菌、藍色細菌(シアノバクテリア)など様々な系統を含む生物群である。通常1-10 µmほどの微生物であり、球菌や桿菌、螺旋菌など様々な形状が知られている。真核生物と比較した場合、非常に単純な構造を持つ一方で、はるかに多様な代謝系や栄養要求性を示す。細菌を研究する科学分野は微生物学(または細菌学)と呼ばれる。
細菌と古細菌は合わせて原核生物と呼ばれる。核を持たないという点で古細菌と類似するが、古細菌と細菌の分岐は古い。古細菌と比較して、遺伝システムやタンパク質合成系の一部に異なる機構を採用し、ペプチドグリカンより構成される細胞壁や、エステル型脂質より構成される細胞膜を持っているという点からも細菌は古細菌と区別される。1977年までは古細菌は細菌に含まれると考えられていたが、現在では両者はドメインレベルで別の生物とされる。
細菌の生息環境は非常に広く、例えば土壌、淡水・海水、酸性温泉、放射性廃棄物、そして地殻地下生物圏といった極限環境に至るまで、地球上のあらゆる環境(生物圏)に存在している。地球上の全細胞数は5×1030に及ぶと推定されており、その生物量は膨大である。また、その代謝系は非常に多様であり、細菌は光合成や窒素固定、有機物の分解過程など、物質循環において非常に重要な位置を占めている。熱水噴出孔や冷水湧出帯などの環境では、硫化水素やメタンなどの海水中に溶解した化学化合物が細菌によりエネルギーに変換され、近隣環境に生息する様々な生物が必要とする栄養素を供給している。植物や動物と共生・寄生の関係になる細菌系統も多く知られている。地球上に存在する細菌種の大半は、未だ十分に研究がされておらず、その生態や物質循環における役割が不明である。研究報告がなされた細菌種は全体の約2%に過ぎないとも推定され[3]、実験室での培養系が確立していないものが大半である。
腸内細菌や発酵細菌、病原菌など、ヒト(人間)をはじめとする他の生物との関わりも深い。通常、ヒトなどの大型生物は、何百万もの常在菌と共存している。例えば腸内細菌群は、多くの動物において食物の消化過程に欠かすことのできない要素である。ヒト共生細菌の大半は無害であるか、免疫系の保護効果によって無害になっている。多くの細菌、特に腸内細菌は宿主となる動物にとって有益な存在である。共生細菌に限らず、細菌の大半は病気などを引き起こす存在とは考えられていない。
しかし極一部のものは病原細菌として、ヒトや動物の感染症の原因になる。例えばコレラ、梅毒、炭疽菌、ハンセン病、腺ペスト、呼吸器感染症など病原性を持ち感染症を引き起こす細菌が知られている。このような感染症を治療するために、ストレプトマイシンやクロラムフェニコール、テトラサイクリンなど、様々な細菌由来の抗生物質が探索され発見されてきた。抗生物質は細菌感染症の治療や農業で広く使用されている一方、病原性細菌の抗生物質耐性の獲得が社会的な問題となっている。
また、下水処理や流出油の分解、鉱業における金・パラジウム・銅等の金属回収などにも、細菌は広く応用利用されている。食品関係においては、微生物学が展開するはるか以前から、人類はチーズ、納豆、ヨーグルトなどの発酵過程において微生物を利用している。
細菌は対立遺伝子を持たず、遺伝子型がそのまま表現型をとり、世代時間が短く変異体が得られやすく、さらに形質転換系の確立によって遺伝子操作が容易である。このような理由から、近年の分子生物学を中心とした生物学は、細菌を中心に研究が発展してきた。特に大腸菌などは、分子生物学の有用なツールとして現在でも頻繁に使用されている。
呼称
[編集]各言語での呼称は、ラテン語が Bacterium、日本語および中国語が「細菌」である。1828年、クリスチャン・ゴットフリート・エーレンベルクが、顕微鏡で観察した微生物が細い棒状であったため、古代ギリシア語で「小さな杖」を意味する βακτήριον (baktḗrion)から造語し、ラテン語で “Bacterium” と呼んだことに由来する。この複数形が Bacteria である[4][5][6]。日本語の「細菌」の語の発案者は不明であるが、1895年(明治28年)には『細菌学雑誌』が創刊され、19世紀末には既に使われていた[要出典]。
なお、「細菌」には「菌」という漢字が使用されているが、狭義の菌類(真菌)には含まれない。同様に、細菌とは別グループの生物である「古細菌」には細菌という語が使われているが、この記事が説明する狭義の細菌に含まれない。分類学上の「菌類」(Fungi)、「細菌」(Bacteria)、「古細菌」(Archaea)は、別々の独立した生物である。
このほかの呼称としては、真正細菌(Eubacteria)や Monera(モネラ)などがあるが、いずれも古い用語であり、使用頻度は下がっている[要出典]。真正細菌(Eubacteria)は、かつて古細菌が細菌とみなされていた時代に(Archaeabacteria と呼ばれていた)、これと区別するために使用されていた単語である。ただし、現在でもトーマス・キャバリエ=スミスら著名な研究者の一部がこの語を用いている[要出典]。
起源と初期の進化
[編集]
地球上において、細菌は古細菌とともに生命発生の最初期の頃から存在すると考えられている[8][9][10]。ストロマトライトなどの細菌由来と想定される化石が存在しているものの、大部分が単細胞性で極めて小さく、独自の特徴的な形態などを持っていないため、地質学的に細菌の進化史を解明するには多くの困難がある。一方で、現生の細菌がもつゲノム情報を検討することで、細菌の系統学的な進化プロセスが推定されており、細菌と古細菌の分岐は真核生物の誕生よりも前に遡ることが示されている[11]。
細菌と古細菌の共通祖先(最終共通祖先、LUCA)は、35-40億年前頃に生息していた超好熱菌の一種であるとする仮説が出されている[12][13][14]。ただし、それら初期生命体の生息環境が海であったのか陸地であったのかさえ定説は存在しない[15][16]。
細菌は、古細菌とともに真核生物の誕生と進化に深く関与している[17]。例えば、アルファプロテオバクテリア網に属する細菌が、真核生物の祖先となる古細菌内に細胞内共生ののち細胞内器官として取り込まれ、現在の全ての真核生物が持つミトコンドリアやハイドロジェノソームの元となった、というシナリオが考えられている。さらには、ミトコンドリアを既に保持していた一部の真核生物が新たにシアノバクテリアを細胞内に取り込み、今日の藻類や植物が持つ葉緑体を形成したと考えられている。これは一次共生(primary endosymbiosis)として知られている[18]。
生育環境
[編集]細菌は、通常の土壌や湖沼はもちろん、地殻、大気圏、熱水鉱床、水深1万m以上の深海底、南極の氷床といった、生物圏とされている地球上のほぼ全ての環境に分布する[19][20]。地球上には、約2×1030細胞もの細菌が存在していると見積もられている[21]。
細菌は湖や海、北極の氷、さらには地熱温泉[22]などでも豊富に見られ、温泉環境などでは硫化水素やメタンなどの溶解した化合物をエネルギーに変換することで、生命を維持するために必要な栄養素を作り出している[23]。特に土壌は細菌が非常に豊富に存在する環境であり、数グラムに約1億個の細菌が含まれている[24]。細菌は有毒な廃棄物を分解し、栄養素をリサイクルする存在として、土壌生態学の観点からも不可欠な存在である。
細菌は大気中にも見られ、1立方メートルの空気中には約1億個の細菌細胞が存在している[24]。海洋には約3×1026細胞もの細菌が存在しており、これらの一部が行う光合成によって、人間が呼吸する酸素の最大50%が供給されていると見積もられている[24]。
一部の細菌は芽胞という乾燥に強い形態を取ることも知られている[25]。
また多細胞生物体内部や表面にも多数の細菌が付着生育しており、共生関係にある。ただし、健康な生物体の血液中、筋肉、骨格など消化管以外の臓器からはほとんど検出されない[要出典]。消化管においては、食物の分解プロセスの一部を細菌が担っている。共生の例は、ルーメンやマメ科植物の根圏における窒素固定菌の共生などに見ることができる[要出典]。また、一部の昆虫類では菌細胞と呼ばれる共生細菌を維持するための細胞を分化させ、その細胞質内に細菌を共生させるが、これら細胞質内共生細菌のなかには、カルソネラ・ルディアイ(Candidatus Carsonella ruddii)のように宿主の細胞外で生存あるいは増殖が出来ないものがある[要出典]。
バイオマスの観点では、細菌は植物を超える存在である[26]。土壌では、4000m2あたり2トンの微生物(真菌、古細菌を含む)が含まれていると見積もられている[要出典]。また海洋においては、栄養状態にかかわらず1ミリリットル(mL)あたり50細胞程度の細菌が存在しており(沿岸や生物の死体周辺ではmLあたり105細胞以上生息している)、海洋だけでも地上の真核生物量をはるかに凌駕する計算がなされている[要出典]。
形状・大きさ
[編集]

細菌は様々な細胞形態や配置を示す。一般に、大きさはおおむね0.5-5 µm程度であり、古細菌と同規模で真核生物よりは一桁小さい。桿菌の中では、長いものは15 µmほどになる。さらに肉眼でも見ることができるサイズになるものもあり、例えばThiomargarita namibiensisは500 µmほどに[28]、Epulopiscium fishelsoniは700 µm程度にも達する[29]。最大で2 cmにもなる細菌も発見されている[30]。逆に最小のバクテリアとしては、わずか0.3 µmのマイコプラズマ属の種が知られている[31]。これよりも小さい細菌が存在する可能性も示唆されているが、支持されていない[32]。
細菌の細胞は、藍藻類など一部を除いて、多くの場合種同士で見分けが付かない。古細菌の細胞とも酷似している。ただしドメイン全体で見ると、らせん菌など様々な形態が存在する。桿菌ではしばしば細胞壁が連なって長大な糸状になる。一部は多細胞性を示し、群体や菌糸を形成する。なかでも粘液細菌は細胞性粘菌とよく似た生活環を持つことで知られる。大半の細菌種は、球状の球菌(ギリシャ語のkókkosから、coccusと呼ばれる)や棒状の桿菌(ラテン語のbaculusから、bacillusと呼ばれる)のいずれの形態をとる[33]。他のものとしては、ビブリオ属などの細菌はわずかに湾曲した棒状の形をとる他、spirillaはらせん状の形態をもち、特にスピロヘータはしっかりと巻かれた螺旋状の形態を取る。また、星型など、他にも珍しい形状を持つ細菌種が知られている[34]。このような形状の多様性は、細菌の細胞壁と細胞骨格によって決定されており、それぞれの形状は細菌が栄養素を獲得したり、表面に付着し、液体を泳ぎ、捕食者から逃れたりする能力などに影響を与える可能性があるため、生態的にも重要である[35][36]。

多くの細菌種は単一の細胞として存在しているが、例外も知られている。例えばナイセリア属(Neisseria)は二倍体(ペア)を形成し、連鎖球菌はその名の通り鎖状の構造をとり、ブドウ球菌も名の通りブドウの房のようなクラスター構造を取る。他にも、放線菌に見られるような細長いフィラメント状になったり、粘液細菌種のように凝集体を構築したり、ストレプトマイセス属種のように複雑な菌糸を出したりなど、より大きな多細胞構造を形成するための機能をもっているものも知られている[38]。このような多細胞構造は、しばしば特定の条件でのみ見られることがある。たとえば粘液細菌は、生育環境中のアミノ酸が不足するとクオラムセンシングと呼ばれるプロセスを通じて周囲の細胞を認識し、互いに向かい合うように移動し、約100,000個の細菌細胞が凝集して長さ最大500マイクロメートル程度の子実体を形成する[39]。これらの子実体では、凝集した細胞は別々の機能を担う。たとえば、細胞の約10分の1が子実体の上部に移動し、乾燥やその他の悪環境条件に対してより耐性のある粘液胞子と呼ばれる特殊な休眠状態に分化する[40]。
細菌はしばしば何かしらの物質の表面に付着し、バイオフィルムと呼ばれる密集した凝集体を形成して大きな形成物(微生物マット)を形成する[41][42]。バイオフィルムは数マイクロメートルから最大0.5メートル程度までの厚さを持ち、複数の種類の細菌や原生生物、古細菌が混合している場合がある。バイオフィルムに生息する細菌は、細胞と細胞外成分が複雑に絡み合い、マイクロコロニーなどの二次構造を形成している。この構造を介して、栄養素をより良い形で拡散するようなネットワークを形成している[43][44]。土壌や植物の表面などの自然環境では、細菌の大部分はバイオフィルムの表面に結合している[45]。臨床分野においても、バイオフィルムは、例えば慢性的な細菌感染症や人体に埋め込まれた医療機器を介した感染症において良く見られる。バイオフィルムの内部は外部刺激から保護されている状態であるため、単独で存在する細菌細胞と比べて殺菌することがはるかに困難である[46]。
細胞構造
[編集]
細菌の細胞は、鞭毛、線毛、莢膜、細胞壁、ペリプラズム、細胞膜、細胞質などから構成されており、主にリン脂質からできている細胞膜に囲まれている。この膜は細胞の内容物を囲み、細胞内細胞質に栄養素やタンパク質、その他の必須成分を保持するためのバリアとして機能する[47]。真核細胞とは異なり、一般的に細菌は核やミトコンドリア、葉緑体および他の細胞小器官など、真核細胞に存在するような大きな膜結合組織を欠いている[48]。ただし例外として、一部の細菌はカルボキシソームのような、細胞質内にタンパク質に結合した細胞小器官を持っている[49][50][51]。さらに、細菌は、細胞内のタンパク質と核酸の局在を制御し、細胞分裂を駆動するための多成分から成る細胞骨格を持っている[52][53][54]。
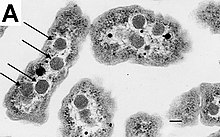
エネルギー生成などの多くの重要な生化学反応は、膜全体の濃度勾配に基づいて発生し、バッテリーのように電気化学ポテンシャルを生み出す。一般的な細菌では電子伝達などの反応は細胞質と細胞の外側やペリプラズムとの間で細胞膜を横切るようにして発生する[55]。多くの光合成細菌では、原形質膜は高度に折りたたまれており、細胞の大部分が集光膜の層で満たされている[56]。これらの集光性複合体は、緑色硫黄細菌のクロロソームと呼ばれる脂質で囲まれた構造を形成することもある[57]。
細菌は通常、膜で閉ざされた核のような構造物を持たない。DNAなどの遺伝物質は単一の環状細菌染色体であり、細胞質の中で核様体と呼ばれる不規則な形状を取っている[58]。核様体には、染色体とそれに関連するタンパク質およびRNAが含まれている。他のすべての生物と同様に、細菌にはタンパク質を生成するためのリボソームが含まれているが、細菌のリボソームの構造は真核生物や古細菌の構造とは異なっている[59]。
一部の細菌は、グリコーゲン[60]、ポリリン酸塩[61]、硫黄[62]、またはポリヒドロキシアルカノエート[63]などの細胞内栄養素貯蔵顆粒を生成する(例えば、ポリリン酸蓄積細菌)。光合成シアノバクテリアなどの細菌は、細胞質に液胞を作り、これを利用してさまざまな光強度と栄養レベルの水層に上下に移動できるように浮力を調整している[64]。
細胞膜外構造
[編集]細胞膜の外周には細胞壁がある。細菌の細胞壁はペプチドグリカン(ムレイン)でできており、D-アミノ酸を含むペプチドによって架橋された多糖鎖から作られている[65]。これは、細胞壁が主にセルロースからできている植物や、キチンでできている菌類とは異なる特徴である[66]。また、ペプチドグリカンを含まない古細菌の細胞壁とも異なる特徴である。細胞壁は多くの細菌にとって生存に不可欠である。抗生物質の一種であるペニシリン(ペニシリウムと呼ばれる真菌によって産生される)は、ペプチドグリカンの合成段階を阻害することによって細菌を殺すことができる[66]。
細菌は、細胞壁がグラム染色で染色されるタイプとされないタイプの2種類に大きく分類することができる。それぞれのタイプの細菌グループは、グラム陽性菌とグラム陰性菌と呼ばれ、この特徴は細菌種を分類するために利用されている[67]。
グラム陽性菌は、ペプチドグリカンとタイコ酸から成る層を複数含む、厚い細胞壁を持っている。対照的にグラム陰性菌は、リポ多糖とリポタンパク質を含む2番めの脂質膜(外膜)と内膜とも呼ばれる細胞質膜の間に囲まれたペリプラズム(空間)と呼ばれる間隙に、数層の薄いペプチドグリカンを持つ[25]。大半の細菌はグラム陰性であり、ファーミキューテスと放線菌(以前はそれぞれ低GCグラム陽性細菌と高GCグラム陽性細菌と呼ばれていた)のみがグラム陽性細菌である[68]。細胞壁の構造の違いにより、抗生物質感受性に違いが出ることが知られている。たとえばバンコマイシンはグラム陽性菌のみを殺すことができインフルエンザ菌や緑膿菌などのグラム陰性病原菌に対しては効果がない[69]。また、一部の細菌は、古典的なグラム陽性菌でもグラム陰性菌でもない細胞壁構造を持っている。これには、グラム陽性菌のように厚いペプチドグリカン細胞壁を持ち、同時に脂質からなる2番目の外層も持つ、マイコバクテリアなどの臨床的に重要な細菌が含まれている[70]。

多くの細菌では、堅く配列されたタンパク質分子のS層が細胞の外側を覆っている[71]。この層は、細胞表面を化学的および物理的に保護し、高分子の拡散バリアとして機能している。S層は多様な機能を持ち、例えばカンピロバクターでは病原性因子として作用し、バチルス・ステアロサーモフィラス(Bacillus stearothermophilus)では表面酵素を含んでいることが知られている[72][73]。
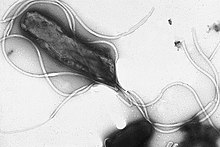
鞭毛は堅いタンパク質構造で、直径は約20ナノメートル、最大20マイクロメートルになる。細菌の運動(遊泳)に使用される。フラジェリンというタンパク質が重合した鞭毛は螺旋状の繊維であり、細胞膜を横切る電気化学的勾配に沿って引き起こされるイオンの移動(水素イオン濃度勾配やナトリウムイオン濃度勾配)に伴うエネルギーによって駆動される[74]。古細菌の鞭毛と見た目は酷似するが、その起源と構造は異なると考えられている[要出典]。
鞭毛よりも小型の繊維構造として、線毛がある。線毛(「付着線毛」と呼ばれることもある)は、ピリンというタンパク質が主要構成分の細いフィラメントで、通常は直径2〜10ナノメートル、長さは最大数マイクロメートル程度である。それらは細胞の表面全体に分布しており、電子顕微鏡で見ると細い毛のように見える[75][25]。線毛は、固体表面または他の細胞への付着に関与していると考えられており、いくつかの細菌性病原体の病原性に不可欠である[76]。細胞から突起している、繊毛よりも若干大きいような線毛は、細胞接合を通じて細胞間で遺伝物質を転送することができるような繊毛である。これは共役線毛又は性線毛と呼ばれる[77]。また、タイプIV線毛と呼ばれる繊毛では、細胞の運動性を作り出すこともできる[78]。
グリコカリックスは多くの細菌で見られ、細胞を取り囲むように生成される[79]。構造化されていない無秩序な粘液層による細胞外高分子物質から、高度に構造化された莢膜まで、多様な複雑さの構造が見られる。これらの構造は、マクロファージ(ヒト免疫系の一部)などの真核細胞による飲み込みから細胞の保護に役立つ[80]。それらはまた、抗原として作用し、細胞認識に関与するだけでなく、表面への付着やバイオフィルム形成に寄与する[81]。
このような細胞外構造の形成には、分泌システムが大きく関係している。分泌システムはタンパク質を細胞質からペリプラズムまたは細胞周辺の環境に移動させる機能を持つ。多くの種類の分泌システムが知られており、これらの構造は病原体の病原性に不可欠であることが多いため、集中的に研究されている[82]。
細胞膜内構造
[編集]
細胞膜は真核生物と同じくsn-グリセロール3-リン酸に脂肪酸が結合したエステル型脂質であり、sn-グリセロール1-リン酸にイソプレノイドアルコールが結合している古細菌とは明確に区別される(古細菌の項も参照)[83]。細胞膜には電子伝達系や各種輸送体、各種センサーなどに関連するタンパク質が分布している[要出典]。内部構造は真核生物の様な明瞭な単位膜系はあまりないが、種によってはチラコイド、DNAを包む核膜様構造が見られることもある(PVCグループの一部)[84]。DNAはHU[85]と呼ばれるタンパク質と結合して核様態という形で凝集しているが、真核生物や古細菌の様にヒストンに巻きついてクロマチン構造をとることはない[要出典]。DNAは環状一分子が一般的だが、稀に直線状のDNAを持つ細菌や、複数のDNAを持つ細菌もいる[要出典]。
内生胞子
[編集]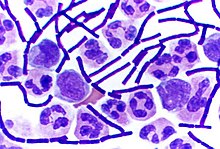
バチルス、クロストリジウム、Sporohalobacter、Anaerobacter、Heliobacteriumなどのグラム陽性菌のいくつかの属は、内生胞子(芽胞、endospore)と呼ばれる非常に耐性のある休眠構造を形成することがある[87]。内生胞子は細胞の細胞質内で発達する。一般的に、各細胞ごとに単一の内生胞子が発生する[88]。各内生胞子は皮質層に囲まれ、ペプチドグリカンや様々なタンパク質で構成される多層の堅いコートで保護されたDNAとリボソームのコアを含んでいる[88]。
内生胞子からは代謝活動は検出されず、高レベルの紫外線やガンマ線、洗剤、消毒剤、熱、凍結、圧力、乾燥などの極端な物理的および化学的ストレスに耐えることができる[89]。この休眠状態において、これらの生物は、何百万年も「生存」し続けることができる[90][91][92]。さらに、内生胞子は宇宙空間の真空や放射線にも耐えることができるため、細菌は宇宙ダストや流星物質、小惑星、彗星、プラネトイド、有向パンスペルミアなどを通じて、宇宙空間中を移動し分散することも可能なのではないかと考えられている[93][94]。内生胞子を形成する細菌にはまた、疾患引き起こすものが知られている。例えば炭疽症は吸入された炭疽菌(Bacillus anthracis)の内生胞子によって引き起こされることがある。破傷風は破傷風菌の芽胞が原因で引き起こされることがあり、これと類似してボツリヌス症も芽胞から成長した細胞が分泌する毒素によって引き起こされる[95]。医療現場で問題となるクロストリジウム・ディフィシル感染症も胞子形成細菌によって引き起こされる場合がある[96]。
代謝と物質循環
[編集]バクテリアは非常に多種多様な代謝を示す[97]。細菌のグループ内の代謝特性の分布は、伝統的に細菌の分類法を定義する際に利用されてきた。ただしこれらの特性は、現在主流となっている遺伝学的な系統分類法とは対応がつかないものも多い[98]。細菌の代謝はエネルギー源、電子供与体、および成長に使用される炭素源、という3つの主要な基準に基づいた栄養グループに分類される[99]。それぞれの資源としてどのようなものを利用できるかによって以下のような分類がある。(詳細は「栄養的分類」参照)
- エネルギー源
- 炭素源
これらの、エネルギー源および炭素源の組み合わせによって、多くの生物の栄養要求性を説明できる。動物は主として有機物を酸化してエネルギーを得る化学合成従属栄養生物であり、植物は光エネルギーにて二酸化炭素を還元して固定する光合成独立栄養生物である。しかしながら微生物には、これら以外にも光合成従属栄養性と化学合成独立栄養性を示す生物群がいる。この二つの特徴ある生物群のうち、化学合成独立栄養性を示すものについては物質循環の中でも重要な役割を担っている。また硫黄酸化細菌、水素細菌などは、太陽エネルギーに依存しない生態系である深海熱水孔や地下生物圏での一次生産者の役割を果たしていると考えられている[要出典]。
細菌は、太陽光から光合成を通じて得られたエネルギーを利用するもの(光栄養生物;phototrophy)や、化学化合物を酸化反応によってエネルギーを獲得するもの(化学合成生物;chemotrophy)が含まれる[100]。化学合成生物は、酸化還元反応により特定の電子供与体から末端電子受容体に電子を移動させることにより、エネルギー源として化学化合物を利用している。化学栄養生物は、電子を伝達するために利用している化合物の種類によって、さらに細かく分類される。例えば電子源として水素や一酸化炭素、アンモニアなどの無機化合物を使用する細菌はリソトロフ(lithotrophs)と呼ばれ、有機化合物を利用するものはオルガノトロフ(organotrophs)と呼ばれる[100]。電子を受け取るために使用される化合物もまた、細菌の分類にも利用されている。例えば好気性生物と呼ばれるグループは末端電子受容体として酸素を利用し、嫌気性生物は硝酸塩、硫酸塩、二酸化炭素などの他の化合物を使用する[100]。
有機化合物から炭素を取得し細胞生育に利用する細菌グループは、従属栄養と呼ばれる。一方で、シアノバクテリアや一部の紅色細菌などの細菌は独立栄養性であり、二酸化炭素を固定することで細胞生育に利用する炭素を獲得する[101]。特殊な環境において見られるメタノトロフと呼ばれるグループでは、ガス状のメタンを炭素源として使用し、かつ電子供与体として活用している[102]。
| 栄養タイプ | エネルギー源 | 炭素源 | 例 |
|---|---|---|---|
| 光合成生物 | 日光 | 有機化合物(光合成従属栄養生物)または炭素固定(光合成従属栄養生物) | シアノバクテリア、緑色硫黄細菌、クロロフレクサス菌、紅色細菌 |
| リソトロフ | 無機化合物 | 有機化合物(リソヘテロトロフ)または炭素固定(リソオートトロフ) | サーモデスルフォバクテリア、ヒドロゲノフィラス、ニトロスピラ |
| 有機栄養素 | 有機化合物 | 有機化合物(化学ヘテロトロフ)または炭素固定(化学オートトロフ) | Bacillus、Clostridium、Enterobacteriaceaeなど |
細菌の代謝は、生態学的安定性を与えるとともに、人間社会にも役立っている。例えば、窒素固定菌(diazotrophs)は、空気中に安定して存在している窒素をニトロゲナーゼを利用して窒素固定する機能を持つ[103]。この環境的に重要な特性を持つような細菌種は、上記の表中のほぼすべての代謝タイプで知られている[104]。窒素固定の機能は、脱窒や硫酸塩還元、酢酸生成といった生態学的に重要な下流のプロセスにつながる[105]。また、窒素はタンパク質のアミノ基に含まれるなど生物体の構成要素として非常に重要である。
細菌の代謝過程は、汚染に対する生物学的反応においても重要である。たとえば硫酸塩還元細菌は、環境中での毒性の高い形態の水銀(メチル水銀およびジメチル水銀)の生成に大きく関与している[106]。非呼吸性嫌気性菌は発酵を利用してエネルギーを獲得し、代謝副産物(醸造中のエタノールなど)を廃棄物として分泌する。通性嫌気性菌は、自分自身がいる環境条件に応じて、発酵と異なる末端電子受容体を切り替えることができる[107]。
細菌は生物量としても真核生物を凌駕しており、またその呼吸活性においても同様で、多細胞生物体と細菌1gの呼吸活性を比較すると細菌のほうが数百倍大きいと言われている[要出典]。肥沃な土壌4000m2あたりの細菌の呼吸活性は数万人の人間に等しいとされる[要出典]。これは細胞が小さく体積あたりの呼吸活性を示す表面積の割合が大きいこと、世代時間が短いことがその要因であろう[要出典]。呼吸速度(炭素、水素、酸素の循環)のみならず、生物を構成している窒素、硫黄の地球全体の物質循環に寄与しているが、後者の多くは酸素を嫌う嫌気性呼吸を伴う[要出典]。
硫黄循環
[編集]硫黄は主に地殻中に豊富に存在し、元素状硫黄は不溶性だが、これも光反応や高熱により硫化水素や硫酸イオンとして自然界に存在する。これを有機物の形で取り入れ、再び水溶性の硫酸塩や硫化水素として排出していく過程を硫黄循環と呼ぶ。有機物中に存在する硫黄は反応性が高く重要なアミノ酸に含まれている(メチオニン、システインなど)。硫酸塩のみが植物によって同化されるが、有機物態硫黄の分解(最終産物は硫化水素)、硫黄酸化(硫化水素から硫酸塩に戻す)、硫酸還元(硫酸塩を異化的に還元する)などは細菌に特有な代謝系である(古細菌にもこのような代謝系を有するものが見つかっている)[要出典]。
成長と増殖
[編集]
多細胞生物とは異なり、単細胞生物では細胞サイズの増加(細胞増殖)と細胞分裂は密接に関連している。細菌細胞は一定のサイズに成長し、その後、無性生殖の一形態である二分裂によって細胞数を増加させる[108]。最適な条件下では細菌は非常に急速に分裂増殖し、ある種の細菌では17分ごとに2倍のスピードで増殖することが知られている[109]。細胞分裂では、2つの同一のクローン娘細胞が生成される。一部の細菌はより複雑な生殖構造を形成し、新しく形成された娘細胞を分散させる。例えば、粘液細菌による子実体の形成や、ストレプトマイセス種による気中菌糸の形成、または出芽などが挙げられる。出芽には、細胞が突起を形成し、それが壊れて娘細胞を生成する形態も知られている[110]。また、同時に3つ以上に分裂する場合や、出芽によって増えるもの、接合してDNAの一部を交換するもの、芽胞などを形成するものが存在する[要出典]。
増殖に際してはDNA複製が行われる。DNA複製は真核生物、細菌で異なる点がある(古細菌ではよく分かっていないが真核生物に類似すると考えられている[要出典])。細菌では大腸菌で最もDNA複製機構の研究が進んでいる[要出典]。複製はDNA上に一箇所存在する複製開始点から開始され、双方向へ複製が進んでいく。
実験室では、細菌は通常、固体または液体の培地を利用して培養する[111]。寒天プレートなどの固体培地は、細菌株の純粋な培養物を分離するために使用される。一方で液体培地は、大量の細胞が必要となる場合に利用される。液体培地での培養では細菌細胞が均一に懸濁されるため、その中から単一の細菌種を分離することは困難である、培養物を簡単に分割したり移動させることができます。選択培地(特定の栄養素を追加したり不足させたりしている培地や、抗生物質などが添加されている培地)を使用すると、特定の機能を持つ生物種だけを選択的に培養させることができる[112]。
実験室においては多くの場合、非常に富栄養な培地を利用して大量の細胞を安価かつ迅速に生産するように培養することが一般的である[111]。しかしながら本来の自然環境では栄養素は限られており、細菌が無期限に繁殖し続けることができない。この栄養制限は、さまざまな成長戦略の進化をもたらしてきており、例えばR-K選択説などが有名である。夏期に湖で頻繁に発生する藻類(およびシアノバクテリア)の異常発生などに見られるように、環境中で利用可能な栄養素が増加することで、一部の生物は非常に急速に成長することがある[113]。また別の戦略として、放線菌などに見られるように複数の抗生物質を生産などして、競合する微生物の成長を阻害する戦略で過酷な環境に適応するものもいる[114]。自然界では多くの微生物は、栄養素の供給を増やし環境ストレスから保護することができるようなコミュニティ(バイオフィルムなど)に生息している[115]。このような関係は、特定の細菌系統において生育に不可欠な要素であることがあり、栄養共生(syntrophy)と呼ばれる[116]。
細菌の増殖は4つの段階をたどる。細菌集団が最初に高栄養環境に晒されると、細胞はその新しい環境に適応する必要がある。そのため、成長の最初の段階は遅滞期であり、これは細胞が高栄養環境に適応し、急速な成長の準備をしているときのゆっくりとした成長期間であるとみなせる。遅滞期は、急速な成長に必要なタンパク質が生成されるため、生合成速度が高まる[117][118]。成長の第2段階は対数増殖段階であり、指数増殖段階とも呼ばれる。対数期は急速な指数関数的成長によって特徴づけられる。この段階で細胞が成長する速度は成長速度(k)と呼ばれ、細胞が2倍になるのにかかる時間は生成時間(g)と呼ばれる。対数期の間、栄養素が枯渇し成長に制限がかかり始めるまで、栄養素は最大速度で代謝され続ける。成長の第3段階は定常期であり、栄養素の枯渇によって引き起こされる。細胞は代謝活性を低下させ、必須ではない細胞タンパク質を消費してゆく。定常期は急速な成長からストレス反応への状態移行であり、DNA修復、抗酸化代謝、栄養素輸送に関与する遺伝子の発現が増加する[119]。最終段階は、細菌が全ての栄養素を使い果たして死ぬ段階である[120]。
ゲノムと遺伝子
[編集]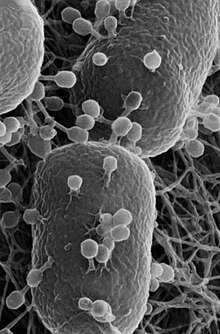
ほとんどの細菌は単一の環状染色体を持っており、そのサイズは、内共生細菌Carsonella ruddiiではわずか160,000塩基対[122]であるのに対し、土壌性細菌Sorangium cellulosumでは12,200,000塩基対(12.2 Mbp)と、さまざまである[123]。また染色体の形と数にも例外が知られており、たとえば一部のストレプトマイセス属とボレリア属の種は単一の線形染色体を持ち[124][125]、一部のビブリオ属種は複数の染色体を持っている[126]。細菌はまた、プラスミドなどのDNAの小さな染色体外分子を持ち、ここに抗生物質耐性、代謝能力、病原性因子などの様々な機能遺伝子を含むことがある[127]。
細菌ゲノムは通常、数百から数千の遺伝子をコードしている。細菌ゲノムにおいては通常、遺伝子は単純に連続してDNA状に分布しているが、まれに異なるタイプのイントロンが存在するものもある[128]。
細菌は無性生物であり、細胞分裂の際には親のゲノムと同一のコピーを継承するクローン体である。
しかし、全ての細菌は、遺伝子組換えや突然変異によって遺伝物質DNAに変化が引き起こされ、その変異が選択されることによって進化してゆく。突然変異は、DNAの複製中に生じたエラーや変異原物質(例えば紫外線や放射線など)への曝露によって生じる。突然変異率は、細菌の種類によって大きく異なり、また単一細菌のクローン内であっても大きく異なる[129]。細菌ゲノムの遺伝的変化は、複製中のランダムな突然変異以外にも、ストレス指向性の突然変異からも生じ、この場合、特定の成長制限プロセスに関与する遺伝子の突然変異率が高くなる[130]。
一部の細菌は、細胞間で遺伝物質を移動させる。これには、主に3つの方法が知られている。
- 1つ目は形質転換と呼ばれるプロセスで、細胞外の外因性DNAを取り込む仕組みである[131]。多くの細菌はこの取り込み機能を持っているが、DNAを取り込むためには化学的な誘導が必要となる細菌もいる[132]。自然界でのDNA取り込み能力の発達は、環境からのストレスの多さと関連しており、細胞のDNA損傷の修復を促進するための適応であると考えられている[133]。
- 2番めは形質導入と呼ばれるプロセスであり、これはバクテリオファージの感染によって外来DNAの遺伝物質が細胞内の染色体に導入されるものである。非常に多様なバクテリオファージが存在することが知られており、それらには宿主細菌に感染して溶菌してしまうものもあれば、プロファージとして細菌の染色体に挿入されるものもある[134]。バクテリアは、外来DNAを分解する制限修飾システム[135]や、バクテリアが過去に接触したファージのゲノムの断片を保持するためにCRISPR-Casシステムを通じて、ファージ感染に抵抗する[136][137]。
- 遺伝子導入の3番目の方法は接合とよばれるプロセスであり、DNAは細胞接触によって他の細菌細胞から直接導入される。通常の状況では、形質導入、接合、および形質転換には、同じ種間でのDNA移動が含まれるほか、異なる細菌種の個体間での移動も発生する場合があり、これは抗生物質耐性の移動などの重大な結果をもたらす可能性がある[138][139]。このような場合、他の細菌や環境からの遺伝子獲得は遺伝子水平伝播と呼ばれ、自然条件下で広範に発生していると考えられている[140]。
運動性
[編集]
多くの細菌には運動性があり、様々なメカニズムを使用して自分自身を動かすことができる。最もよく研究されている運動機構は鞭毛である。これは、分子モーターによって回転する長いフィラメント状の組織であり、プロペラのような動きを生み出すことで推進力を得るものである[141]。細菌のべん毛は約20のタンパク質でできており、その調節と組み立てにはさらに約30のタンパク質が必要である[141]。べん毛は、ベースとなる可逆モーターによって駆動される回転構造をとり、細胞膜を貫通する電気化学的勾配を利用してエネルギーを供給している[142]。

細菌は様々な方法で鞭毛を使用することで、多様な種類の動きを生み出すことができる。大腸菌などの多くの細菌は、前進(遊泳)とタンブリング(回転)という2つの異なる移動モードがあります。タンブリングにより細菌は移動方向を変えることができ、3次元空間をランダムウォークすることができる[143]。細菌の種によって、表面のべん毛の数と配置は異なり、単一の鞭毛を持つ単毛性(monotrichous)、細胞の各端部に一本ずつ鞭毛を持つ両毛性(amphitrichous)、細胞の片極に多数の鞭毛を持つ叢毛性(lophotrichous)、細胞の表面全体に鞭毛が分布している周毛性、に分類される。他にも、スピロヘータの鞭毛は、ペリプラズム空間の2つの膜の間に見られ、細胞がねじれながら移動するような独特の螺旋状の細胞形状をとっている[144]。
他のタイプの細菌の動きとしては、IV型線毛と呼ばれる構造に依存する痙攣運動[145]と、また別のメカニズムを利用した滑走運動と呼ばれる運動が知られている。痙攣運動では、棒状の線毛が細胞から伸び、基質との結合と収縮を繰り返すことで、細胞を前方に引っ張ることで移動する[146]。
運動性細菌は、走光性、磁気走性、走化性など、特定の刺激に対して引き寄せられたり逃げ出したりする「走性」と呼ばれる行動パターンを示す[147][148][149]。また走性以外としては、粘液細菌で見られるように、個々の細菌が一緒に移動して細胞の波を形成し、次に分化して胞子を含む子実体を形成するような例も知られている[150]。粘液細菌は、液体・固体の両方の培地で運動性を示す大腸菌のような細菌とは異なり、固体表面上でのみ運動性を示す[151]。
リステリア菌と赤痢菌のいくつかの種は、細胞骨格を利用して宿主細胞内を移動する。細胞骨格は通常、細胞内の細胞小器官を移動させるために使用される機関である。細菌細胞の一方の極でアクチン重合を促進させることで、宿主細胞の細胞質内を移動するような「尾」を形成することができる[152]。
細胞間コミュニケーション
[編集]細菌の一部には、生物発光の化学システムを持つものが知られている。例えば魚と共生している発光細菌では、魚はその光を利用して、他の魚や動物を引き付け捕食することに役立てている[153]。
細菌はしばしば、多細胞凝集体(バイオフィルム)という構造をとり、様々な分子シグナルを交換し合う細胞間コミュニケーションを行い、協調した多細胞行動をとっている[154] [155]。多細胞間で協力し合うことは、細胞間での分業を可能にしたり、単一細胞では効果的に使用できないリソースを分配することに役立つほか、拮抗薬に対する集合的な防御や、異なる細胞型への分化による集団生存の最適化にも寄与している[156]。たとえば、バイオフィルム内の細菌は、同じ種の個々の浮遊性(自由生活性)細菌よりも抗菌剤に対する耐性が500倍以上高くなる例が報告されている[157]。
分子信号による細胞間コミュニケーションの1つのタイプは、クオラムセンシングと呼ばれている。これは、ある特定の生物プロセスを実施するのに十分な細胞密度がその環境に存在しているのか、を判断する際に利用される。具体的には、細菌が細胞分裂を繰り返し、ある程度の密度に達した際に初めて消化酵素を分泌したり発光を始めたりする例が知られており、この生物プロセスの開始タイミングの調整にクオラムセンシングが利用されている[158][159]。クオラムセンシングにより、細菌は遺伝子発現を調整し、細胞集団の成長とともに蓄積する自己誘導物質やフェロモンを生成、放出、および検出することができる[160]。
系統分類と同定
[編集]
※細菌の右半分(紫色)を占めるCPR群は、2010年代に報告された未培養系統群。

類似性に基づいて生物に名前を付けてグループ化し、細菌種の多様性を説明しようとすることは、分類と呼ばれる。細菌は、細胞構造(直接観察による構造的・解剖学的性質)、細胞代謝(最終電子受容体など、代謝系に関わる生理・生化学的性質)、あるいはDNAや脂肪酸、色素、抗原、キノン、などの細胞成分の違いに基づいて分類することができる[162]。このスキームは細菌株の識別と分類を可能にしたが、実際のところこのような観察可能な違いは、種間の違いを表しているのか、あるいは同じ種内での株間の違いを表しているに過ぎないのか、などを判断することは困難である。このような不確実性が生まれる原因としては、ほとんどの細菌は特徴的な構造を持っていないことや、無関係の種間でも遺伝子水平伝播が発生していまうことが挙げられる[163]。また逆に、遺伝子の水平伝播により、密接に関連する細菌であっても、形態や代謝が大きく異なるものも知られている。
このような不確実性を克服するために、現代の細菌分類では、DNA中のグアニン・シトシンの比率(GC含量)やゲノム-ゲノムハイブリダイゼーション(DNA-DNA分子交雑法)などの遺伝学的手法、およびrRNA遺伝子のように水平伝播が発生しにくく生物に保存されやすい遺伝子の配列情報を利用して、分子系統を解析することが広く行われている[164]。細菌の分類は、International Journal of Systematic Bacteriology [165]およびBergey's Manual of Systematic Bacteriologyに掲載されることで定義される[166]。国際原核生物分類命名委員会(International Committee on Systematics of Prokaryotes; ICSP)は、細菌や分類学的カテゴリの命名とその階層化のための国際ルールを、国際細菌命名規約として策定している[167]。分類には属以上の単位として科、目、綱、門、界、ドメインなどが与えられている。
歴史的には、バクテリアはかつて植物界であるPlantaeの一部と見なされ、「Schizomycetes」(分裂菌)と呼ばれていた[168]。そのため、宿主内の集団細菌やその他の微生物は、しばしば"flora"(「植物相」)と呼ばれる[169]。また、「細菌」という用語は伝統的に、全ての微視的な単一細胞の原核生物に適用されていた。しかしながら分子分類学の発展により、原核生物には2つの別々のドメインから構成されていることが分かっている。この2つのドメインは、元々は真正細菌(Eubacteria )と古細菌(Archaebacteria)と呼ばれていたが、現在は細菌(Bacteria)と古細菌(Archaea)と呼ばれて、両者は共通祖先から分岐し独立に進化してきたものだと考えられている[170]。そして真核生物は、細菌よりも古細菌により近縁なドメインであると考えられている。細菌と古細菌という2つのドメインは、真核生物と併せて、3ドメイン説の基礎となっており、今日の微生物学分野においても最も一般的に受け入れられている分類システムである[171]。とはいえ、分子系統学は比較的近年に導入された手法であり、利用可能なゲノム配列の数は今日でも急速に増加しているため、細菌分類は頻繁に変更され拡大している分野である[172][173]。
医学分野においては、感染を引き起こす細菌種によって異なる治療法が選択されることがあるため、実験室での細菌の同定が重要になる。そのため、「人間の病原体を特定する」ということは、細菌を特定する技術を開発する上で主要な推進力となってきた[174]。

1884年にハンス・クリスチャン・グラムによって開発されたグラム染色法は、細胞壁の構造的特徴に基づいて細菌を特徴づけることができる[176][177]。グラム陽性細菌では細胞壁のペプチドグリカンの厚い層は紫色に染まり、逆にグラム陰性細菌の薄い細胞壁はピンク色に見える[176]。細胞形態とグラム染色を組み合わせることにより、ほとんどの細菌は、4つのグループ(グラム陽性球菌、グラム陽性桿菌、グラム陰性球菌、およびグラム陰性桿菌)のいずれかに属するものとして分類することができる。いくつかの系統ではグラム染色以外の染色方法がより同定に適していることが知られている。例えばマイコバクテリアやノカルジアは抗酸菌であり、抗酸染色によって識別することができる[178]。細菌系統によっては単純な染色では同定できず、特別な培地での培養や血清学などの他の技術も利用する必要がある場合もある[179]。
培養は、サンプル内で他の細菌の増殖を制限しながら、特定の細菌のみ増殖を促進し識別できるようにするための技法である[180]。これらの技術はしばしば、特定の検体サンプルを対象として設計される場合がある。たとえば、肺炎の原因菌を特定するために喀痰サンプルを利用する手法や、下痢の原因菌を特定するために便検体を選択培地で培養する手法などがある。一方で、血液、尿、髄液など、通常は無菌である検体は、考えられる全ての生物を増殖させるように設計された条件下で培養される[181][182]。病原性生物が分離されると、その形態、成長パターン(好気性または嫌気性成長など)、溶血のパターン、および染色によって、さらに詳細な特徴づけが可能となる[183]。
細菌の分類と同様に、細菌の同定にも分子生物学的な手法や[184]質量分析法を利用した手法が使われる[185]。地球上の大半の細菌は未だ特徴付けられておらず、また実験室で培養することが困難であると考えられている[186]。ポリメラーゼ連鎖反応(PCR)などのDNAベースの診断手法は、培養ベースの方法と比較して特異性や診断時間の短縮化の点で優位である[187]。これらの方法はまた、代謝的に活性であるが分裂しない生存可能であるが培養不可能な細胞(VNC)の検出と同定も可能にする[188]。しかし、これらの改良された方法を使用しても、膨大に存在すると考えられる細菌種の総数を見積もることは困難である。2011年の段階で、細菌や古細菌には9,300弱の原核生物種が分類として報告され登録されている[189]。しかし、細菌多様性の真数を推定した研究では合計で107~1010種いると予想されており、さらにはこの数字でさえ何桁も過小評価している可能さえある[190][191]。
近年では、16SrRNAだけでなくゲノム規模の比較に基づいてより正確な分類を目指す動きが活発となっている(例えばGTDB)。その結果、古典的によく知られた細菌の分類が解体されることも珍しくなく、現在も絶えず新しいグループの追加と既存のグループの書き換えが進んでいる(例えばデルタプロテオバクテリア)。GTDBによれば、2021年7月時点で、127の門(Phylum)が記載されている。
細菌ドメインの主な分類
[編集]細菌は系統学的に分類されているが、メタゲノム情報の蓄積などにより新しく発見される種も増え続けており、分類体系は確立していない。大まかな枠組みは以下の通りである[192]。
これらの枠組みに含まれない種も多数存在する(英語版記事「Bacterial phyla」などを参照)。
他の生物との相互作用
[編集]
細菌は他の生物と複雑に相互作用することが知られている。このような共生的な相互作用は、寄生、相利共生、片利共生の3種類に分類することができる[194]。
共生
[編集]片利共生(commensalism)という言葉は、「同じ食卓で食べる」という意味のcommensalに由来しており[195] 、あらゆる動植物には片利共生細菌が存在している。例えば人間や他の動物においては、何百万もの細菌が、皮膚や気道、腸、その他の開口部に生息している[196][197] 。常在菌(normal flora) [198]や片利共生体(commensals)[199]と呼ばれるこれらの細菌は、通常は害を及ぼすことはないが、場合によっては体内に侵入して感染症を引き起こす可能性がある。例えば大腸菌は人間の腸内でよく見られる共生生物の一種であるが、尿路感染症を引き起こすものが知られている[200]。同様に、正常なヒトの口腔内で一般的に見られる連鎖球菌は、心臓病(亜急性細菌性心内膜炎)を引き起こす可能性がある[201]。
捕食
[編集]一部の細菌種は、他の微生物を殺して消化吸収するため、捕食性細菌と呼ばれる[202]。例えば、Myxococcus xanthusは集合体を形成し、接触した他の細菌を捕食する[203]。他には、獲物の細胞に付着して消化し栄養素を吸収するものや、細胞に侵入して細胞質内で増殖するような捕食性細菌が知られている[204]。これらの略奪的な細菌は、死んだ微生物を消費したサプロファージから、他の生物を捕らえて殺すことができるように適応することによって進化したと考えられている[205]。
相利共生者
[編集]特定の細菌は、生存に不可欠な密接した空間的相互作用を形成し、これは相利共生と呼ばれる。例えば種間水素移動(interspecies hydrogen transfer)と呼ばれる相互作用では、酪酸やプロピオン酸などの有機酸を消費して水素を生成する嫌気性細菌クラスターと、水素を消費するメタン生成古細菌との間で発生する[206]。この関係性において、水素生成細菌は自身が生成した水素が細胞外に蓄積してしまうため、有機物を周辺環境から吸収し消費することができなくなってしまう。そのため、水素を消費する古細菌と相互作用することによって、成長できる程度に細胞周辺の水素濃度を低く保っている[207]。
土壌では、根圏(根の表面や近接する土壌)に存在する微生物が窒素固定を行い、窒素ガスを窒素化合物に変換する[208]。これは、窒素自体を固定できない多くの植物に、吸収しやすい形の窒素を提供するのに役立っている。他の多くの細菌は、人間や他の生物の共生細菌として発見されている。例えば、正常なヒトにおいて1,000以上の細菌種が腸内細菌として存在しており、それらはの腸は、腸免疫や、葉酸、ビタミンK、ビオチンなどを含むビタミンの合成、乳糖の乳酸への変換(ラクトバチルスを参照)、複雑な難消化性炭水化物の発酵、など様々なプロセスに寄与している[209][210][211]。この腸内細菌叢の存在はまた、潜在的に競合相手の排除などによって病原性細菌の増殖を阻害しており、このような有益な細菌は実際にプロバイオティクス栄養補助食品として市販されている[212]。
一部の共生細菌および古細菌はビタミンB 12(コバラミン)合成に必要な酵素遺伝子を有しており、ほぼ全ての動物は食物連鎖を通してこのような細菌が生産するビタミンの恩恵を受けている。ビタミンB 12は水溶性ビタミンであり、DNA合成や脂肪酸代謝、アミノ酸代謝における補因子として、ヒトのあらゆる細胞の代謝に関与している。ミエリンの合成における役割のため、神経系の正常な機能においても重要である[213]。
病原性細菌
[編集]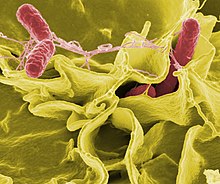
ヒトや動物の体は、皮膚や粘膜で成長する有益な共生細菌や、主に土壌や腐生植物で成長する腐生細菌など、多くの種類の細菌に常にさらされている。血液や組織液には、多くの細菌の成長を維持するのに十分な栄養素が含まれている。体には、微生物が組織内に侵入することに対抗し、多くの微生物に対する自然免疫を獲得するといった、防御機構が存在する[214]。ウイルスとは異なり細菌は比較的ゆっくりと進化するため、ある動物に感染する細菌が異なる種の動物にも感染することも良く見られる[215]。
細菌が他の生物と寄生的な関係を形成する場合、それらは病原体として分類される[216]。様々な病原性細菌がヒトの病気や死を引き起こすことが知られている。例えば、破傷風(破傷風菌)、腸チフス、ジフテリア、梅毒、コレラ、食中毒、ハンセン病(Micobacterium(らい菌))、結核(結核菌)、などが代表的である[217]。ヘリコバクター・ピロリによる消化性潰瘍の場合のように、既知の医学的疾患の病原性の原因が何年も後に発見される可能性もある[218]。細菌性疾患はまた農業分野においても重要であり、例えばすすかび病や火傷病、ヨーネ病、乳腺炎などを引き起こす細菌の存在や、家畜に感染するサルモネラや炭疽菌などの存在が知られている[219]。

それぞれの病原体は、その宿主であるヒトとの間で、特徴的な相互作用を引き起こす。ブドウ球菌や連鎖球菌などの一部の生物は、皮膚感染症、肺炎、髄膜炎、敗血症、全身性炎症反応によるショックを引き起こし、大量の血管拡張、そして死を引き起こす可能性がある[220]。しかし、これらの細菌は通常のヒト常在菌の一部でもあり、普段は病気をまったく引き起こすことなく皮膚や鼻に存在している。また他の例としては、他の生物の細胞内でのみ成長および繁殖することができる細胞内寄生細菌であるリケッチアが挙げられる。リケッチアの1つの種は発疹チフスを引き起こし、別の種はロッキー山紅斑熱を引き起こす。別の門の細胞内寄生細菌であるクラミジアは、肺炎や尿路感染症を引き起こす可能性があり、冠状動脈性心臓病に関与している可能性のある種が含まれている[221]。緑膿菌(Pseudomonas aeruginosa),やBurkholderia cenocepacia、Mycobacterium avium,などの一部の種は日和見病原体であり、主に免疫抑制や嚢胞性線維症に苦しむ人々に対して病気を引き起こす[222][223]。一部の細菌は毒素を産生し、それが病気を引き起こす[224]。これらには、壊れた細菌細胞に由来するエンドトキシンと、細胞外に分泌されるエキソトキシンが含まれる[225]。たとえばボツリヌス菌は呼吸麻痺を引き起こす強力なエキソトキシンを生成し、サルモネラ菌は胃腸炎を引き起こすエンドトキシンを生成する[225]。一部のエンドトキシンはトキソイドに変換することができ、トキソイドは病気を予防するためのワクチンとして使用される場合がある[226]。
多くの細菌感染症は、抗生物質で治療することができる。抗生物質は、細菌を殺す場合は殺菌性、細菌の増殖を防ぐだけの場合は静菌性に分類される。抗生物質には多くの種類があり、それぞれは宿主には悪影響を与えず、病原体が行う生物プロセスのみを阻害する。例えば、クロラムフェニコールとピューロマイシンは細菌のリボソームを阻害するが、構造的に異なる真核生物のリボソームは阻害しないため、細菌に対して選択的に毒性が発揮される[227]。抗生物質は、人間の病気治療や集約農業、動物の成長促進などの目的で使用される。抗生物質は、その乱用と繁用により細菌集団における抗生物質耐性の急速な発達に寄与していると指摘されている[228]。すなわち抗生物質が「効かない」例が激増している。
感染は、注射器の針で皮膚を刺す前に皮膚を殺菌するなどといったような消毒によっても防ぐことができる。細菌による汚染を防ぐために、外科用や歯科用の器具は常に使用前に滅菌されている。漂白剤などの消毒剤も、物体表面の細菌やその他の病原体を殺して汚染を防ぎ、感染のリスクをさらに減らすために使用される[229]。
産業技術への応用
[編集]ラクトバチルスやラクトコッカスといった乳酸菌は、酵母やカビなどと組み合わせられて、チーズや漬物、醤油、ザワークラウト、酢、ワイン、ヨーグルトといった様々な発酵食品の生産において何千年もの間使用されてきた[230][231]。
また、細菌が様々な有機化合物を分解する能力は顕著であり、廃棄物処理やバイオレメディエーションにも使用されている。石油に含まれる炭化水素を消化することができる細菌は、石油流出の浄化によく利用されている[232]。プリンス・ウィリアム湾では、1989年のエクソンバルディーズ号原油流出事故後、自然発生する石油分解細菌の成長を促進するために肥料が追加された。これは、油であまり厚く覆われていないビーチにおいては効果的であった。細菌はさらに、産業毒性廃棄物のバイオレメディエーションにも使用される[233]。化学産業において、医薬品や農薬として利用できるような鏡像異性的を持つ純粋化学物質の生産においても、細菌は重要な存在である[234]。
細菌は、生物的害虫駆除において農薬の代わりに使用することもできる。これには通常、グラム陽性の土壌に生息する細菌であるバチルス・チューリンゲンシス(Bacillus thuringiensis、BTとも呼ばれる)が含まれる。この細菌の亜種は、DipelやThuricideなどの商品名で、鱗翅目特有の殺虫剤として使用されている[235]。それらの特異性のために、人間、野生生物、花粉交配者、および他のほとんどの益虫にほとんど影響を与えないため、これらの農薬は環境にやさしいと見なされている[236][237]。
細菌は急速に成長する能力を持ち、操作が比較的容易であるため、分子生物学、遺伝学、生化学など分野で頻繁に利用される。例えば細菌のDNAを変異させ、その表現型を調べることで、細菌の遺伝子、酵素、代謝経路の機能を調べることができ、得られた知識をより複雑な生物に適用することができる[238]。細胞の生化学を理解することで、様々な生物において、大量の酵素動態や遺伝子発現データに基づく数学的モデルを作ることができる。いくつかのよく研究された細菌では研究が進められており、例えば大腸菌の代謝モデルが作成され検証が進められている[239][240]。細菌の代謝や遺伝学の理解は、インスリンや成長因子、抗体などの治療用タンパク質の生産のために細菌を利用するという、バイオエンジニアリングのためのバイオテクノロジーに繋がる[241][242]。
細菌株のサンプルは研究において重要であるため、生物学的リソースセンター(Biological Resource Centers)で分離および保存される。これにより、世界中の科学者が菌株を確実に入手できる体制が整っている[243]。
歴史
[編集]
発酵は古代利用されていたが、細菌の発見は17世紀に遡る。1676年にアントニ・ファン・レーウェンフックによって発見され原生動物と合わせて“animalcules”(微小動物)と呼ばれた。この時は、彼自身が設計した単一レンズの顕微鏡を使用して最初に観察された。その後、彼はロンドン王立学会に一連の手紙で彼の観察を発表した[244]。バクテリアは、レーウェンフックの最も注目すべき顕微鏡的発見であった。ただし、細菌は彼の作成した単純なレンズが見ることができる限界のサイズであったため、他の誰も1世紀以上それらを再び見ることができず、科学の歴史の中で最も印象的な休止の1つであったと言える[245]。彼の観察には、彼がアニマルキュールと呼んだ原生動物も含まれており、後世に提案された細胞説に併せて彼の発見は再度注目されることになった[246]。
1828年、クリスチャン・ゴットフリート・エーレンベルクは、顕微鏡で観察した微生物が細い棒状であったため、ギリシア語で「小さな杖」を意味するβακτήριονから“Bacterium”と呼んだ。しかしながら実際に彼が観察した「Bacterium」は、非芽胞形成の桿菌を含んだ属であり[247]、これは1835年にエーレンバーグ自身によって芽胞形成桿菌属であるバチルス(Bacillus)として定義された[248]。
1859年にはルイ・パスツールが、アルコール発酵が微生物によって引き起こされることを示し、さらに発酵が自然発生的な現象ではないことを示すことで、自然発生説を否定した。このとき、パスツールは発酵を起こす微生物を細菌だと考えたが、実際には菌類である(発酵に関連するものは細菌ではなく、一般的には酵母やカビといった真菌である)。ロベルト・コッホとともに、パスツールは病原菌理論の初期の提唱者であった[249]。ただし、彼らの以前にも、センメルヴェイス・イグナーツとジョゼフ・リスターによって、医療業務における手指の消毒の重要性は認識されていた。センメルヴェイズのアイデアは却下され、このトピックに関する彼の本は医学界によって当時は非難されたが、その後にリスターが1870年代から手の消毒を始めた。 1840年代に病院での手洗いに関する規則から始めたセンメルワイスは、細菌自体に関する考えの先駆者であった。後にリスターは、病気は「動物の有機物の分解」に起因すると考えて消毒の重要性を説いた[250]。
細菌培養法の基礎は、医療微生物学のパイオニアであるロベルト・コッホによって確立され、一連の研究により炭疽菌、結核菌、コレラ菌が病原性の細菌によって引き起こされることが証明された。特に結核に関する彼の研究により、コッホはついに病原菌理論を証明し、1905年にノーベル賞を受賞した[251]。また、コッホの原則という、生物が病気の原因であるかどうかをテストするための基準が提唱され、これは今日でも使用されているものである[252]。
フェルディナント・コーンは細菌学の創始者であり、1870年から細菌を研究していた。コーンは、細菌をその形態に基づいて分類した最初の人物である[253][254]。
19世紀には多くの研究により、細菌が様々な病気の原因であることが判明したが、効果的な抗菌治療法は不明であり、対症療法しか存在しなかった[255]。20世紀に入ると培養法が確立されたことも相まって、細菌の研究が進んでいく。1910年、 パウル・エールリヒは、梅毒トレポネーマ(梅毒の原因となるスピロヘータを選択的に染色する染料を、病原体を選択的に殺す化合物に変えることにより、最初の抗生物質を開発した[256]。エーリッヒは、免疫学の研究で1908年のノーベル賞を受賞し、細菌を検出および識別するための染色の使用を開拓し、グラム染色と抗酸染色の基礎となった[257]。1910年、パウル・エールリヒと秦佐八郎によって初の抗菌剤サルバルサンが開発され、1929年にはアレクサンダー・フレミングによって抗生物質ペニシリンが発見された。
細菌についての知識が深まるにつれ、分類学上での細菌の位置づけはしばしば変更されている。発見時は2界説に従い植物界に振り分けられ、1866年にはエルンスト・ヘッケルによって単細胞生物をまとめた原生生物界に組み入れられた(3界説)。1930年頃になると原核生物と真核生物の違いが認識され、2帝説原核生物帝(1937年)、次いで4界説(のち5界説)モネラ界(1956年)が提唱された。現在に至る一般の細菌のイメージは5界説における原核生物に対応している(藍色細菌は、旧名藍藻の概念としては除くこともある[要出典])。
しかし、1977年、カール・ウーズらによって原生生物界の単系統性に疑問が投げかけられ、メタン生成菌(のち高度好塩菌と一部の好熱菌も)を除く原核生物として、Kingdom Eubacteria(真正細菌界)が定義された[258]。1990年には16S rRNA配列に基づいて、当時の古細菌(メタン生成菌、高度好塩菌、一部の好熱菌)を除く原核生物としてDomain Bacteria(細菌ドメイン)が定義され、同時に古細菌はDomain Archaea (古細菌ドメイン)として新たに定義される、3ドメイン説が提唱された[170]。
カール・ウーズにより提唱された3ドメイン説(細菌、古細菌、真核生物)は現在も広い支持を得ているが、各ドメインの進化上の関係性は現在も議論が続いている。近年になって分子系統解析の進歩、および真核生物に非常に近縁の古細菌(アスガルド古細菌)が発見されるに至って[259]、真核生物は古細菌の一部から進化したとする説が優勢になりつつある(2ドメイン説とも呼ばれる)。2ドメイン説では、細菌は原始の地球に出現した生命体の2つのグループの内の一つということになる(もう一つは古細菌)。さらに近年では、それまで知られていた細菌のグループとは全く別系統に属する新種の細菌グループ(Candidate Phyla Radiation;CPR)が見つかり、その規模は既知の細菌全体に匹敵するとも推測されている[192]。そのため細菌ドメインの範囲は現在もさらに拡大している。
脚注
[編集]出典
[編集]- ^ Jean P. Euzéby, Aidan C. Parte. “Domain Bacteria”. List of Prokaryotic names with Standing in Nomenclature. 2024年7月17日閲覧。
- ^ bacteria (n.)Online Etymology Dictionary
- ^ Krasner 2014, p. 38.
- ^ “bacteria | Origin and meaning of bacteria by Online Etymology Dictionary”. Online Etymology Dictionary. 2020年4月18日閲覧。
- ^ βακτηρία in Liddell and Scott.
- ^ Harper, Douglas. "bacteria". Online Etymology Dictionary.
- ^ C R Woese, O Kandler, M L Wheelis (June 1990). “Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya”. Proceedings of the National Academy of Sciences of the United States of America 87 (12): 4576–79. Bibcode: 1990PNAS...87.4576W. doi:10.1073/pnas.87.12.4576. PMC 54159. PMID 2112744.
- ^ Filipa Godoy-Vitorino (July 2019). “Human microbial ecology and the rising new medicine”. Annals of Translational Medicine 7 (14): 342. doi:10.21037/atm.2019.06.56. PMC 6694241. PMID 31475212.
- ^ J W Schopf (July 1994). “Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic”. Proceedings of the National Academy of Sciences of the United States of America 91 (15): 6735–42. Bibcode: 1994PNAS...91.6735S. doi:10.1073/pnas.91.15.6735. PMC 44277. PMID 8041691.
- ^ Edward F. DeLong, Norman R. Pace (August 2001). “Environmental diversity of bacteria and archaea”. Systematic Biology 50 (4): 470-478. doi:10.1080/10635150118513.
- ^ J R Brown, W F Doolittle (December 1997). “Archaea and the prokaryote-to-eukaryote transition”. Microbiology and Molecular Biology Reviews 61 (4): 456-502. doi:10.1128/mmbr.61.4.456-502.1997.
- ^ Bertram Daum, Vicki Gold (June 2018). “Twitch or swim: towards the understanding of prokaryotic motion based on the type IV pilus blueprint”. Biological Chemistry 399 (7): 799-808. doi:10.1515/hsz-2018-0157. PMID 29894297.
- ^ “The universal ancestor and the ancestor of bacteria were hyperthermophiles”. Journal of Molecular Evolution 57 (6): 721–30. (December 2003). Bibcode: 2003JMolE..57..721D. doi:10.1007/s00239-003-2522-6. PMID 14745541.
- ^ Battistuzzi, Fabia U.; Feijao, Andreia; Hedges, S. Blair (November 2004). “A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land”. BMC Evolutionary Biology 4: 44. doi:10.1186/1471-2148-4-44. PMC 533871. PMID 15535883.
- ^ Homann, Martin (23 July 2018). “Microbial life and biogeochemical cycling on land 3,220 million years ago”. Nature Geoscience 11 (9): 665–671. Bibcode: 2018NatGe..11..665H. doi:10.1038/s41561-018-0190-9et al.
- ^ Van Kranendonk, Martin J.; Baumgartner, Raphael; Djokic, Tara; Ota, Tsutomu; Steller, Luke; Garbe, Ulf; Nakamura, Eizo (2021-01-01). “Elements for the Origin of Life on Land: A Deep-Time Perspective from the Pilbara Craton of Western Australia” (英語). Astrobiology 21 (1): 39–59. doi:10.1089/ast.2019.2107. ISSN 1531-1074.
- ^ López-García, Purificación; Moreira, David (2020-05). “The Syntrophy hypothesis for the origin of eukaryotes revisited” (英語). Nature Microbiology 5 (5): 655–667. doi:10.1038/s41564-020-0710-4. ISSN 2058-5276.
- ^ “Why is primary endosymbiosis so rare?”. The New Phytologist 231 (5): 1693–1699. (May 2021). doi:10.1111/nph.17478. PMID 34018613.
- ^ Stephens, Timothy G. and Gabr, Arwa and Calatrava, Victoria and Grossman, Arthur R. and Bhattacharya, Debashish (October 2020). “Extremophilic Microorganisms for the Treatment of Toxic Pollutants in the Environment”. Molecules (Basel, Switzerland) 25 (21): 4916. doi:10.3390/molecules25214916. PMC 7660605. PMID 33114255.
- ^ “Life in acid: pH homeostasis in acidophiles”. Trends in Microbiology 15 (4): 165–71. (April 2007). doi:10.1016/j.tim.2007.02.005. PMID 17331729.
- ^ Flemming, Hans-Curt; Wuertz, Stefan (April 2019). “Bacteria and archaea on Earth and their abundance in biofilms”. Nature Reviews. Microbiology 17 (4): 247–260. doi:10.1038/s41579-019-0158-9. PMID 30760902.
- ^ Wheelis 2008, p. 362.
- ^ Kushkevych, Ivan and Procházka, Jiří and Gajdács, Márió and Rittmann, Simon K.-M. R. and Vítězová, Monika (June 2021). “Molecular Physiology of Anaerobic Phototrophic Purple and Green Sulfur Bacteria”. International Journal of Molecular Sciences 22 (12): 6398. doi:10.3390/ijms22126398. PMC 8232776. PMID 34203823.
- ^ a b c Pommerville 2014, p. 3–6.
- ^ a b c 平松啓一・中込治 編集「第III章 細菌学総論」『標準微生物学』(10版)、2009年。ISBN 978-4-260-00638-5。
- ^ Yinon M. Bar-On, Rob Phillips, Ron Milo (June 2018). “The biomass distribution on Earth”. Proceedings of the National Academy of Sciences of the United States of America 115 (25): 6506–11. doi:10.1073/pnas.1711842115. PMC 6016768. PMID 29784790.
- ^ Krasner 2014, p. 74.
- ^ Heide N. Schulz; Bo Barker Jørgensen (2001). “Big bacteria”. Annual Review of Microbiology 55: 105–37. doi:10.1146/annurev.micro.55.1.105. PMID 11544351.
- ^ Williams, Caroline (2011). “Who are you calling simple?”. New Scientist 211 (2821): 38–41. doi:10.1016/S0262-4079(11)61709-0.
- ^ Jean-Marie Volland and Silvina Gonzalez-Rizzo and Olivier Gros and Tomáš Tyml and Natalia Ivanova and Frederik Schulz and Danielle Goudeau and Nathalie H. Elisabeth and Nandita Nath and Daniel Udwary and Rex R. Malmstrom and Chantal Guidi-Rontani and Susanne Bolte-Kluge and Karen M. Davies and Maïtena R. Jean and Jean-Louis Mansot and Nigel J. Mouncey and Esther R. Angert and Tanja Woyke and Shailesh V. Date (2022). “A centimeter-long bacterium with DNA contained in metabolically active, membrane-bound organelles”. Science 376 (6600): 1453-1458. doi:10.1126/science.abb3634.
- ^ J Robertson, M Gomersall, P Gill (November 1975). “Mycoplasma hominis: growth, reproduction, and isolation of small viable cells”. Journal of Bacteriology 124 (2): 1007–18. doi:10.1128/JB.124.2.1007-1018.1975. PMC 235991. PMID 1102522.
- ^ Branko Velimirov (2001). “Nanobacteria, Ultramicrobacteria and Starvation Forms: A Search for the Smallest Metabolizing Bacterium”. Microbes and Environments 16 (2): 67–77. doi:10.1264/jsme2.2001.67.
- ^ Dusenbery, David B (2009).
- ^ Desirée C. Yang, Kris M. Blair, Nina R. Salama (March 2016). “Staying in Shape: the Impact of Cell Shape on Bacterial Survival in Diverse Environments”. Microbiology and Molecular Biology Reviews 80 (1): 187–203. doi:10.1128/MMBR.00031-15. PMC 4771367. PMID 26864431.
- ^ “Bacterial cell shape”. Nature Reviews. Microbiology 3 (8): 601–10. (August 2005). doi:10.1038/nrmicro1205. PMID 16012516.
- ^ Kevin D. Young (September 2006). “The selective value of bacterial shape”. Microbiology and Molecular Biology Reviews 70 (3): 660–703. doi:10.1128/MMBR.00001-06. PMC 1594593. PMID 16959965.
- ^ Crawford 2007, p. xi.
- ^ Claessen, Dennis; Rozen, Daniel E.; Kuipers, Oscar P.; Søgaard-Andersen, Lotte; van Wezel, Gilles P. (February 2014). “Bacterial solutions to multicellularity: a tale of biofilms, filaments and fruiting bodies”. Nature Reviews. Microbiology 12 (2): 115–24. doi:10.1038/nrmicro3178. PMID 24384602.
- ^ Lawrence J. Shimkets (1999). “Intercellular signaling during fruiting-body development of Myxococcus xanthus”. Annual Review of Microbiology 53: 525–49. doi:10.1146/annurev.micro.53.1.525. PMID 10547700.
- ^ Dale Kaiser (2004). “Signaling in myxobacteria”. Annual Review of Microbiology 58: 75–98. doi:10.1146/annurev.micro.58.030603.123620. PMID 15487930.
- ^ Wheelis 2008, p. 75.
- ^ Abhishek Mandal, Ahana Dutta, Reshmi Das, Joydeep Mukherjee (June 2021). “Role of intertidal microbial communities in carbon dioxide sequestration and pollutant removal: A review”. Marine Pollution Bulletin 170: 112626. doi:10.1016/j.marpolbul.2021.112626. PMID 34153859.
- ^ Rodney M. Donlan (September 2002). “Biofilms: microbial life on surfaces”. Emerging Infectious Diseases 8 (9): 881–90. doi:10.3201/eid0809.020063. PMC 2732559. PMID 12194761.
- ^ Steven S. Branda, Åshild Vik, Lisa Friedman, Roberto Kolter (January 2005). “Biofilms: the matrix revisited”. Trends in Microbiology 13 (1): 20–26. doi:10.1016/j.tim.2004.11.006. PMID 15639628.
- ^ Mary Ellen Davey, George A. O'toole (December 2000). “Microbial biofilms: from ecology to molecular genetics”. Microbiology and Molecular Biology Reviews 64 (4): 847–67. doi:10.1128/MMBR.64.4.847-867.2000. PMC 99016. PMID 11104821.
- ^ Rodney M. Donlan and J. William Costerton (April 2002). “Biofilms: survival mechanisms of clinically relevant microorganisms”. Clinical Microbiology Reviews 15 (2): 167–93. doi:10.1128/CMR.15.2.167-193.2002. PMC 118068. PMID 11932229.
- ^ Microbiology : an Evolving Science (Third ed.). New York: W W Norton. (2013). p. 82. ISBN 978-0393123678
- ^ Feijoo-Siota, Lucía; Rama, José Luis R.; Sánchez-Pérez, Angeles; Villa, Tomás G. (July 2017). “Considerations on bacterial nucleoids”. Applied Microbiology and Biotechnology 101 (14): 5591–602. doi:10.1007/s00253-017-8381-7. PMID 28664324.
- ^ Thomas A. Bobik (May 2006). “Polyhedral organelles compartmenting bacterial metabolic processes”. Applied Microbiology and Biotechnology 70 (5): 517–25. doi:10.1007/s00253-005-0295-0. PMID 16525780.
- ^ Yeates, Todd O.; Kerfeld, Cheryl A.; Heinhorst, Sabine; Cannon, Gordon C.; Shively, Jessup M. (September 2008). “Protein-based organelles in bacteria: carboxysomes and related microcompartments”. Nature Reviews. Microbiology 6 (9): 681–91. doi:10.1038/nrmicro1913. PMID 18679172.
- ^ Kerfeld, Cheryl A and Sawaya, Michael R and Tanaka, Shiho and Nguyen, Chau V and Phillips, Martin and Beeby, Morgan and Yeates, Todd O (August 2005). “Protein structures forming the shell of primitive bacterial organelles”. Science 309 (5736): 936–38. Bibcode: 2005Sci...309..936K. doi:10.1126/science.1113397. PMID 16081736.
- ^ Gitai, Zemer (March 2005). “The new bacterial cell biology: moving parts and subcellular architecture”. Cell 120 (5): 577–86. doi:10.1016/j.cell.2005.02.026. PMID 15766522.
- ^ Yu-Ling Shih, Lawrence Rothfield (September 2006). “The bacterial cytoskeleton”. Microbiology and Molecular Biology Reviews 70 (3): 729–54. doi:10.1128/MMBR.00017-06. PMC 1594594. PMID 16959967.
- ^ Norris, Vic and Den Blaauwen, Tanneke and Cabin-Flaman, Armelle and Doi, Roy H and Harshey, Rasika and Janniere, Laurent and Jimenez-Sanchez, Alfonso and Jin, Ding Jun and Levin, Petra Anne and Mileykovskaya, Eugenia and others (March 2007). “Functional taxonomy of bacterial hyperstructures”. Microbiology and Molecular Biology Reviews 71 (1): 230–53. doi:10.1128/MMBR.00035-06. PMC 1847379. PMID 17347523.
- ^ Pommerville 2014, pp. 120–121.
- ^ Bryant, Donald A and Frigaard, Niels-Ulrik (November 2006). “Prokaryotic photosynthesis and phototrophy illuminated”. Trends in Microbiology 14 (11): 488–96. doi:10.1016/j.tim.2006.09.001. PMID 16997562.
- ^ Pšenčík, J., et al. (August 2004). “Lamellar organization of pigments in chlorosomes, the light harvesting complexes of green photosynthetic bacteria”. Biophysical Journal 87 (2): 1165–72. Bibcode: 2004BpJ....87.1165P. doi:10.1529/biophysj.104.040956. PMC 1304455. PMID 15298919.
- ^ Martin Thanbichler, Sherry C. Wang, Lucy Shapiro (October 2005). “The bacterial nucleoid: a highly organized and dynamic structure”. Journal of Cellular Biochemistry 96 (3): 506–21. doi:10.1002/jcb.20519. PMID 15988757.
- ^ Poehlsgaard, Jacob and Douthwaite, Stephen (November 2005). “The bacterial ribosome as a target for antibiotics”. Nature Reviews. Microbiology 3 (11): 870–81. doi:10.1038/nrmicro1265. PMID 16261170.
- ^ Yeo, Marcus and Chater, Keith (March 2005). “The interplay of glycogen metabolism and differentiation provides an insight into the developmental biology of Streptomyces coelicolor”. Microbiology 151 (Pt 3): 855–61. doi:10.1099/mic.0.27428-0. PMID 15758231.
- ^ Shiba, T and Tsutsumi, K and Ishige, K and Noguchi, T (March 2000). “Inorganic polyphosphate and polyphosphate kinase: their novel biological functions and applications”. Biochemistry. Biokhimiia 65 (3): 315–23. PMID 10739474.
- ^ Brune, Daniel C (June 1995). “Isolation and characterization of sulfur globule proteins from Chromatium vinosum and Thiocapsa roseopersicina”. Archives of Microbiology 163 (6): 391–99. doi:10.1007/BF00272127. PMID 7575095.
- ^ Kadouri, Daniel; Jurkevitch, Edouard; Okon, Yaacov; Castro-Sowinski, Susana (2005). “Ecological and agricultural significance of bacterial polyhydroxyalkanoates”. Critical Reviews in Microbiology 31 (2): 55–67. doi:10.1080/10408410590899228. PMID 15986831.
- ^ A E Walsby (March 1994). “Gas vesicles”. Microbiological Reviews 58 (1): 94–144. doi:10.1128/MMBR.58.1.94-144.1994. PMC 372955. PMID 8177173.
- ^ Heijenoort, Jean van (March 2001). “Formation of the glycan chains in the synthesis of bacterial peptidoglycan”. Glycobiology 11 (3): 25R–36R. doi:10.1093/glycob/11.3.25R. PMID 11320055.
- ^ a b Arthur L. Koch (October 2003). “Bacterial wall as target for attack: past, present, and future research”. Clinical Microbiology Reviews 16 (4): 673–87. doi:10.1128/CMR.16.4.673-687.2003. PMC 207114. PMID 14557293.
- ^ Gram, H Christian (1884). “Über die isolierte Färbung der Schizomyceten in Schnitt- und Trockenpräparaten”. Fortschr. Med. 2: 185–89.
- ^ Hugenholtz, Philip (2002). “Exploring prokaryotic diversity in the genomic era”. Genome Biology (Springer) 3 (2): 1-8. doi:10.1186/gb-2002-3-2-reviews0003. PMC 139013. PMID 11864374.
- ^ Fiona M Walsh and Sebastian GB Amyes (October 2004). “Microbiology and drug resistance mechanisms of fully resistant pathogens”. Current Opinion in Microbiology 7 (5): 439–44. doi:10.1016/j.mib.2004.08.007. PMID 15451497.
- ^ Alderwick, Luke J and Harrison, James and Lloyd, Georgina S and Birch, Helen L (March 2015). “The Mycobacterial Cell Wall – Peptidoglycan and Arabinogalactan”. Cold Spring Harbor Perspectives in Medicine 5 (8): a021113. doi:10.1101/cshperspect.a021113. PMC 4526729. PMID 25818664.
- ^ Alderwick, Luke J and Harrison, James and Lloyd, Georgina S and Birch, Helen L (March 2014). “Biogenesis and functions of bacterial S-layers”. Nature Reviews. Microbiology 12 (3): 211–22. doi:10.1038/nrmicro3213. PMID 24509785.
- ^ Thompson, Stuart A (December 2002). “Campylobacter surface-layers (S-layers) and immune evasion”. Annals of Periodontology 7 (1): 43-53. doi:10.1902/annals.2002.7.1.43. PMC 2763180. PMID 16013216.
- ^ Beveridge TJ, Pouwels PH, Sára M, et al. (June 1997). “Functions of S-layers”. FEMS Microbiology Reviews 20 (1–2): 99-149. doi:10.1111/j.1574-6976.1997.tb00305.x. PMID 9276929.
- ^ Seiji Kojima, David F Blair (2004). The bacterial flagellar motor: structure and function of a complex molecular machine. International Review of Cytology. 233. pp. 93–134. doi:10.1016/S0074-7696(04)33003-2. ISBN 978-0-12-364637-8. PMID 15037363
- ^ Wheelis 2008, p. 76.
- ^ Cheng, Rachel A and Wiedmann, Martin (2020). “Recent Advances in Our Understanding of the Diversity and Roles of Chaperone-Usher Fimbriae in Facilitating Salmonella Host and Tissue Tropism”. Frontiers in Cellular and Infection Microbiology 10: 628043. doi:10.3389/fcimb.2020.628043. PMC 7886704. PMID 33614531.
- ^ “Towards a structural biology of bacterial conjugation”. Molecular Microbiology 23 (3): 423–29. (February 1997). doi:10.1046/j.1365-2958.1997.2411604.x. PMID 9044277.
- ^ “Secretion systems in Gram-negative bacteria: structural and mechanistic insights”. Nature Reviews. Microbiology 13 (6): 343–59. (June 2015). doi:10.1038/nrmicro3456. PMID 25978706.
- ^ “Dismantling the bacterial glycocalyx: Chemical tools to probe, perturb, and image bacterial glycans”. Bioorganic & Medicinal Chemistry 42: 116268. (July 2021). doi:10.1016/j.bmc.2021.116268. ISSN 0968-0896. PMC 8276522. PMID 34130219.
- ^ “The glycan-rich outer layer of the cell wall of Mycobacterium tuberculosis acts as an antiphagocytic capsule limiting the association of the bacterium with macrophages”. Infection and Immunity 72 (10): 5676–86. (October 2004). doi:10.1128/IAI.72.10.5676-5686.2004. PMC 517526. PMID 15385466.
- ^ “The Mycobacterium tuberculosis capsule: a cell structure with key implications in pathogenesis”. The Biochemical Journal 476 (14): 1995–2016. (July 2019). doi:10.1042/BCJ20190324. PMC 6698057. PMID 31320388.
- ^ Kalscheuer, Rainer, et al. (July 2019). “The Mycobacterium tuberculosis capsule: a cell structure with key implications in pathogenesis”. The Biochemical Journal 476 (14): 1995–2016. doi:10.1042/BCJ20190324. PMC 6698057. PMID 31320388.
- ^ Koga, Yosuke (2011-03-01). “Early Evolution of Membrane Lipids: How did the Lipid Divide Occur?” (英語). Journal of Molecular Evolution 72 (3): 274–282. doi:10.1007/s00239-011-9428-5. ISSN 1432-1432.
- ^ Fuerst, John A.; Sagulenko, Evgeny (2011-06). “Beyond the bacterium: planctomycetes challenge our concepts of microbial structure and function” (英語). Nature Reviews Microbiology 9 (6): 403–413. doi:10.1038/nrmicro2578. ISSN 1740-1526.
- ^ Azam, Talukder Ali; Ishihama, Akira (1999-11-12). “Twelve Species of the Nucleoid-associated Protein from Escherichia coli: SEQUENCE RECOGNITION SPECIFICITY AND DNA BINDING AFFINITY *” (English). Journal of Biological Chemistry 274 (46): 33105–33113. doi:10.1074/jbc.274.46.33105. ISSN 0021-9258. PMID 10551881.
- ^ Jernigan, John A., et al. (2001). “Bioterrorism-Related Inhalational Anthrax: The First 10 Cases Reported in the United States”. Emerging Infectious Diseases 7 (6): 933–44. doi:10.3201/eid0706.010604. PMC 2631903. PMID 11747719.
- ^ Nicholson, Wayne L and Munakata, Nobuo and Horneck, Gerda and Melosh, Henry J and Setlow, Peter (September 2000). “Resistance of Bacillus endospores to extreme terrestrial and extraterrestrial environments”. Microbiology and Molecular Biology Reviews 64 (3): 548–72. doi:10.1128/MMBR.64.3.548-572.2000. PMC 99004. PMID 10974126.
- ^ a b “The Bacillus subtilis endospore: assembly and functions of the multilayered coat”. Nature Reviews. Microbiology 11 (1): 33–44. (January 2013). doi:10.1038/nrmicro2921. PMID 23202530.
- ^ “Bacterial endospores and their significance in stress resistance”. Antonie van Leeuwenhoek 81 (1–4): 27–32. (August 2002). doi:10.1023/A:1020561122764. PMID 12448702.
- ^ “Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal”. Nature 407 (6806): 897–900. (October 2000). Bibcode: 2000Natur.407..897V. doi:10.1038/35038060. PMID 11057666.
- ^ “Revival and identification of bacterial spores in 25- to 40-million-year-old Dominican amber”. Science 268 (5213): 1060–64. (May 1995). Bibcode: 1995Sci...268.1060C. doi:10.1126/science.7538699. PMID 7538699.
- ^ “Row over ancient bacteria” (英語). BBC News. (7 June 2001) 26 April 2020閲覧。
- ^ “The solar UV environment and bacterial spore UV resistance: considerations for Earth-to-Mars transport by natural processes and human spaceflight”. Mutation Research 571 (1–2): 249–64. (April 2005). doi:10.1016/j.mrfmmm.2004.10.012. PMID 15748651.
- ^ “Colonising the galaxy is hard. Why not send bacteria instead?”. The Economist. (12 April 2018). ISSN 0013-0613 26 April 2020閲覧。
- ^ “Virulence Plasmids of the Pathogenic Clostridia”. Microbiology Spectrum 7 (3). (May 2019). doi:10.1128/microbiolspec.GPP3-0034-2018. PMID 31111816.
- ^ “How to: prophylactic interventions for prevention of Clostridioides difficile infection”. Clinical Microbiology and Infection 27 (12): 1777–1783. (July 2021). doi:10.1016/j.cmi.2021.06.037. PMID 34245901.
- ^ “Post-Viking microbiology: new approaches, new data, new insights”. Origins of Life and Evolution of the Biosphere 29 (1): 73–93. (January 1999). Bibcode: 1999OLEB...29...73N. doi:10.1023/A:1006515817767. PMID 11536899.
- ^ “Microbial ecology in the age of genomics and metagenomics: concepts, tools, and recent advances”. Molecular Ecology 15 (7): 1713–31. (June 2006). doi:10.1111/j.1365-294X.2006.02882.x. PMID 16689892.
- ^ “Comparative biochemistry of Archaea and Bacteria”. Current Opinion in Genetics & Development 1 (4): 544–51. (December 1991). doi:10.1016/S0959-437X(05)80206-0. PMID 1822288.
- ^ a b c Microbiology: An Evolving Science (3 ed.). WW Norton & Company. pp. 491–44
- ^ “Photobiology of bacteria”. Antonie van Leeuwenhoek 65 (4): 331–47. (1994). doi:10.1007/BF00872217. PMID 7832590.
- ^ “The Leeuwenhoek Lecture 2000 the natural and unnatural history of methane-oxidizing bacteria”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 360 (1458): 1207–22. (June 2005). doi:10.1098/rstb.2005.1657. PMC 1569495. PMID 16147517.
- ^ “Diazotrophs for Lowering Nitrogen Pollution Crises: Looking Deep Into the Roots”. Frontiers in Microbiology 12: 637815. (2021). doi:10.3389/fmicb.2021.637815. PMC 8180554. PMID 34108945.
- ^ “Nitrogenase gene diversity and microbial community structure: a cross-system comparison”. Environmental Microbiology 5 (7): 539–54. (July 2003). doi:10.1046/j.1462-2920.2003.00451.x. PMID 12823187.
- ^ “Wastewater Treatment using the "Sulfate Reduction, DenitrificationAnammox and Partial Nitrification (SRDAPN)" Process”. Chemosphere 256: 127092. (October 2020). Bibcode: 2020Chmsp.256l7092K. doi:10.1016/j.chemosphere.2020.127092. PMID 32559887.
- ^ “The chemical cycle and bioaccumulation of mercury”. Annual Review of Ecology and Systematics 29: 543–66. (1998). doi:10.1146/annurev.ecolsys.29.1.543.
- ^ “How to define obligatory anaerobiosis? An evolutionary view on the antioxidant response system and the early stages of the evolution of life on Earth”. Free Radical Biology & Medicine 140: 61–73. (August 2019). doi:10.1016/j.freeradbiomed.2019.03.004. PMID 30862543.
- ^ “Control of the bacterial cell cycle by cytoplasmic growth”. Critical Reviews in Microbiology 28 (1): 61–77. (2002). doi:10.1080/1040-840291046696. PMID 12003041.
- ^ Pommerville 2014, p. 138.
- ^ Pommerville 2014, p. 557.
- ^ a b Wheelis 2008, p. 42.
- ^ “Laboratory diagnosis of central nervous system infections”. Infectious Disease Clinics of North America 15 (4): 1047–71. (December 2001). doi:10.1016/S0891-5520(05)70186-0. PMID 11780267.
- ^ “Harmful freshwater algal blooms, with an emphasis on cyanobacteria”. TheScientificWorldJournal 1: 76–113. (April 2001). doi:10.1100/tsw.2001.16. PMC 6083932. PMID 12805693.
- ^ “Synergy and contingency as driving forces for the evolution of multiple secondary metabolite production by Streptomyces species”. Proceedings of the National Academy of Sciences of the United States of America 100 Suppl 2 (90002): 14555–61. (November 2003). Bibcode: 2003PNAS..10014555C. doi:10.1073/pnas.1934677100. PMC 304118. PMID 12970466.
- ^ “Microbial biofilms: from ecology to molecular genetics”. Microbiology and Molecular Biology Reviews 64 (4): 847–67. (December 2000). doi:10.1128/MMBR.64.4.847-867.2000. PMC 99016. PMID 11104821.
- ^ “Quantitative steps in symbiogenesis and the evolution of homeostasis”. Biological Reviews of the Cambridge Philosophical Society 78 (3): 435–63. (August 2003). doi:10.1017/S1464793102006127. PMID 14558592.
- ^ “Lag Phase is a Dynamic, Organized, Adaptive, and Evolvable Period that Prepares Bacteria for Cell Division”. Journal of Bacteriology 201 (7): e00697-18. (2019). doi:10.1128/JB.00697-18. PMC 6416914. PMID 30642990.
- ^ “Individual-based modelling of bacterial cultures to study the microscopic causes of the lag phase”. Journal of Theoretical Biology 241 (4): 939–53. (August 2006). Bibcode: 2006JThBi.241..939P. doi:10.1016/j.jtbi.2006.01.029. PMID 16524598.
- ^ General stress response of Bacillus subtilis and other bacteria. Advances in Microbial Physiology. 44. (2001). pp. 35–91. doi:10.1016/S0065-2911(01)44011-2. ISBN 978-0-12-027744-5. PMID 11407115
- ^ Microbiology: An Evolving Science (3 ed.). WW Norton & Company. p. 143
- ^ Leppänen, Miika; Sundberg, Lotta-Riina; Laanto, Elina; De Freitas Almeida, Gabriel Magno; Papponen, Petri; Maasilta, Ilari J. (2017). “Imaging Bacterial Colonies and Phage-Bacterium Interaction at Sub-Nanometer Resolution Using Helium-Ion Microscopy”. Advanced Biosystems 1 (8): e1700070. doi:10.1002/adbi.201700070. PMID 32646179.
- ^ “The 160-kilobase genome of the bacterial endosymbiont Carsonella”. Science 314 (5797): 267. (October 2006). doi:10.1126/science.1134196. PMID 17038615.
- ^ “Characterisation, genome size and genetic manipulation of the myxobacterium Sorangium cellulosum So ce56”. Archives of Microbiology 178 (6): 484–92. (December 2002). doi:10.1007/s00203-002-0479-2. PMID 12420170.
- ^ “Linear plasmids and chromosomes in bacteria”. Molecular Microbiology 10 (5): 917–22. (December 1993). doi:10.1111/j.1365-2958.1993.tb00963.x. PMID 7934868.
- ^ “The chromosomal DNA of Streptomyces lividans 66 is linear”. Molecular Microbiology 10 (5): 923–33. (December 1993). doi:10.1111/j.1365-2958.1993.tb00964.x. PMID 7934869.
- ^ “Management of multipartite genomes: the Vibrio cholerae model”. Current Opinion in Microbiology 22: 120–26. (December 2014). doi:10.1016/j.mib.2014.10.003. PMID 25460805.
- ^ “Historical events that spawned the field of plasmid biology”. Microbiology Spectrum 2 (5): 3. (October 2014). doi:10.1128/microbiolspec.PLAS-0019-2013. PMID 26104369.
- ^ “Prokaryotic introns and inteins: a panoply of form and function”. Journal of Bacteriology 177 (14): 3897–903. (July 1995). doi:10.1128/jb.177.14.3897-3903.1995. PMC 177115. PMID 7608058.
- ^ “Evolution of mutation rates in bacteria”. Molecular Microbiology 60 (4): 820–27. (May 2006). doi:10.1111/j.1365-2958.2006.05150.x. PMID 16677295.
- ^ “Stress-directed adaptive mutations and evolution”. Molecular Microbiology 52 (3): 643–50. (May 2004). doi:10.1111/j.1365-2958.2004.04012.x. PMID 15101972.
- ^ “DNA uptake during bacterial transformation”. Nature Reviews. Microbiology 2 (3): 241–49. (March 2004). doi:10.1038/nrmicro844. PMID 15083159.
- ^ “Natural genetic transformation: prevalence, mechanisms and function”. Research in Microbiology 158 (10): 767–78. (December 2007). doi:10.1016/j.resmic.2007.09.004. PMID 17997281.
- ^ Bernstein H, Bernstein C, Michod RE (2012).
- ^ “Phages and the evolution of bacterial pathogens: from genomic rearrangements to lysogenic conversion”. Microbiology and Molecular Biology Reviews 68 (3): 560–602, table of contents. (September 2004). doi:10.1128/MMBR.68.3.560-602.2004. PMC 515249. PMID 15353570.
- ^ “Biology of DNA restriction”. Microbiological Reviews 57 (2): 434–50. (June 1993). doi:10.1128/MMBR.57.2.434-450.1993. PMC 372918. PMID 8336674.
- ^ “CRISPR provides acquired resistance against viruses in prokaryotes”. Science 315 (5819): 1709–12. (March 2007). Bibcode: 2007Sci...315.1709B. doi:10.1126/science.1138140. PMID 17379808.
- ^ “Small CRISPR RNAs guide antiviral defense in prokaryotes”. Science 321 (5891): 960–64. (August 2008). Bibcode: 2008Sci...321..960B. doi:10.1126/science.1159689. PMC 5898235. PMID 18703739.
- ^ “Adaptive value of sex in microbial pathogens”. Infection, Genetics and Evolution 8 (3): 267–85. (May 2008). doi:10.1016/j.meegid.2008.01.002. PMID 18295550.
- ^ “Antibiotic-induced lateral transfer of antibiotic resistance”. Trends in Microbiology 12 (9): 401–14. (September 2004). doi:10.1016/j.tim.2004.07.003. PMID 15337159.
- ^ “Genetic exchange between bacteria in the environment”. Plasmid 42 (2): 73–91. (September 1999). doi:10.1006/plas.1999.1421. PMID 10489325.
- ^ a b “Electron microscopic observations of prokaryotic surface appendages”. Journal of Microbiology (Seoul, Korea) 55 (12): 919–26. (December 2017). doi:10.1007/s12275-017-7369-4. PMID 29214488.
- ^ “The bacterial flagellum: reversible rotary propellor and type III export apparatus”. Journal of Bacteriology 181 (23): 7149–53. (December 1999). doi:10.1128/JB.181.23.7149-7153.1999. PMC 103673. PMID 10572114.
- ^ “Collective bacterial dynamics revealed using a three-dimensional population-scale defocused particle tracking technique”. Applied and Environmental Microbiology 72 (7): 4987–94. (July 2006). Bibcode: 2006ApEnM..72.4987W. doi:10.1128/AEM.00158-06. PMC 1489374. PMID 16820497.
- ^ “Electron microscopic observations of prokaryotic surface appendages”. Journal of Microbiology (Seoul, Korea) 55 (12): 919–26. (December 2017). doi:10.1007/s12275-017-7369-4. PMID 29214488.
- ^ Mattick, John S (2002). “Type IV Pili and Twitching Motility”. Annual Review of Microbiology 56: 289–314. doi:10.1146/annurev.micro.56.012302.160938. PMID 12142488.
- ^ “Pilus retraction powers bacterial twitching motility”. Nature 407 (6800): 98–102. (September 2000). Bibcode: 2000Natur.407...98M. doi:10.1038/35024105. PMID 10993081.
- ^ “Chemotaxis-guided movements in bacteria”. Critical Reviews in Oral Biology and Medicine 15 (4): 207–20. (July 2004). doi:10.1177/154411130401500404. PMID 15284186.
- ^ “Bacterial energy taxis: a global strategy?”. Archives of Microbiology 192 (7): 507–20. (July 2010). doi:10.1007/s00203-010-0575-7. PMC 2886117. PMID 20411245.
- ^ “Magneto-aerotaxis in marine coccoid bacteria”. Biophysical Journal 73 (2): 994–1000. (August 1997). Bibcode: 1997BpJ....73..994F. doi:10.1016/S0006-3495(97)78132-3. PMC 1180996. PMID 9251816.
- ^ “Signaling in myxobacteria”. Annual Review of Microbiology 58: 75–98. (2004). doi:10.1146/annurev.micro.58.030603.123620. PMID 15487930.
- ^ “Uncovering the mystery of gliding motility in the myxobacteria”. Annual Review of Genetics 45: 21–39. (2011). doi:10.1146/annurev-genet-110410-132547. PMC 3397683. PMID 21910630.
- ^ “Actin-based motility of intracellular microbial pathogens”. Microbiology and Molecular Biology Reviews 65 (4): 595–626, table of contents. (December 2001). doi:10.1128/MMBR.65.4.595-626.2001. PMC 99042. PMID 11729265.
- ^ “Bacterial Semiochemicals and Transkingdom Interactions with Insects and Plants”. Insects 10 (12): 441. (December 2019). doi:10.3390/insects10120441. PMC 6955855. PMID 31817999.
- ^ “Thinking about bacterial populations as multicellular organisms”. Annual Review of Microbiology 52: 81–104. (1998). doi:10.1146/annurev.micro.52.1.81. PMID 9891794.
- ^ “Microbial biofilms”. Annual Review of Microbiology 49: 711–45. (1995). doi:10.1146/annurev.mi.49.100195.003431. PMID 8561477.
- ^ “Thinking about bacterial populations as multicellular organisms”. Annual Review of Microbiology 52: 81–104. (1998). doi:10.1146/annurev.micro.52.1.81. PMID 9891794.
- ^ “Microbial biofilms”. Annual Review of Microbiology 49: 711–45. (1995). doi:10.1146/annurev.mi.49.100195.003431. PMID 8561477.
- ^ “Signaling systems in oral bacteria”. Advances in Experimental Medicine and Biology 1197: 27–43. (2019). doi:10.1007/978-3-030-28524-1_3. ISBN 978-3-030-28523-4. PMID 31732932.
- ^ “Bacterial Quorum sensing and microbial community interactions”. mBio 9 (3). (May 2018). doi:10.1128/mBio.02331-17. PMC 5964356. PMID 29789364.
- ^ “Quorum sensing in bacteria”. Annual Review of Microbiology 55: 165–99. (2001). doi:10.1146/annurev.micro.55.1.165. PMID 11544353.
- ^ Zhu, Qiyun; Mai, Uyen; Pfeiffer, Wayne; Janssen, Stefan; Asnicar, Francesco; Sanders, Jon G.; Belda-Ferre, Pedro; Al-Ghalith, Gabriel A. et al. (2019). “Phylogenomics of 10,575 genomes reveals evolutionary proximity between domains Bacteria and Archaea”. Nature Communications 10 (1): 5477. Bibcode: 2019NatCo..10.5477Z. doi:10.1038/s41467-019-13443-4. PMC 6889312. PMID 31792218.
- ^ “Laboratory diagnosis of central nervous system infections”. Infectious Disease Clinics of North America 15 (4): 1047–71. (December 2001). doi:10.1016/S0891-5520(05)70186-0. PMID 11780267.
- ^ “Lateral gene transfer and the origins of prokaryotic groups”. Annual Review of Genetics 37: 283–328. (2003). doi:10.1146/annurev.genet.37.050503.084247. PMID 14616063.
- ^ “The winds of (evolutionary) change: breathing new life into microbiology”. Journal of Bacteriology 176 (1): 1–6. (January 1994). doi:10.2172/205047. PMC 205007. PMID 8282683.
- ^ “IJSEM Home”. Ijs.sgmjournals.org (28 October 2011). 19 October 2011時点のオリジナルよりアーカイブ。4 November 2011閲覧。
- ^ “Bergey's Manual Trust”. Bergeys.org. 7 November 2011時点のオリジナルよりアーカイブ。4 November 2011閲覧。
- ^ “The changing landscape of microbial biodiversity exploration and its implications for systematics”. Systematic and Applied Microbiology 38 (4): 231–36. (June 2015). doi:10.1016/j.syapm.2015.03.003. PMID 25921438.
- ^ "Schizomycetes.”
- ^ “Staphylococcus epidermidis-Skin friend or foe?”. PLOS Pathogens 16 (11): e1009026. (November 2020). doi:10.1371/journal.ppat.1009026. PMC 7660545. PMID 33180890.
- ^ a b Hall 2008, p. 145.
- ^ “The natural evolutionary relationships among prokaryotes”. Critical Reviews in Microbiology 26 (2): 111–31. (2000). doi:10.1080/10408410091154219. PMID 10890353.
- ^ “The uncultured microbial majority”. Annual Review of Microbiology 57: 369–94. (2003). doi:10.1146/annurev.micro.57.030502.090759. PMID 14527284.
- ^ “Evolutionary aspects of whole-genome biology”. Current Opinion in Structural Biology 15 (3): 248–53. (June 2005). doi:10.1016/j.sbi.2005.04.001. PMID 15963888.
- ^ Pommerville 2014, p. 15−31.
- ^ Castelle, C.J., Banfield, J.F. (2018-03-08). “Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life”. Cell 172 (6): 1181-1197. doi:10.1016/j.cell.2018.02.016. PMID 29522741.
- ^ a b Krasner 2014, p. 77.
- ^ Gram, HC (1884). “Über die isolierte Färbung der Schizomyceten in Schnitt- und Trockenpräparaten”. Fortschr. Med. 2: 185–89.
- ^ “Detection of infection or infectious agents by use of cytologic and histologic stains”. Clinical Microbiology Reviews 9 (3): 382–404. (July 1996). doi:10.1128/CMR.9.3.382. PMC 172900. PMID 8809467.
- ^ “Assessment and Comparison of Molecular Subtyping and Characterization Methods for Salmonella”. Frontiers in Microbiology 10: 1591. (2019). doi:10.3389/fmicb.2019.01591. PMC 6639432. PMID 31354679.
- ^ Krasner 2014, p. 87–89.
- ^ “Laboratory diagnosis of central nervous system infections”. Infectious Disease Clinics of North America 15 (4): 1047–71. (December 2001). doi:10.1016/S0891-5520(05)70186-0. PMID 11780267.
- ^ “Clinical importance of blood cultures”. Clinics in Laboratory Medicine 14 (1): 9–16. (March 1994). doi:10.1016/S0272-2712(18)30390-1. PMID 8181237.
- ^ “Laboratory Methods in Molecular Epidemiology: Bacterial Infections”. Microbiology Spectrum 6 (6). (November 2018). doi:10.1128/microbiolspec.AME-0004-2018. PMID 30387415.
- ^ “The Loop-Mediated Isothermal Amplification Technique in Periodontal Diagnostics: A Systematic Review”. Journal of Clinical Medicine 10 (6): 1189. (March 2021). doi:10.3390/jcm10061189. PMC 8000232. PMID 33809163.
- ^ “MALDI-TOF Mass Spectroscopy Applications in Clinical Microbiology”. Advances in Pharmacological and Pharmaceutical Sciences 2021: 9928238. (2021). doi:10.1155/2021/9928238. PMC 8121603. PMID 34041492.
- ^ “Novel Microbial Diversity and Functional Potential in the Marine Mammal Oral Microbiome”. Current Biology 27 (24): 3752–62. (2017). doi:10.1016/j.cub.2017.10.040. PMID 29153320.
- ^ “The role of DNA amplification technology in the diagnosis of infectious diseases”. CMAJ 163 (3): 301–09. (August 2000). doi:10.1016/s1381-1169(00)00220-x. PMC 80298. PMID 10951731.
- ^ “The viable but nonculturable state in bacteria”. Journal of Microbiology 43 Spec No: 93–100. (February 2005). PMID 15765062.
- ^ “Number of published names”. List of Prokaryotic names with Standing in Nomenclature (8 December 2011). 19 January 2012時点のオリジナルよりアーカイブ。10 December 2011閲覧。
- ^ “Estimating prokaryotic diversity and its limits”. Proceedings of the National Academy of Sciences of the United States of America 99 (16): 10494–99. (August 2002). Bibcode: 2002PNAS...9910494C. doi:10.1073/pnas.142680199. PMC 124953. PMID 12097644.
- ^ “Status of the microbial census”. Microbiology and Molecular Biology Reviews 68 (4): 686–91. (December 2004). doi:10.1128/MMBR.68.4.686-691.2004. PMC 539005. PMID 15590780.
- ^ a b Hug, Laura A.; Baker, Brett J.; Anantharaman, Karthik; Brown, Christopher T.; Probst, Alexander J.; Castelle, Cindy J.; Butterfield, Cristina N.; Hernsdorf, Alex W. et al. (2016-05). “A new view of the tree of life” (英語). Nature Microbiology 1 (5): 16048. doi:10.1038/nmicrobiol.2016.48. ISSN 2058-5276.
- ^ Fisher, Bruce; Harvey, Richard P; Champe, Pamela C (2007). “Chapter 33”. Lippincott's Illustrated Reviews: Microbiology (Lippincott's Illustrated Reviews Series). Hagerstwon, MD: Lippincott Williams & Wilkins. pp. 367–92. ISBN 978-0-7817-8215-9
- ^ “Rethinking "mutualism" in diverse host-symbiont communities”. BioEssays 38 (1): 100–8. (January 2016). doi:10.1002/bies.201500074. PMID 26568407.
- ^ Harper, Douglas. "commensalism". Online Etymology Dictionary.
- ^ “A dynamic partnership: celebrating our gut flora”. Anaerobe 11 (5): 247–51. (October 2005). doi:10.1016/j.anaerobe.2005.05.001. PMID 16701579.
- ^ “Commensal Bacteria: An Emerging Player in Defense Against Respiratory Pathogens”. Frontiers in Immunology 10: 1203. (2019). doi:10.3389/fimmu.2019.01203. PMC 6554327. PMID 31214175.
- ^ “Normal flora and mucosal immunity of the head and neck”. Infectious Disease Clinics of North America 2 (1): 1–19. (March 1988). doi:10.1016/S0891-5520(20)30163-X. PMID 3074102.
- ^ “Commensal bacteria and fungi differentially regulate tumor responses to radiation therapy”. Cancer Cell 39 (9): 1202–1213.e6. (July 2021). doi:10.1016/j.ccell.2021.07.002. PMID 34329585.
- ^ “MALT Lymphoma of the Urinary Bladder Shows a Dramatic Female Predominance, Uneven Geographic Distribution, and Possible Infectious Etiology”. Research and Reports in Urology 13: 49–62. (2021). doi:10.2147/RRU.S283366. PMC 7873029. PMID 33575225.
- ^ “Infective endocarditis in paediatric population”. European Journal of Pediatrics 180 (10): 3089–3100. (April 2021). doi:10.1007/s00431-021-04062-7. PMID 33852085.
- ^ “Predatory prokaryotes: an emerging research opportunity”. Journal of Molecular Microbiology and Biotechnology 4 (5): 467–77. (September 2002). PMID 12432957.
- ^ “Experimental social evolution with Myxococcus xanthus”. Antonie van Leeuwenhoek 81 (1–4): 155–64. (August 2002). doi:10.1023/A:1020546130033. PMID 12448714.
- ^ “Bacterial Predation on Cyanobacteria”. Microbial Physiology 31 (2): 99–108. (May 2021). doi:10.1159/000516427. ISSN 2673-1665. PMID 34010833.
- ^ “Bacterial predators”. Current Biology 19 (2): R55–56. (January 2009). doi:10.1016/j.cub.2008.10.043. PMID 19174136.
- ^ “Exocellular electron transfer in anaerobic microbial communities”. Environmental Microbiology 8 (3): 371–82. (March 2006). doi:10.1111/j.1462-2920.2006.00989.x. PMID 16478444.
- ^ “Cross-protection from hydrogen peroxide by helper microbes: the impacts on the cyanobacterium Prochlorococcus and other beneficiaries in marine communities”. Environmental Microbiology Reports 10 (4): 399–411. (August 2018). doi:10.1111/1758-2229.12625. PMID 29411546.
- ^ “Microbial co-operation in the rhizosphere”. Journal of Experimental Botany 56 (417): 1761–78. (July 2005). doi:10.1093/jxb/eri197. PMID 15911555.
- ^ “The gut flora as a forgotten organ”. EMBO Reports 7 (7): 688–93. (July 2006). doi:10.1038/sj.embor.7400731. PMC 1500832. PMID 16819463.
- ^ “A microbial world within us”. Molecular Microbiology 59 (6): 1639–50. (March 2006). doi:10.1111/j.1365-2958.2006.05056.x. PMID 16553872.
- ^ “Lactic acid bacteria and human health”. Annals of Medicine 22 (1): 37–41. (February 1990). doi:10.3109/07853899009147239. PMID 2109988.
- ^ “Probiotics that modify disease risk”. The Journal of Nutrition 135 (5): 1294–98. (May 2005). doi:10.1093/jn/135.5.1294. PMID 15867327.
- ^ “Vitamin B12 sources and microbial interaction”. Experimental Biology and Medicine (Maywood, N.J.) 243 (2): 148–58. (January 2018). doi:10.1177/1535370217746612. PMC 5788147. PMID 29216732.
- ^ Pommerville 2014, pp. 16–21.
- ^ Clark 2010, p. 215.
- ^ Wheelis 2008, p. 44.
- ^ Clark 2010, pp. 30, 195, 233, 236.
- ^ “Helicobacter pylori infection causes both protective and deleterious effects in human health and disease”. Genes and Immunity 22 (4): 218–226. (July 2021). doi:10.1038/s41435-021-00146-4. PMC 8390445. PMID 34244666.
- ^ “40 years of veterinary papers in JAC – what have we learnt?”. The Journal of Antimicrobial Chemotherapy 71 (10): 2681–90. (October 2016). doi:10.1093/jac/dkw363. PMID 27660260.
- ^ “Optimal antimicrobial therapy for sepsis”. American Journal of Health-System Pharmacy 59 Suppl 1: S13–19. (February 2002). doi:10.1093/ajhp/59.suppl_1.S13. PMID 11885408.
- ^ “Chlamydia pneumoniae and atherosclerosis”. Cellular Microbiology 6 (2): 117–27. (February 2004). doi:10.1046/j.1462-5822.2003.00352.x. PMID 14706098.
- ^ “Diseases associated with immunosuppression”. Environmental Health Perspectives 43: 9–19. (February 1982). doi:10.2307/3429162. JSTOR 3429162. PMC 1568899. PMID 7037390.
- ^ “Microbiology of early CF lung disease”. Paediatric Respiratory Reviews 5 Suppl A: S367–69. (2004). doi:10.1016/S1526-0542(04)90065-6. PMID 14980298.
- ^ Pommerville 2014, p. 118.
- ^ a b Pommerville 2014, pp. 646–47.
- ^ Krasner 2014, pp. 165, 369.
- ^ “Ribosomal crystallography: initiation, peptide bond formation, and amino acid polymerization are hampered by antibiotics”. Annual Review of Microbiology 58: 233–51. (2004). doi:10.1146/annurev.micro.58.030603.123822. PMID 15487937.
- ^ “Agricultural use of antibiotics and the evolution and transfer of antibiotic-resistant bacteria”. CMAJ 159 (9): 1129–36. (November 1998). PMC 1229782. PMID 9835883.
- ^ “Disinfection Processes”. Water Environment Research 89 (10): 1206–44. (October 2017). doi:10.2175/106143017X15023776270278. PMID 28954657.
- ^ “Major technological advances and trends in cheese”. Journal of Dairy Science 89 (4): 1174–78. (April 2006). doi:10.3168/jds.S0022-0302(06)72186-5. PMID 16537950.
- ^ Krasner 2014, pp. 25–26.
- ^ “Bioremediation of oil by marine microbial mats”. International Microbiology 5 (4): 189–93. (December 2002). doi:10.1007/s10123-002-0089-5. PMID 12497184.
- ^ “Biofiltration methods for the removal of phenolic residues”. Applied Biochemistry and Biotechnology 129–132 (1–3): 130–52. (2006). doi:10.1385/ABAB:129:1:130. PMID 16915636.
- ^ “Production of fine chemicals using biocatalysis”. Current Opinion in Biotechnology 10 (6): 595–603. (December 1999). doi:10.1016/S0958-1669(99)00040-3. PMID 10600695.
- ^ “Why Bacillus thuringiensis insecticidal toxins are so effective: unique features of their mode of action”. FEMS Microbiology Letters 195 (1): 1–8. (February 2001). doi:10.1111/j.1574-6968.2001.tb10489.x. PMID 11166987.
- ^ “Susceptibility of adult Coccinella septempunctata (Coleoptera: Coccinellidae) to insecticides with different modes of action”. Pest Management Science 62 (7): 651–54. (July 2006). doi:10.1002/ps.1221. PMID 16649191.
- ^ “Bacterial insecticidal toxins”. Critical Reviews in Microbiology 30 (1): 33–54. (2004). doi:10.1080/10408410490270712. PMID 15116762.
- ^ “A functional update of the Escherichia coli K-12 genome”. Genome Biology 2 (9): RESEARCH0035. (2001). doi:10.1186/gb-2001-2-9-research0035. PMC 56896. PMID 11574054.
- ^ “Global organization of metabolic fluxes in the bacterium Escherichia coli”. Nature 427 (6977): 839–43. (February 2004). arXiv:q-bio/0403001. Bibcode: 2004Natur.427..839A. doi:10.1038/nature02289. PMID 14985762.
- ^ “An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR)”. Genome Biology 4 (9): R54. (2003). doi:10.1186/gb-2003-4-9-r54. PMC 193654. PMID 12952533.
- ^ “Therapeutic insulins and their large-scale manufacture”. Applied Microbiology and Biotechnology 67 (2): 151–59. (April 2005). doi:10.1007/s00253-004-1809-x. PMID 15580495.
- ^ “Manufacturing of recombinant therapeutic proteins in microbial systems”. Biotechnology Journal 1 (2): 164–86. (February 2006). doi:10.1002/biot.200500051. PMID 16892246.
- ^ “Diversity of phage types among archived cultures of the Demerec collection of Salmonella enterica serovar Typhimurium strains”. Applied and Environmental Microbiology 70 (2): 664–69. (February 2004). Bibcode: 2004ApEnM..70..664R. doi:10.1128/aem.70.2.664-669.2004. PMC 348941. PMID 14766539.
- ^ Wheelis 2008.
- ^ Asimov's Biographical Encyclopedia of Science and Technology (2nd ed.). Garden City, NY: Doubleday and Company. (1982). p. 143]
- ^ Pommerville 2014, p. 7.
- ^ “The Status of the Generic Term Bacterium Ehrenberg 1828”. Journal of Bacteriology 31 (5): 517–18. (May 1936). doi:10.1128/jb.31.5.517-518.1936. PMC 543738. PMID 16559906.
- ^ (ドイツ語) Dritter Beitrag zur Erkenntniss grosser Organisation in der Richtung des kleinsten Raumes. [Third contribution to the knowledge of great organization in the direction of the smallest space]. Berlin: Physikalische Abhandlungen der Koeniglichen Akademie der Wissenschaften. (1835). pp. 143–336
- ^ “Pasteur's Papers on the Germ Theory”. LSU Law Center's Medical and Public Health Law Site, Historic Public Health Articles. 18 December 2006時点のオリジナルよりアーカイブ。23 November 2006閲覧。
- ^ ‘Wash your hands’ was once controversial medical advice, National Geographic.
- ^ “The Nobel Prize in Physiology or Medicine 1905”. Nobelprize.org. 10 December 2006時点のオリジナルよりアーカイブ。22 November 2006閲覧。
- ^ “HIV causes AIDS: Koch's postulates fulfilled”. Current Opinion in Immunology 8 (5): 613–18. (October 1996). doi:10.1016/S0952-7915(96)80075-6. PMID 8902385.
- ^ Chung. “Ferdinand Julius Cohn (1828–1898): Pioneer of Bacteriology”. Department of Microbiology and Molecular Cell Sciences, The University of Memphis. 27 July 2011時点のオリジナルよりアーカイブ。2019年3月23日閲覧。
- ^ Drews, Gerhart (1999). “Ferdinand Cohn, a founder of modern microbiology”. ASM News 65 (8): 547–52.
- ^ “Of blood, inflammation and gunshot wounds: the history of the control of sepsis”. The Australian and New Zealand Journal of Surgery 70 (12): 855–61. (December 2000). doi:10.1046/j.1440-1622.2000.01983.x. PMID 11167573.
- ^ “Paul Ehrlich's magic bullets”. The New England Journal of Medicine 350 (11): 1079–80. (March 2004). doi:10.1056/NEJMp048021. PMID 15014180.
- ^ “Biography of Paul Ehrlich”. Nobelprize.org. 28 November 2006時点のオリジナルよりアーカイブ。26 November 2006閲覧。
- ^ “Phylogenetic structure of the prokaryotic domain: the primary kingdoms”. Proceedings of the National Academy of Sciences of the United States of America 74 (11): 5088–90. (November 1977). Bibcode: 1977PNAS...74.5088W. doi:10.1073/pnas.74.11.5088. PMC 432104. PMID 270744.
- ^ Zaremba-Niedzwiedzka, Katarzyna; Caceres, Eva F.; Saw, Jimmy H.; Bäckström, Disa; Juzokaite, Lina; Vancaester, Emmelien; Seitz, Kiley W.; Anantharaman, Karthik et al. (2017-01). “Asgard archaea illuminate the origin of eukaryotic cellular complexity” (英語). Nature 541 (7637): 353–358. doi:10.1038/nature21031. ISSN 0028-0836.
