翻訳 (生物学)

(上) 開始段階: メチオニンとそのアンチコドンを輸送するイニシエーターtRNAが、リボソームのP部位でmRNAの開始コドンAUGと遭遇する。
(下) 伸長段階: リボソームが5'から3'方向に移動する。そのとき、ペプチド結合でP部位のtRNAに結合したアミノ酸がA部位のtRNAに結合し、mRNA上のコドンに基づいて長いアミノ酸鎖を合成する。リボソームが移動する際、tRNAはEサイトを通ってリボソームから離れ、新しいtRNAがA部位に入って、アミノ酸鎖の伸長を継続する。

①開始 (Initiation): ポリメラーゼがDNA鎖に結合してから、リボソーム小サブユニットがDNAに結合するまで。
②伸長 (Elongation): 大サブユニットが結合すると伸長が開始する。
③終止 (Termination): 伸長の過程が終了する。
分子生物学や遺伝学において、翻訳(ほんやく、英: translation)とは、細胞質または小胞体でリボソームがタンパク質を合成する過程であり、これは細胞の核でDNAを元にRNAが合成される転写に続くものである。この一連の過程は、遺伝子発現と呼ばれる。
概説
[編集]翻訳では、メッセンジャーRNA(mRNA)が核の外にあるリボソームで解読され、特定のアミノ酸鎖(ポリペプチドともよぶ)が作られる。その後、ポリペプチドは活性タンパク質に折り畳まれ、細胞内でその役割を果たす。リボソームは、mRNAコドンに相補的なtRNAアンチコドン配列の結合を導くことによって解読を進める。転移RNA(tRNA)は特定のアミノ酸を輸送し、mRNAがリボソームを通過して「読み取られる」ときにそのアミノ酸がポリペプチドに連結される。
翻訳は次の3つの段階で進行する。
- 開始: 標的mRNAの周囲にリボソームが集結する。最初のtRNAがmRNAの開始コドンに結合する。
- 伸長: リボソーム小サブユニットによって確認された(収容)tRNAは、運んだアミノ酸をリボソーム大サブユニットに移し、その前に収容されたアミノ酸に結合する(ペプチド転移)。その後、リボソームは次のmRNAコドンに移動してプロセスを継続し(転座)、アミノ酸の鎖を形成する。
- 終結: 終止コドンに到達すると、リボソームはポリペプチドを放出する。リボソーム複合体はそのまま残り、次に翻訳されるmRNAへ移動する。
原核生物(細菌および古細菌)の場合、翻訳は細胞質基質で行われ、リボソームの大サブユニットと小サブユニットがmRNAに結合する。真核生物の場合、翻訳は細胞質内または小胞体の膜を越えて共翻訳転座と呼ばれる過程で行われる。共翻訳転座では、リボソームとmRNAの複合体全体が粗面小胞体(ER)の外膜に結合し、新しいタンパク質が合成されてER内に放出される。新しく作られたポリペプチドは、将来の小胞輸送や細胞外への分泌のためにER内に貯蔵されるか、または直ちに分泌される。
転移RNA(tRNA)、リボソームRNA(rRNA)、核内低分子RNA(snRNA)など、多くの種類の転写RNAはタンパク質に翻訳されない。
多くの抗生物質は、翻訳を阻害することで働く。たとえば、アニソマイシン、シクロヘキシミド、クロラムフェニコール、テトラサイクリン、ストレプトマイシン、エリスロマイシン、およびピューロマイシンがある。細菌などの原核生物のリボソームは真核生物のリボソームと構造が異なるため、抗生物質は真核生物の宿主細胞に害を与えることなく、感染した細菌を特異的に標的にすることができる。
基本的な機構
[編集]

タンパク質を生成する基本的な過程は、タンパク質の末端にアミノ酸を1つずつ付加することである。この働きは、リボソームによって行われる[1]。リボソームは、小サブユニットと大サブユニットの2つのサブユニットから構成されている。これらのサブユニットは、mRNAがタンパク質に翻訳される前に一体となり、翻訳が行われてポリペプチドが生成される場所を提供する。ポリペプチドに付加するアミノ酸の種類と配列はmRNA分子によって決定される[2]。付加された各アミノ酸は、mRNA上の3連ヌクレオチド配列(トリプレットまたは三連符という)と符合する。このようなトリプレットの組み合わせ可能なそれぞれについて、対応するアミノ酸が認められる。鎖に追加された連続したアミノ酸は、mRNAの連続したヌクレオチド・トリプレットに符合する。このようにして、mRNA鎖のヌクレオチド配列が鋳型となり、生成されるアミノ酸鎖のアミノ酸の配列を決定する[3]。アミノ酸の付加はペプチドのC末端で起こるので、翻訳はアミンからカルボキシルへ向かうと呼ばれる[4]。
リボヌクレオチドの配列としてコード化された遺伝情報は、mRNAによって染色体からリボソームへ送られる。リボヌクレオチドは、コドンと呼ばれるヌクレオチド・トリプレットの配列として翻訳機構によって読み取られる。これらのトリプレットはそれぞれ特定のアミノ酸をコードしている。
リボソーム分子は、このコードを特定のアミノ酸の配列へ翻訳する。リボソームは、rRNAとタンパク質からなる多サブユニット構造体であり、アミノ酸をタンパク質に組み立てる「工場」の役割を担っている。tRNAは、アミノ酸をリボソームへ輸送する小さなノンコーディングRNA鎖(74-93ヌクレオチド)であり、アミノ酸が結合する部位と、アンチコドンと呼ばれる部位を持っている。アンチコドンはRNAのトリプレットで、積荷のアミノ酸をコードするmRNAトリプレットに相補的になっている。
酵素であるアミノアシルtRNA合成酵素は、特定のtRNAと、そのアンチコドン配列に対応するアミノ酸との結合を触媒する。この反応の産物はアミノアシルtRNAである。細菌の場合、このアミノアシルtRNAは転写因子EF-Tuによってリボソームへ輸送され、そこでmRNAコドンと特定のtRNAアンチコドンが相補的塩基対を形成することで符合する。アミノアシルtRNA合成酵素が誤ったアミノ酸とtRNAとの対合を形成すると、誤った結合のアミノアシルtRNAが生成し、その結果、タンパク質の対応する位置に不適切なアミノ酸が生じる可能性がある。このような遺伝暗号の「誤訳」は、ほとんどの生物で低いレベルで自然に起こるが[5]、ある種の細胞環境では、mRNA解読の許容範囲が増して細胞の利益につながる場合もある。
リボソームには、tRNAと結合する部位が2つある。それぞれ、アミノアシル部位(Aと略す)、ペプチジル部位/出口部位(P/Eと略す)である。リボソームはmRNAの3'末端に向かって移動するため、mRNAに対して3つの部位は5'から3'へE-P-Aの順で方向付いている(最初の図を参照)。A部位では、入ってくるtRNAをmRNA上の相補的なコドンと結合する。P/E部位では、そのtRNAと成長中のポリペプチド鎖を保持する。アミノアシルtRNAがmRNA上の対応するコドンと最初に結合するのはA部位で行われる。次に、A部位にあるtRNAのアミノ酸と、P/E部位にある充填tRNA(charged tRNA)のアミノ酸との間にペプチド結合が形成される。成長するポリペプチド鎖はA部位のtRNAに転移する。P/E部位のアミノ酸を持たないtRNAが移動して転座が起こる。A部位にあったtRNAは、ポリペプチド鎖と結合した状態でP/E部位に移動し、そのtRNAは離れて別のアミノアシルtRNAがA部位に入り、この過程が繰り返される[6]。
新しいアミノ酸が鎖に付加され、tRNAがリボソームから細胞質へ遊離した後、トランスロカーゼ(転移酵素)であるEF-G(細菌の場合)またはa/eEF-2(真核生物および古細菌の場合)に結合したグアノシン三リン酸(GTP)の加水分解で供給されるエネルギーによって、リボソームは3'末端の方向へ1コドン移動する。タンパク質の翻訳に必要なエネルギーはかなり多い。n個のアミノ酸を含むタンパク質の場合、翻訳に必要な高エネルギーリン酸結合の数は4n-1個である[7]。翻訳速度はさまざまで、原核細胞は(1秒間に17-21個のアミノ酸残基)真核細胞(1秒間に6-9個のアミノ酸残基)よりもかなり速い[8]。
一般に、リボソームは正確でプロセッシブ(mRNAに沿って連続してアミノ酸鎖を合成すること)な機械と考えられているが、翻訳プロセスは誤りを前提としていて、tRNAが誤ったコドンに結合したり誤ったアミノ酸に結合するために、誤ったタンパク質の合成や翻訳の早期断念につながる可能性がある[9]。タンパク質の合成における誤り率は、実験条件にもよるが、1/105から1/103の間でアミノ酸が誤って取り込まれると推定されている[10]。一方、翻訳を早期に断念する割合は、翻訳されたコドンあたり10−4イベントのオーダーであると推定されている[11]。アミノアシルトランスフェラーゼによって、正しいアミノ酸と正しい転移RNA(tRNA)が共有結合する。アミノ酸はカルボキシル基によってtRNAの3'OHにエステル結合する。アミノ酸が結合したtRNAは「チャージしている(charged)」と呼ばれる。タンパク質合成の開始過程では、開始因子(IF)の助けを借りて、リボソーム小サブユニットがmRNAの5'末端に結合する。細菌および少数種の古細菌の場合、開始には、シャイン・ダルガノ配列と呼ばれるmRNA上のプリンに富む開始配列の認識を伴う。シャイン・ダルガノ配列は、30Sリボソームサブユニットの16S rRNA部分の3'末端にある相補的なピリミジンに富む配列と結合する。これらの相補的な配列が結合することによって、30SリボソームサブユニットがmRNAと結合し、開始コドンがP部位の30S部分に配置されるように確実に整列する。mRNAと30Sサブユニットが適切に結合すると、開始因子がイニシエーターtRNA・アミノ酸複合体のfMet-tRNAを30SのP部位に輸送する。50Sサブユニットが30Sサブユニットに結合し、活性型70Sリボソームが形成されると開始段階が終了する[12]。ポリペプチドの終止は、リボソームのA部位がmRNA上の終止コドン(UAA、UAG、UGA)に占有されたときに起こり、タンパク質の一次構造が形成される。tRNAは通常、終止コドンを認識することも、結合することもできない。その代わりに終止コドンは、リボソームのペプチジルトランスフェラーゼ活性中心[1]からポリペプチド鎖を加水分解することによって、リボソームとmRNA複合体全体の分解を促す終結因子というタンパク質(RF1およびRF2)の結合を誘導する[13][14]。ある種の薬物やmRNA上の特殊な配列モチーフによってリボソーム構造が変化し、終結因子の代わりに、近同族性tRNAが終止コドンに結合することがある。このような「翻訳リードスルー(translational readthrough)」の場合、リボソームが次の終止コドンに遭遇するまで翻訳が継続される[15]。
真核生物、原核生物のいずれにおいても翻訳過程は高度に制御されている。翻訳が制御されると、細胞の代謝や増殖状態と密接に関連するタンパク質合成の全体的な速度に影響を与える可能性がある。さらに、最近の研究では、遺伝的な相違や、その後のmRNAとしての発現も、RNA特異的に翻訳速度に影響を与える可能性があることが明らかになった[16]。
臨床的意義
[編集]翻訳制御は、がんの発生と生存にとって重要な要素である。がん細胞は、遺伝子発現の翻訳段階を随時制御する必要があるが、なぜ転写のような段階よりも翻訳が標的とされるのか、その理由は完全には理解されていない。がん細胞は、しばしば遺伝的に改変された翻訳因子を持っているが、既存の翻訳因子の量を変化させることの方がはるかに一般的である[17]。RAS-MAPK、PI3K/ACT/mTOR、MYC、およびWNT-β-カテニン経路など、いくつかの主要な発がん性シグナル伝達経路は、最終的に翻訳を介してゲノムを再プログラムする[18]。また、がん細胞は細胞ストレスに適応するために翻訳を制御している。ストレスがかかると、細胞はそのストレスを緩和して存続を図るためのmRNAを翻訳する。この例として、さまざまながんにおけるAMPKの発現がある。AMPKの活性化は、栄養不足によって引き起こされるアポトーシス(プログラム細胞死)から、がん細胞が最終的に回避するためのカスケードを引き起こす。将来のがん治療では、細胞の翻訳機構を妨害することで、がんの下流効果に対抗できる可能性がある[17]。
翻訳の数理モデル
[編集]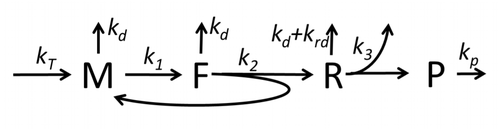
転写 - 翻訳プロセスは、最も基本的な「基礎」過程のみ言及して、次のような構成で記述される。
- mRNA分子の生成(スプライシングを含む)。
- 開始因子の助けを借りた各々の分子の導入(たとえば(一般に必要ではないが)環状化の段階を含むことがある)。
- 翻訳の開始、リボソーム小サブユニットの動員。
- 完全なリボソームの構築。
- 伸長、すなわちタンパク質の生産を伴うmRNAに沿ったリボソームの移動。
- 翻訳の終了。
- mRNA分子の分解。
- タンパク質の分解。
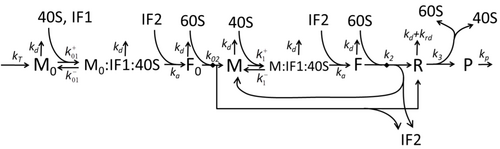
翻訳によってアミノ酸が組み合わさりタンパク質が生成される過程は、その確率的側面を考慮した初期の詳細な動力学モデルから始まり[20]、コンピュータシミュレーションを使用したさまざまな物理モデルの対象となってきた。過去40年間で、化学反応速度論に基づくタンパク質合成モデルが数多く開発され、解析されてきた[21][22]。化学反応速度論のほかにも、タンパク質合成の詳細な動力学やその一部の段階をモデル化するために、TASEP(Totally Asymmetric Simple Exclusion Process)[22]、PBN(確率論的論理ネットワーク)、ペトリネット、およびmax-plus代数などのさまざまなモデリング形式論が適用されてきた。「有用なモデルは単純で拡張可能である」という規範に従って[23]、前述した8つの基本的工程をすべて考慮したタンパク質合成の基本モデルが開発された[19]。最も単純なモデルM0は、反応速度論的な機構(図M0)で表される。これは一般化されて、40S、60S、開始因子(IF)結合を含むようになった(図M1')。さらに、マイクロRNAがタンパク質合成におよぼす影響を含めて拡張された[24]。この階層に含まれるほとんどのモデルは解析的に解くことができる。これらの解法を用いて、合成制御におけるさまざまな特異的な機構の「速度論的特徴」が得られた。
遺伝暗号
[編集]遺伝暗号を手作業で翻訳することも(短い配列の場合)または(後述のように適切にプログラミングした)コンピュータで翻訳することもできる。これにより、生物学者や化学者は、コード化タンパク質の化学構造を紙に描いて解釈することができる。
まず、下に示すように、鋳型DNAの各塩基をRNA相補体に変換する(アデニン(A)の相補体はウラシル(U)であることに注意すること)。なお、DNAの鋳型鎖はRNAが重合したものであり、もう一方のDNA鎖はRNAと同じであるが、ウラシルの代わりにチミン(T)を持つことに留意すること。
DNA -> RNA A -> U T -> A C -> G G -> C A=T-> A=U
次に、RNAをトリプレット(3塩基のグループ)に分割する。コードを読み始める場所によって、3つの翻訳ウィンドウ、すなわちリーディングフレームがあることに注意を要す。最後に、遺伝暗号の翻訳表を使用して、上記を化学で使われる構造式に変換する。
これによってタンパク質の一次構造(アミノ酸配列)が得られる。しかし、タンパク質は、鎖に沿った親水性セグメントと疎水性セグメントの部分に依存して折り畳まれる傾向がある。二次構造(部分的な立体構造)を推測できることは多いが、適切な三次構造(立体構造)を決定することは非常に困難である。タンパク質の三次構造やその他の側面は高度なアルゴリズムを用いて予測するしかないが、アミノ酸配列は核酸配列から翻訳表を使って単独で決定することができる。
この方法では、特に、セレノシステインのような型にあてはまらないアミノ酸がタンパク質に組み込まれる場合に、そのタンパク質の正しいアミノ酸組成を得られないことがある。セレノシステインは、型どおりの終止コドンと下流のヘアピン構造(SECIS)の組み合わせによってコード化されている。
DNA/RNA配列をタンパク質配列に翻訳することができるコンピュータプログラムは数多く存在する。通常、この処理は標準遺伝コード(Standard Genetic Code)を用いて行われるが、生物学的に重要な代替開始コドンの使用など、すべての「特別な」場合を扱えるプログラムはほとんどない。たとえば、まれな代替開始コドンであるCTGは、開始コドンとして使用された場合はメチオニンをコードし、その他の全ての位置ではロイシンをコードする。
例: 標準遺伝暗号に基づき要約された翻訳表(NCBIタクソノミーのウェブページより)[25]。
AAs = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG Starts = ---M---------------M---------------M---------------------------- Base1 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG Base2 = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG Base3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
「開始(Starts)」の行は、3つの開始コドン、UUG、CUG、そして非常に一般的なAUGを示す。また、開始位置として解釈した場合の最初のアミノ酸残基も示す。この場合、それらはすべてメチオニンである。
翻訳表
[編集]酵母ゲノムのような通常の真核生物の配列を扱う場合でも、ミトコンドリア遺伝子翻訳表など、別の翻訳表(translation table)を使用したい場合がよくある。2019年現在、GenBankの配列の翻訳では、NCBIタクソノミ-グループ(NCBI Taxonomy Group)によって以下の翻訳表が定義されている[25]。
- DNAコドン表
- 脊椎動物のミトコンドリアコード
- 酵母のミトコンドリアコード
- カビ、原生動物、腔腸動物のミトコンドリアコードとマイコプラズマ/スピロプラズマコード
- 無脊椎動物のミトコンドリアコード
- 繊毛虫、ダシクラダ科、ヘキサミタ属の核コード
- キネトプラストのコード
- 棘皮動物と扁形動物のミトコンドリアコード
- ユープロテス目の核コード
- 細菌、古細菌、植物の色素体コード
- 代替酵母の核コード
- ホヤ類のミトコンドリアコード
- 扁形動物の代替ミトコンドリアコード
- ブレファリスマ属の核コード
- 葉緑体のミトコンドリアコード
- トレマトード綱のミトコンドリアコード
- セネデスムス属のミトコンドリアコード
- スラウストキトリウム属のミトコンドリアコード
- 翼鰓類(いさいるい)のミトコンドリアコード
- 候補門SR1およびグラシリバクテリアのコード
- パチソレン・タノフィルスの核コード
- カリオレリクタの核コード
- コンジロストマの核コード
- メソジニウムの核コード
- ペリトリッチの核コード
- ブラストクリシジアの核コード
- エラフサカツギ科のミトコンドリアコード
関連項目
[編集]- 細胞 - 生物の基本的な構造・機能単位
- 細胞分裂 - 親細胞が2つの娘細胞に分裂する過程
- DNAとRNAのコドン表 - 遺伝暗号をアミノ酸配列に変換するのに用いる表
- エピジェネティクス - DNA塩基配列の変化を伴わない表現型の変化を研究する学問
- 拡張遺伝暗号 - 人為的に修正された遺伝暗号
- 遺伝子発現 - 遺伝子の情報がタンパク質などの生成を経て表現型に影響を及ぼす過程
- 遺伝子発現の調節 - 細胞が特定の遺伝子産物(タンパク質またはRNA)の産生を増加または減少させる機構
- 遺伝子 - 遺伝の基本単位
- ゲノム - 生物のすべての遺伝情報
- 生命 - シグナル伝達や自立プロセスなど生物学的プロセスを持つ物質
- タンパク質法 - タンパク質を研究するために用いられる技術
- 開始コドン - メッセンジャーRNA(mRNA)上の最初のコドン
脚注
[編集]- ^ a b Tirumalai MR, Rivas M, Tran Q, Fox GE (November 2021). “The Peptidyl Transferase Center: a Window to the Past”. Microbiol Mol Biol Rev 85 (4): e0010421. doi:10.1128/MMBR.00104-21. PMC 8579967. PMID 34756086.
- ^ Brooker RJ, Widmaier EP, Graham LE, Stiling PD (2014). Biology (Third international student ed.). New York, NY: McGraw Hill Education. pp. 249. ISBN 978-981-4581-85-1
- ^ Neill, Campbell (1996). Biology (Fourth ed.). The Benjamin/Cummings Publishing Company. pp. 309–310. ISBN 0-8053-1940-9
- ^ Stryer, Lubert (2002). Biochemistry (Fifth ed.). W. H. Freeman and Company. p. 826. ISBN 0-7167-4684-0
- ^ Moghal A, Mohler K, Ibba M (November 2014). “Mistranslation of the genetic code”. FEBS Letters 588 (23): 4305–10. doi:10.1016/j.febslet.2014.08.035. PMC 4254111. PMID 25220850.
- ^ Griffiths, Anthony (2008). “9”. Introduction to Genetic Analysis (9th ed.). New York: W.H. Freeman and Company. pp. 335–339. ISBN 978-0-7167-6887-6
- ^ “Computational Analysis of Genomic Sequences utilizing Machine Learning”. scholar.googleusercontent.com. 2022年1月12日閲覧。
- ^ Ross JF, Orlowski M (February 1982). “Growth-rate-dependent adjustment of ribosome function in chemostat-grown cells of the fungus Mucor racemosus”. Journal of Bacteriology 149 (2): 650–3. doi:10.1128/JB.149.2.650-653.1982. PMC 216554. PMID 6799491.
- ^ Ou X, Cao J, Cheng A, Peppelenbosch MP, Pan Q (March 2019). “Errors in translational decoding: tRNA wobbling or misincorporation?”. PLOS Genetics 15 (3): 2979–2986. doi:10.1371/journal.pgen.1008017. PMC 3158919. PMID 21930591.
- ^ Wohlgemuth I, Pohl C, Mittelstaet J, Konevega AL, Rodnina MV (October 2011). “Evolutionary optimization of speed and accuracy of decoding on the ribosome”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 366 (1580): 2979–86. doi:10.1098/rstb.2011.0138. PMC 6438450. PMID 30921315.
- ^ Sin C, Chiarugi D, Valleriani A (April 2016). “Quantitative assessment of ribosome drop-off in E. coli”. Nucleic Acids Research 44 (6): 2528–37. doi:10.1093/nar/gkw137. PMC 4824120. PMID 26935582.
- ^ Nakamoto T (February 2011). “Mechanisms of the initiation of protein synthesis: in reading frame binding of ribosomes to mRNA”. Molecular Biology Reports 38 (2): 847–55. doi:10.1007/s11033-010-0176-1. PMID 20467902.
- ^ Baggett NE, Zhang Y, Gross CA (March 2017). “Global analysis of translation termination in E. coli”. PLOS Genetics 13 (3): e1006676. doi:10.1371/journal.pgen.1006676. PMC 5373646. PMID 28301469.
- ^ Mora L, Zavialov A, Ehrenberg M, Buckingham RH (December 2003). “Stop codon recognition and interactions with peptide release factor RF3 of truncated and chimeric RF1 and RF2 from Escherichia coli”. Molecular Microbiology 50 (5): 1467–76. doi:10.1046/j.1365-2958.2003.03799.x. PMID 14651631.
- ^ Schueren F, Thoms S (August 2016). “Functional Translational Readthrough: A Systems Biology Perspective”. PLOS Genetics 12 (8): e1006196. doi:10.1371/JOURNAL.PGEN.1006196. PMC 4973966. PMID 27490485.
- ^ Cenik C, Cenik ES, Byeon GW, Grubert F, Candille SI, Spacek D, Alsallakh B, Tilgner H, Araya CL, Tang H, Ricci E, Snyder MP (November 2015). “Integrative analysis of RNA, translation, and protein levels reveals distinct regulatory variation across humans”. Genome Research 25 (11): 1610–21. doi:10.1101/gr.193342.115. PMC 4617958. PMID 26297486.
- ^ a b Xu Y, Ruggero D (March 2020). “The Role of Translation Control in Tumorigenesis and Its Therapeutic Implications”. Annual Review of Cancer Biology 4 (1): 437–457. doi:10.1146/annurev-cancerbio-030419-033420.
- ^ Truitt ML, Ruggero D (April 2016). “New frontiers in translational control of the cancer genome”. Nature Reviews. Cancer 16 (5): 288–304. doi:10.1038/nrc.2016.27. PMC 5491099. PMID 27112207.
- ^ a b c Gorban AN, Harel-Bellan A, Morozova N, Zinovyev A (July 2019). “Basic, simple and extendable kinetic model of protein synthesis”. Mathematical Biosciences and Engineering 16 (6): 6602–6622. doi:10.3934/mbe.2019329. PMID 31698578.
- ^ MacDonald CT, Gibbs JH, Pipkin AC (1968). “Kinetics of biopolymerization on nucleic acid templates”. Biopolymers 6 (1): 1–5. doi:10.1002/bip.1968.360060102. PMID 5641411.
- ^ Heinrich R, Rapoport TA (September 1980). “Mathematical modelling of translation of mRNA in eucaryotes; steady state, time-dependent processes and application to reticulocytes”. Journal of Theoretical Biology 86 (2): 279–313. Bibcode: 1980JThBi..86..279H. doi:10.1016/0022-5193(80)90008-9. PMID 7442295.
- ^ a b Skjøndal-Bar N, Morris DR (January 2007). “Dynamic model of the process of protein synthesis in eukaryotic cells”. Bulletin of Mathematical Biology 69 (1): 361–93. doi:10.1007/s11538-006-9128-2. PMID 17031456.
- ^ Coyte KZ, Tabuteau H, Gaffney EA, Foster KR, Durham WM (April 2017). “Reply to Baveye and Darnault: Useful models are simple and extendable”. Proceedings of the National Academy of Sciences of the United States of America 114 (14): E2804–E2805. Bibcode: 2017PNAS..114E2804C. doi:10.1073/pnas.1702303114. PMC 5389313. PMID 28341710.
- ^ Morozova N, Zinovyev A, Nonne N, Pritchard LL, Gorban AN, Harel-Bellan A (September 2012). “Kinetic signatures of microRNA modes of action”. RNA 18 (9): 1635–55. doi:10.1261/rna.032284.112. PMC 3425779. PMID 22850425.
- ^ a b “The Genetic Codes”. アメリカ国立生物工学情報センター (NCBI) (January 2019). 31 May 2022閲覧。
推薦文献
[編集]- ストライヤー生化学 (第8版). Jeremy Mark Berg, John L Tymoczko, Gregory Joseph Gatto, Tatsuro Irimura, Hiroto Okayama, Takao Shimizu. Tokyokagakudojin. (2018.8). ISBN 978-4-8079-0929-2. OCLC 1052771234
- Champe, Pamela C; Harvey, Richard A; Ferrier, Denise R (2004). Lippincott's Illustrated Reviews: Biochemistry (3rd ed.). Hagerstwon, MD: Lippincott Williams & Wilkins. ISBN 0-7817-2265-9
- Cox, Michael; Nelson, David R.; Lehninger, Albert L (2005). Lehninger principles of biochemistry (4th ed.). San Francisco...: W.H. Freeman. ISBN 0-7167-4339-6
- Malys N, McCarthy JE (March 2011). “Translation initiation: variations in the mechanism can be anticipated”. Cellular and Molecular Life Sciences 68 (6): 991–1003. doi:10.1007/s00018-010-0588-z. PMID 21076851.
外部リンク
[編集]ツール
[編集]- Translate - Expasy - DNAやRNA配列をタンパク質配列に翻訳する
解説動画
[編集]- 理化学研究所オミックス基盤研究領域「セントラルドグマ」2008年
- 理化学研究所ゲノム科学総合研究センター遺伝子構造・機能研究グループ「セントラルドグマ ~ゲノム情報からタンパク質ができるまで~」2007年
- 原核生物の翻訳開始機構「Animation of Protein Synthesis (Translation) in Prokaryotes.」
- 真核生物の翻訳開始機構「Eukaryotic Translation (Protein Synthesis), Animation.」
