リボヌクレアーゼH
| retroviral ribonuclease H | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 識別子 | |||||||||
| EC番号 | 3.1.26.13 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
リボヌクレアーゼH(英: ribonuclease H、略称: RNase H/RNH)は、配列非特異的エンドヌクレアーゼのファミリーであり、RNA:DNAハイブリッド基質中のRNAの加水分解による切断を触媒する。リボヌクレアーゼHファミリーのメンバーは、細菌や古細菌から真核生物までほぼすべての生物に存在する。
このファミリーはリボヌクレアーゼH1、H2と呼ばれる2つの進化的に関連したグループに分類され、両者は基質選択性がわずかに異なる[2]。ヒトゲノムにはH1とH2の双方がコードされている。ヒトのリボヌクレアーゼH2は3つのサブユニットからなるヘテロ三量体型複合体であり、いずれのサブユニットの変異もエカルディ・グティエール症候群(AGS)と呼ばれる希少疾患の遺伝的原因となる[3]。H1とH2は生命の全てのドメインに存在するのに対し、いくつかの原核生物にはH2と密接に関連した3番目のタイプが存在する[4]。さらに、HIVなどのレトロウイルスにコードされる多ドメイン型逆転写酵素にはリボヌクレアーゼH1に類似したリボヌクレアーゼHドメインが存在し、ウイルスの複製に必要である[5][6]。
真核生物では、リボヌクレアーゼH1はミトコンドリアDNAの複製に関与している。また、H1とH2の双方がRループのプロセシングなど、ゲノムの維持に関与している[2][7]。
分類と命名
[編集]リボヌクレアーゼHはエンドヌクレアーゼのファミリーの1つであり、RNA:DNA二本鎖のRNA鎖に対する特異性を共通して持つ。RNase Hはその定義として、RNAのホスホジエステル骨格を3'ヒドロキシル基と5'リン酸基を残して切断する[7]。RNase Hは、レトロウイルスのインテグラーゼ、DNAトランスポザーゼ、ホリデイジャンクション解離酵素、Piwiタンパク質やArgonauteタンパク質、さまざまなエキソヌクレアーゼ、スプライソソームタンパク質Prp8など、他のヌクレアーゼや核酸プロセシング酵素を含む進化的に関連したスーパーファミリーのメンバーであることが提唱されている[8][9]。
RNase HはH1とH2の2つのサブタイプへと大きく分類され、歴史的経緯により真核生物ではアラビア数字、原核生物ではローマ数字が用いられている。そのため、大腸菌のRNase HIはヒトのRNase H1のホモログである[2][7]。大腸菌やその他の多くの原核生物では、rnhA遺伝子がHIをコードし、rnhB遺伝子がHIIをコードする。いくつかの細菌や古細菌にはHIIIと呼ばれる3番目のクラスが存在し、これらは原核生物のHII酵素と密接に関連している[4]。
構造
[編集]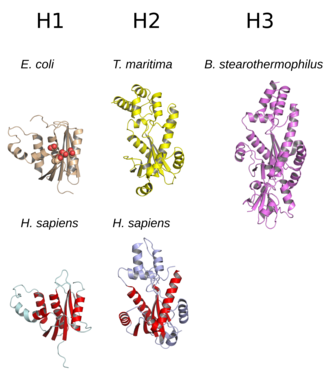
RNase Hは一般的に、5本のストランドからなるβシートがαヘリックスに囲まれた構造をしている[10]。すべてのRNase Hには、アスパラギン酸とグルタミン酸残基からなる保存された配列モチーフ(DEDDモチーフと呼ばれることが多い)を中心とした活性部位が存在する。これらの残基は触媒に必要なマグネシウムイオンと相互作用する[5][7]。
RNase H2はH1よりも大きく、より多くのヘリックスが存在することが多い。各酵素のドメイン構成はさまざまであり、H1グループに属する原核生物型酵素の一部と真核生物型酵素の大部分には、N末端にハイブリッド結合ドメイン(hybrid binding domain)と呼ばれる小さなドメインが存在する。このドメインはRNA:DNAハイブリッド二本鎖への結合を促進し、プロセシビティを高めることがある[2][7][11]。H1グループの全てのメンバーとH2グループの原核生物のメンバーは単量体として機能する一方で、真核生物のH2酵素は必ずヘテロ三量体として機能する[2][7]。原核生物のHIII酵素は広義のH2グループのメンバーであり、構造的特徴の大部分はH2と共通しており、さらにN末端にTATAボックス結合ドメインが存在する[7]。レトロウイルスの多ドメイン型逆転写酵素中に存在するRNase Hドメインの構造は、H1グループときわめて類似している[5]。
RNase H1は構造と酵素活性の関係の研究に広く利用されている。特に大腸菌のホモログは、タンパク質のフォールディング研究のためのモデルシステムとしても利用されている[12][13][14]。H1グループ内では、より大きくより塩基性の高い基質結合表面を形成する、ヘリックスと柔軟なループ構造からなる構造エレメントの存在と、基質結合親和性の高さとの関係が明らかにされている。Cヘリックスと呼ばれるヘリックスは分類学的に散在して分布しており、大腸菌とヒトのRNase H1には存在するが、HIVのRNase Hドメインには存在しない。一方でCヘリックスを持つレトロウイルスのドメインも存在する[15][16]。
機能
[編集]リボヌクレアーゼHはRNA:DNAハイブリッド二本鎖中のRNAのホスホジエステル結合を切断し、切断部位の両側には3'ヒドロキシル基と5'リン酸基が形成される。切断は2金属イオン機構(two-metal-ion mechanism)によって触媒され、触媒機能にはMg2+やMn2+などの2価カチオンが直接関与する[17]。RNase Hはアミノ酸配列の違いによって1型と2型に分類される[7][18]。1型のRNase Hは、原核生物と真核生物のRNase H1、そしてレトロウイルスのRNase Hである。2型のRNase Hは、原核生物と真核生物のRNase H2、そして細菌のRNase HIIIである。RNase Hは単量体として存在するが、真核生物のRNase H2は例外的にヘテロ三量体型として存在する[19][20]。RNase H1とH2は異なる基質選択性を持ち、細胞内で異なるものの重複した機能を持つ。原核生物や下等真核生物ではどちらのタイプの酵素も必須ではないが、高等真核生物では双方が必須であると考えられている[2]。こうした酵素はRループのRNA要素を分解するため、H1とH2の双方の活性がゲノム安定性の維持と関係している[21][22]。
リボヌクレアーゼH1
[編集]| 識別子 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 略号 | RNase H | ||||||||
| Pfam | PF00075 | ||||||||
| Pfam clan | CL0219 | ||||||||
| InterPro | IPR002156 | ||||||||
| PROSITE | PS50879 | ||||||||
| |||||||||
リボヌクレアーゼH1は基質中に少なくとも4つのリボヌクレオチドを含む塩基対を必要とする。そのためリボヌクレオチドを1つだけ持ち、それ以外がデオキシリボヌクレオチドで構成されているような鎖からリボヌクレオチドを除去することはできない。こうした理由により、RNase H1はDNA複製時に岡崎フラグメントのRNAプライマーのプロセシングに関与している可能性は低いと考えられている[2]。RNase H1はこれまで研究が行われている単細胞生物では必須ではないが、大腸菌ではRNase H1のノックアウトによって温度感受性の表現型が生じ[7]、出芽酵母ではストレス応答に欠陥が生じる[23]。
哺乳類を含む多くの真核生物では、RNase H1の遺伝子にはミトコンドリア標的化配列(MTS)が含まれ、MTSを含むアイソフォームと含まないアイソフォームが発現する。その結果、RNase H1はミトコンドリアと核の双方に局在する。ノックアウトマウスモデルでは、RNase H1ヌル変異体はミトコンドリアDNAの複製の欠陥のために胚発生時に致死となる[2][24][25]。RNase H1の喪失によって誘導されるミトコンドリアDNA複製の欠陥は、おそらくRループのプロセシングの欠陥によるものである[22]。
リボヌクレアーゼH2
[編集]| 識別子 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 略号 | RNase HII | ||||||||
| Pfam | PF01351 | ||||||||
| Pfam clan | CL0219 | ||||||||
| InterPro | IPR024567 | ||||||||
| |||||||||
原核生物では、RNase HIIは単量体タンパク質として酵素活性を持つ。真核生物では常にヘテロ三量体を形成しており、触媒サブユニットAと構造サブユニットB、Cから構成される。Aサブユニットは原核生物のRNase HIIと極めて高い相同性を有するものの、B、Cサブユニットに関しては原核生物に明確なホモログは存在せず、真核生物の間でさえも配列レベルでの保存性は低い[26][27]。BサブユニットはH2複合体とPCNAとの間のタンパク質間相互作用を媒介し、replication foci(複製が行われている領域)にH2を局在させる[28]。
原核生物と真核生物のH2はどちらも鎖中に存在するリボヌクレオチドを切断することができる[2]。しかしながら、切断パターンと基質選択性はわずかに異なる。原核生物の酵素はプロセシビティが低く、5'側がデオキシリボヌクレオチドとなっているリボヌクレオチドよりも、リボヌクレオチドが連続しているものをより効率的に加水分解する。一方、真核生物の酵素はプロセシビティが高く、どちらのタイプの基質も同等の効率で加水分解する[2][27]。RNase H2はその基質特異性のため、Rループのプロセシングに加えて、誤ってDNAに取り込まれたリボヌクレオチドを除去するリボヌクレオチド除去修復機構に関与している[28][29][30]。哺乳類の細胞核にはH1とH2の双方が存在するが、RNase H活性としてはH2が優勢であり、ゲノム安定性の維持に重要である[28]。
一部の原核生物には、その他にRNase HIIIと呼ばれるH2型遺伝子が存在する。HIIIタンパク質は配列同一性や構造類似性の観点からはH2グループとより密接に関連しているが、基質選択性の観点からはH1とより密接に関連している[7][31]。HIとHIIはどちらも原核生物の間で広く分布しているのに対し、HIIIは分類学上散在して分布するわずかな種でのみ見つかっている。どちらかといえば古細菌でより多く見られ、またHIを持つ原核生物にHIIIが存在することはほとんど、または決してない[32]。
機構
[編集]
ほぼすべてのRNase Hの活性部位には4つの負に帯電したアミノ酸残基が存在し、DEDDモチーフと呼ばれている。HIV-1、ヒト、大腸菌のRNase Hなどのように、ヒスチジン残基も存在することが多い[2][7]。
荷電残基は、触媒に必要な2つの金属イオンを結合する。生理的条件下では結合しているのはマグネシウムイオンであるが、多くの場合マンガンイオンでも酵素活性は維持される[2][7]。一方、カルシウムイオンや高濃度のマグネシウムイオンは活性を阻害する[11][33][34]。
実験的証拠と計算機シミュレーションによると、金属イオンの1つに結合した水分子を保存されたヒスチジンが活性化する[33][35]。遷移状態は会合体としての性質を示し[17]、プロトン化されたリン酸と脱プロトン化されたアルコキシド脱離基からなる中間体が形成される[35]。脱離基は、pKaが高くプロトン化されている可能性が高いグルタミン酸を介してプロトン化される。この機構はRNase TやCas9のRuvCサブユニットと類似しており、どちらもヒスチジンと2金属イオン機構が利用される。
切断産物の放出機構は未解明である。時間分解結晶構造解析や類似したヌクレアーゼから得られた実験的証拠からは、活性部位にリクルートされた第3のイオンの役割が指摘されている[36][37]。
ヒト
[編集]ヒトゲノムには、RNase Hをコードする4つの遺伝子が存在する。
- RNASEH1 - H1(単量体型)サブタイプ
- RNASEH2A - 三量体型H2複合体触媒サブユニット
- RNASEH2B - 三量体型H2複合体構造サブユニット
- RNASEH2C - 三量体型H2複合体構造サブユニット
ゲノム中にはレトロウイルス起源の遺伝物質が高頻度でみられる。これらはヒト内在性レトロウイルスのゲノムへの組み込みを反映したものである。こうした組み込みはレトロウイルス型逆転写酵素をコードする遺伝子の存在によるものであり、こうした酵素にはRNase Hドメインが含まれている。その一例はERVK6である[38]。LTR型・非LTR型レトロトランスポゾンもゲノム中に広くみられ、これにもRNase Hドメインが含まれることが多く、複雑な進化的歴史が見て取れる[39][40][41]。
疾患における役割
[編集]
小規模な研究では、ヒトのRNase H1の変異はミトコンドリア病の一般的症状である慢性進行性外眼筋麻痺症候群と関係していることが示されている[25]。
RNase H2の3つのサブユニットのいずれの変異もエカルディ・グティエール症候群(AGS)と呼ばれる希少遺伝疾患の原因となることが明確に示されている[3]。この疾患では幼年期に神経・皮膚症状がみられる[43]。AGSの症状は先天性ウイルス感染症の症状ときわめて類似しており、I型インターフェロンの不適切なアップレギュレーションと関係している。AGSはTREX1、SAMHD1、ADAR、MDA5/IFIH1といった他の遺伝子の変異によっても引き起こされ、これらの遺伝子は全て核酸のプロセシングに関与している[44]。AGS患者集団での変異の分布の解析からは、すべてのAGS変異の5%がRNASEH2A、36%が2B、12%が2Cに位置することが明らかにされている[45]。2Bの変異でみられる神経学的異常はいくぶん軽度であり[46]、他のAGS関連遺伝子型の患者でみられるような、インターフェロンによる遺伝子のアップレギュレーションはみられない[44]。
ウイルス
[編集]
2つのグループのウイルスがその生活環の一部として逆転写を利用する。1つはレトロウイルスであり、一本鎖RNAゲノムを持ち、二本鎖DNA中間体を介して複製を行う。もう1つは二本鎖DNA逆転写(dsDNA-RT)ウイルスで、RNAプレゲノム中間体を介して二本鎖DNAゲノムの複製を行う。それぞれ病原体としては、ヒト免疫不全ウイルス(HIV)とB型肝炎ウイルス(HBV)が挙げられる。どちらも巨大な多機能型逆転写酵素(RT)をコードし、RNase Hドメインが含まれる[48][49]。
HIV-1やマウス白血病ウイルスのRTはこのファミリーで最もよく研究が行われている[50][51]。レトロウイルスのRTはウイルスの一本鎖RNAゲノムを二本鎖DNAへ変換する役割を果たす。この過程は3つの段階からなる。まず、RNA依存性DNAポリメラーゼ活性によって(+)鎖RNA鋳型から(-)鎖のDNAが生成され、RNA:DNAハイブリッド中間体が形成される。次に、RNA鎖が破壊される。そして、DNA依存性DNAポリメラーゼ活性によって(+)鎖DNAが合成され、最終産物である二本鎖DNAが形成される。2番目の段階はRTのC末端に位置するRNase Hドメインによって行われる[5][6][52][53]。
RNase Hは、(+)鎖RNAゲノムの非特異的分解、(-)鎖tRNAプライマーの特異的除去、(+)鎖ポリプリントラクト(PPT)プライマーの除去という3種類の作用を行う[54]。RNase Hは(+)鎖のプライミングに関与しているが、新たなプライマー配列の合成といった一般的手法をとるのではなく、RNase Hによる切断に対して抵抗性を持つPPTからプライマーを形成する。PPT以外の全ての塩基が除去されることで、PPTがLTRのU3領域の末端のマーカーとして利用されるようになる[53]。
RNase H活性はウイルスの増殖に必要であるため、このドメインはHIV/AIDSやレトロウイルスによる他の疾患の治療のための抗ウイルス薬開発の標的となると考えられている。レトロウイルスのRNase Hの阻害剤としていくつかの異なる化学種が同定されているが、その多くの作用機序は活性部位のカチオンに対するキレート作用に基づいている[55]。RTのポリメラーゼ機能を特異的に阻害する逆転写酵素阻害薬は臨床で広く用いられているが、RNase H機能の阻害薬は用いられていない。RNase HはHIVにコードされている酵素機能のうち、臨床使用されている薬剤の標的となっていない唯一の機能である[52][56]。
進化
[編集]RNase Hは生物に幅広く分布しており、生命の全てのドメインに存在する。このファミリーはヌクレアーゼスーパーファミリーに属し、これらが進化的祖先であると考えられている[57]。原核生物のゲノムには複数のRNase H遺伝子が存在することが多いが、HI、HII、HIII遺伝子の存在と全体的な系統学的関係の間にはほとんど相関はみられず、これらの酵素の分布には遺伝子の水平伝播が寄与していることが示唆される。RNase HIとHIIIが同じ原核生物のゲノムに存在することは、ほとんどもしくは全くない。また、ある生物のゲノムに複数のRNase H遺伝子が存在する場合、それらには活性レベルで大きな差異が存在することがある。こうした現象は、RNase H遺伝子間での機能的冗長性を最小化する進化パターンを反映したものであることが示唆されている[7][32]。RNase HIIIは原核生物固有のものであるが、その分布は系統学的に散在しており、細菌と古細菌の双方に存在する[32]。これらはきわめて初期にHIIから分岐したものであると考えられている[58]。
真核生物におけるRNase H2の進化的軌跡、特に真核生物ホモログが三量体を形成するようになった機構は明らかではない。B、Cサブユニットは原核生物に明確なホモログが存在しない[2][27]。
応用
[編集]RNase HはRNA:DNAハイブリッド二本鎖中のRNAのみを特異的に分解するため、分子生物学における実験試薬として広く利用されている。大腸菌のRNase HIとHIIの精製試料は市販されている。RNase HIは、逆転写によるcDNA合成後のRNA鋳型の分解に利用されることが多い。また、短い相補性DNA断片の存在下でRNA配列特異的な分解を行うためにも利用される[59]。切断の検出には表面プラズモン共鳴などの高感度技術が用いられる[60][61]。RNase HIIは岡崎フラグメントのRNAプライマー部分の分解やリボヌクレオチドを含む部位への一本鎖ニックの導入に利用される[59]。ホットスタートPCRの一変種であるRNase H依存型PCR(rhPCR)は、超好熱性古細菌Pyrococcus abyssi由来の熱安定性RNase HIIを利用する[62]。一般的に試薬として利用されるリボヌクレアーゼインヒビターは、HIやHIIの活性に対する阻害効果はないことに注意が必要である[59]。
歴史
[編集]リボヌクレアーゼHはPeter Hausenの研究室で最初に発見された。1969年に子牛胸腺でRNA:DNAハイブリッドに対するエンドヌクレアーゼ活性が発見され、ハイブリッド(hybrid)に対する特異性から「リボヌクレアーゼH」と命名された[26][63][64]。その後、RNase H活性は大腸菌でも発見され[65]、またウイルスの逆転写に関する初期の研究の過程でRNAゲノムを持つ腫瘍ウイルス試料中にも発見された[66][67]。後に、子牛胸腺抽出物にはRNase H活性を持つ複数のタンパク質が存在すること[68]、そして大腸菌には2つのRNase Hが存在することが明らかとなった[69][70]。もともと、真核生物で現在RNase H2として知られている酵素はH1、H1はH2と命名されていたが、比較解析を容易にするため大腸菌と一致するように名称が交換され、原核生物型酵素はローマ数字、真核生物型酵素はアラビア数字で表す現在の命名法が確立された[2][26][31][71]。原核生物のRNase HIIIは1999年に報告された最も新しいサブタイプである[31]。
真核生物のRNase H2の特性解析は歴史的に困難なものであったが、その一因はその存在量の少なさである[2]。酵素の精製に関する注意深い努力により、真核生物型酵素は大腸菌のRNase HIIとは異なり、複数のサブユニットから構成されることが示唆された[72]。大腸菌タンパク質の出芽酵母ホモログ(すなわちH2Aサブユニット)は、酵母ゲノムの配列決定がなされた際にバイオインフォマティクスによって容易に同定されたが[73]、対応するタンパク質は単独では酵素活性を示さなかった[2][23]。最終的に、酵母のB、Cサブユニットが共精製によって単離され、酵素活性に必要であることが発見された[74]。しかしながら、酵母のB、Cサブユニットと他の生物のホモログとの間の配列同一性は極めて低く、対応するヒトタンパク質が最終的に決定されたのは、これら3つのサブユニットの変異すべてがエカルディ・グティエール症候群の原因となることが判明してからであった[2][3]。
出典
[編集]- ^ PDB: 1JL1; “Native-state energetics of a thermostabilized variant of ribonuclease HI”. Journal of Molecular Biology 314 (4): 863–71. (December 2001). doi:10.1006/jmbi.2001.5184. PMID 11734003.
- ^ a b c d e f g h i j k l m n o p q “Ribonuclease H: the enzymes in eukaryotes”. The FEBS Journal 276 (6): 1494–505. (March 2009). doi:10.1111/j.1742-4658.2009.06908.x. PMC 2746905. PMID 19228196.
- ^ a b c Crow, Yanick J.; Leitch, Andrea; Hayward, Bruce E.; Garner, Anna; Parmar, Rekha; Griffith, Elen; Ali, Manir; Semple, Colin et al. (2006-08). “Mutations in genes encoding ribonuclease H2 subunits cause Aicardi-Goutières syndrome and mimic congenital viral brain infection”. Nature Genetics 38 (8): 910–916. doi:10.1038/ng1842. ISSN 1061-4036. PMID 16845400.
- ^ a b “Crystal structure of RNase H3-substrate complex reveals parallel evolution of RNA/DNA hybrid recognition”. Nucleic Acids Research 42 (14): 9285–94. (August 2014). doi:10.1093/nar/gku615. PMC 4132731. PMID 25016521.
- ^ a b c d “Crystal structure of the ribonuclease H domain of HIV-1 reverse transcriptase”. Science 252 (5002): 88–95. (April 1991). Bibcode: 1991Sci...252...88D. doi:10.1126/science.1707186. PMID 1707186.
- ^ a b “Identification and characterization of HIV-specific RNase H by monoclonal antibody”. The EMBO Journal 7 (1): 239–43. (January 1988). doi:10.1002/j.1460-2075.1988.tb02805.x. PMC 454263. PMID 2452083.
- ^ a b c d e f g h i j k l m “Ribonuclease H: molecular diversities, substrate binding domains, and catalytic mechanism of the prokaryotic enzymes”. The FEBS Journal 276 (6): 1482–93. (March 2009). doi:10.1111/j.1742-4658.2009.06907.x. PMID 19228197.
- ^ “The RNase H-like superfamily: new members, comparative structural analysis and evolutionary classification”. Nucleic Acids Research 42 (7): 4160–79. (April 2014). doi:10.1093/nar/gkt1414. PMC 3985635. PMID 24464998.
- ^ “Retroviral integrases and their cousins”. Current Opinion in Structural Biology 6 (1): 76–83. (February 1996). doi:10.1016/s0959-440x(96)80098-4. PMID 8696976.
- ^ “Thermal and mechanical multistate folding of ribonuclease H”. The Journal of Chemical Physics 131 (23): 235101. (December 2009). Bibcode: 2009JChPh.131w5101S. doi:10.1063/1.3270167. PMID 20025349.
- ^ a b “Specific recognition of RNA/DNA hybrid and enhancement of human RNase H1 activity by HBD”. The EMBO Journal 27 (7): 1172–81. (April 2008). doi:10.1038/emboj.2008.44. PMC 2323259. PMID 18337749.
- ^ “Direct observation of the three-state folding of a single protein molecule”. Science 309 (5743): 2057–60. (September 2005). Bibcode: 2005Sci...309.2057C. doi:10.1126/science.1116702. PMID 16179479.
- ^ “A thermodynamic comparison of mesophilic and thermophilic ribonucleases H”. Biochemistry 38 (12): 3831–6. (March 1999). doi:10.1021/bi982684h. PMID 10090773.
- ^ “The kinetic folding intermediate of ribonuclease H resembles the acid molten globule and partially unfolded molecules detected under native conditions”. Nature Structural Biology 4 (4): 298–304. (April 1997). doi:10.1038/nsb0497-298. PMID 9095198.
- ^ “RNase H activity: structure, specificity, and function in reverse transcription”. Virus Research 134 (1–2): 86–103. (June 2008). doi:10.1016/j.virusres.2007.12.007. PMC 2464458. PMID 18261820.
- ^ “Ribonuclease H: properties, substrate specificity and roles in retroviral reverse transcription”. The FEBS Journal 276 (6): 1506–16. (March 2009). doi:10.1111/j.1742-4658.2009.06909.x. PMC 2742777. PMID 19228195.
- ^ a b “Making and breaking nucleic acids: two-Mg2+-ion catalysis and substrate specificity”. Molecular Cell 22 (1): 5–13. (April 2006). doi:10.1016/j.molcel.2006.03.013. PMID 16600865.
- ^ “Molecular diversities of RNases H”. Journal of Bioscience and Bioengineering 88 (1): 12–9. (January 1999). doi:10.1016/s1389-1723(99)80168-6. PMID 16232566.
- ^ “PCNA directs type 2 RNase H activity on DNA replication and repair substrates”. Nucleic Acids Research 39 (9): 3652–66. (May 2011). doi:10.1093/nar/gkq980. PMC 3089482. PMID 21245041.
- ^ “The structural and biochemical characterization of human RNase H2 complex reveals the molecular basis for substrate recognition and Aicardi-Goutières syndrome defects”. The Journal of Biological Chemistry 286 (12): 10540–50. (March 2011). doi:10.1074/jbc.M110.181974. PMC 3060507. PMID 21177858.
- ^ “RNase H enables efficient repair of R-loop induced DNA damage”. eLife 5: e20533. (December 2016). doi:10.7554/eLife.20533. PMC 5215079. PMID 27938663.
- ^ a b “Viable RNaseH1 knockout mice show RNaseH1 is essential for R loop processing, mitochondrial and liver function”. Nucleic Acids Research 44 (11): 5299–312. (June 2016). doi:10.1093/nar/gkw350. PMC 4914116. PMID 27131367.
- ^ a b “The absence of ribonuclease H1 or H2 alters the sensitivity of Saccharomyces cerevisiae to hydroxyurea, caffeine and ethyl methanesulphonate: implications for roles of RNases H in DNA replication and repair”. Genes to Cells 5 (10): 789–802. (October 2000). doi:10.1046/j.1365-2443.2000.00373.x. PMID 11029655.
- ^ “Failure to produce mitochondrial DNA results in embryonic lethality in Rnaseh1 null mice”. Molecular Cell 11 (3): 807–15. (March 2003). doi:10.1016/s1097-2765(03)00088-1. PMID 12667461.
- ^ a b “RNASEH1 Mutations Impair mtDNA Replication and Cause Adult-Onset Mitochondrial Encephalomyopathy”. American Journal of Human Genetics 97 (1): 186–93. (July 2015). doi:10.1016/j.ajhg.2015.05.013. PMC 4572567. PMID 26094573.
- ^ a b c Hollis, Thomas; Shaban, Nadine M. (2011-01-01). Nicholson, Allen W.. ed. Ribonucleases. Nucleic Acids and Molecular Biology. Springer Berlin Heidelberg. pp. 299–317. doi:10.1007/978-3-642-21078-5_12. ISBN 978-3-642-21077-8
- ^ a b c “Contributions of the two accessory subunits, RNASEH2B and RNASEH2C, to the activity and properties of the human RNase H2 complex”. Nucleic Acids Research 37 (1): 96–110. (January 2009). doi:10.1093/nar/gkn913. PMC 2615623. PMID 19015152.
- ^ a b c “Ribonuclease H2 in health and disease”. Biochemical Society Transactions 42 (4): 717–25. (August 2014). doi:10.1042/BST20140079. PMID 25109948.
- ^ “RNase H and multiple RNA biogenesis factors cooperate to prevent RNA:DNA hybrids from generating genome instability”. Molecular Cell 44 (6): 978–88. (December 2011). doi:10.1016/j.molcel.2011.10.017. PMC 3271842. PMID 22195970.
- ^ “Mutagenic processing of ribonucleotides in DNA by yeast topoisomerase I”. Science 332 (6037): 1561–4. (June 2011). Bibcode: 2011Sci...332.1561K. doi:10.1126/science.1205016. PMC 3380281. PMID 21700875.
- ^ a b c “Identification of the genes encoding Mn2+-dependent RNase HII and Mg2+-dependent RNase HIII from Bacillus subtilis: classification of RNases H into three families”. Biochemistry 38 (2): 605–18. (January 1999). doi:10.1021/bi982207z. PMID 9888800.
- ^ a b c “Evolution of ribonuclease H genes in prokaryotes to avoid inheritance of redundant genes”. BMC Evolutionary Biology 7: 128. (July 2007). doi:10.1186/1471-2148-7-128. PMC 1950709. PMID 17663799.
- ^ a b “Evidence for a dual functional role of a conserved histidine in RNA·DNA heteroduplex cleavage by human RNase H1”. FEBS Journal 279 (24): 4492–500. (December 2012). doi:10.1111/febs.12035. PMC 3515698. PMID 23078533.
- ^ “Calcium inhibition of ribonuclease H1 two-metal ion catalysis”. Journal of the American Chemical Society 136 (8): 3137–44. (February 2014). doi:10.1021/ja411408x. PMC 3985467. PMID 24499076.
- ^ a b “The Role of Conserved Residues in the DEDDh Motif: the Proton-Transfer Mechanism of HIV-1 RNase H”. ACS Catalysis 11 (13): 7915–7927. (16 June 2021). doi:10.1021/acscatal.1c01493.
- ^ “A stepwise model for double-stranded RNA processing by ribonuclease III”. Mol Microbiol 67 (1): 143–54. (January 2008). doi:10.1111/j.1365-2958.2007.06032.x. PMID 18047582.
- ^ “Cation trafficking propels RNA hydrolysis”. Nature Structural & Molecular Biology 25 (8): 715–721. (August 2019). doi:10.1038/s41594-018-0099-4. PMC 6110950. PMID 30076410.
- ^ “Genomic organization of the human endogenous retrovirus HERV-K(HML-2.HOM) (ERVK6) on chromosome 7”. Genomics 72 (3): 314–20. (March 2001). doi:10.1006/geno.2000.6488. PMID 11401447.
- ^ “Convergence of retrotransposons in oomycetes and plants”. Mobile DNA 8 (1): 4. (14 March 2017). doi:10.1186/s13100-017-0087-y. PMC 5348765. PMID 28293305.
- ^ “Convergent evolution of ribonuclease h in LTR retrotransposons and retroviruses”. Molecular Biology and Evolution 32 (5): 1197–207. (May 2015). doi:10.1093/molbev/msv008. PMC 4408406. PMID 25605791.
- ^ “Ribonuclease H evolution in retrotransposable elements”. Cytogenetic and Genome Research 110 (1–4): 392–401. (2005). doi:10.1159/000084971. PMID 16093691.
- ^ “The structural and biochemical characterization of human RNase H2 complex reveals the molecular basis for substrate recognition and Aicardi-Goutières syndrome defects”. The Journal of Biological Chemistry 286 (12): 10540–50. (March 2011). doi:10.1074/jbc.M110.181974. PMC 3060507. PMID 21177858.
- ^ “Aicardi-Goutieres syndrome”. British Medical Bulletin 89: 183–201. (2009). doi:10.1093/bmb/ldn049. PMID 19129251.
- ^ a b “Aicardi-Goutières syndrome and the type I interferonopathies”. Nature Reviews. Immunology 15 (7): 429–40. (July 2015). doi:10.1038/nri3850. PMID 26052098.
- ^ Crow, Yanick J.; Chase, Diana S.; Lowenstein Schmidt, Johanna; Szynkiewicz, Marcin; Forte, Gabriella M. A.; Gornall, Hannah L.; Oojageer, Anthony; Anderson, Beverley et al. (2015-02). “Characterization of human disease phenotypes associated with mutations in TREX1, RNASEH2A, RNASEH2B, RNASEH2C, SAMHD1, ADAR, and IFIH1”. American Journal of Medical Genetics. Part A 167A (2): 296–312. doi:10.1002/ajmg.a.36887. ISSN 1552-4833. PMC 4382202. PMID 25604658.
- ^ Rice, Gillian; Patrick, Teresa; Parmar, Rekha; Taylor, Claire F.; Aeby, Alec; Aicardi, Jean; Artuch, Rafael; Montalto, Simon Attard et al. (2007-10). “Clinical and molecular phenotype of Aicardi-Goutieres syndrome”. American Journal of Human Genetics 81 (4): 713–725. doi:10.1086/521373. ISSN 0002-9297. PMC 2227922. PMID 17846997.
- ^ “Crystal structure of HIV-1 reverse transcriptase in complex with a polypurine tract RNA:DNA”. The EMBO Journal 20 (6): 1449–61. (March 2001). doi:10.1093/emboj/20.6.1449. PMC 145536. PMID 11250910.
- ^ “Molecular biology of hepatitis B virus infection”. Virology 479-480: 672–86. (May 2015). doi:10.1016/j.virol.2015.02.031. PMC 4424072. PMID 25759099.
- ^ Vicenzi, Elisa, ed (2014-01-01). “RNase H: specificity, mechanisms of action, and antiviral target”. Human Retroviruses. Methods in Molecular Biology. 1087. Humana Press. pp. 71–84. doi:10.1007/978-1-62703-670-2_7. ISBN 978-1-62703-669-6. PMID 24158815
- ^ “Insight into the mechanism of the stabilization of moloney murine leukaemia virus reverse transcriptase by eliminating RNase H activity”. Bioscience, Biotechnology, and Biochemistry 74 (2): 440–2. (February 2010). doi:10.1271/bbb.90777. PMID 20139597.
- ^ “Murine leukemia virus reverse transcriptase: structural comparison with HIV-1 reverse transcriptase”. Virus Research 134 (1–2): 186–202. (June 2008). doi:10.1016/j.virusres.2008.01.001. PMC 2443788. PMID 18294720.
- ^ a b Nowotny, Marcin; Figiel, Małgorzata (2013-01-01). LeGrice, Stuart. ed. Human Immunodeficiency Virus Reverse Transcriptase. Springer New York. pp. 53–75. doi:10.1007/978-1-4614-7291-9_3. ISBN 978-1-4614-7290-2
- ^ a b “HIV-1 Ribonuclease H: Structure, Catalytic Mechanism and Inhibitors”. Viruses 2 (4): 900–26. (April 2010). doi:10.3390/v2040900. PMC 3185654. PMID 21994660.
- ^ “Uncovering the complexities of retroviral ribonuclease H reveals its potential as a therapeutic target”. AIDS Reviews 4 (4): 183–94. (2002). PMID 12555693.
- ^ “HIV-1 RT-associated RNase H function inhibitors: Recent advances in drug development”. Current Medicinal Chemistry 17 (26): 2837–53. (2010). doi:10.2174/092986710792065045. PMID 20858167.
- ^ “Recent progress in the research of small molecule HIV-1 RNase H inhibitors”. Current Medicinal Chemistry 21 (17): 1956–67. (June 2014). doi:10.2174/0929867321666140120121158. PMID 24438523.
- ^ “Characters of very ancient proteins”. Biochemical and Biophysical Research Communications 366 (3): 607–11. (February 2008). doi:10.1016/j.bbrc.2007.12.014. PMID 18073136.
- ^ “Evolutionary history of the TBP-domain superfamily”. Nucleic Acids Research 41 (5): 2832–45. (March 2013). doi:10.1093/nar/gkt045. PMC 3597702. PMID 23376926.
- ^ a b c Nichols, Nicole M.; Yue, Dongxian (2001-01-01). Ribonucleases. Chapter 3. John Wiley & Sons, Inc.. Unit3.13. doi:10.1002/0471142727.mb0313s84. ISBN 978-0-471-14272-0. PMID 18972385
- ^ “A non-PCR SPR platform using RNase H to detect MicroRNA 29a-3p from throat swabs of human subjects with influenza A virus H1N1 infection”. The Analyst 140 (13): 4566–75. (July 2015). Bibcode: 2015Ana...140.4566L. doi:10.1039/C5AN00679A. PMID 26000345.
- ^ “Direct detection of genomic DNA by enzymatically amplified SPR imaging measurements of RNA microarrays”. Journal of the American Chemical Society 126 (13): 4086–7. (April 2004). doi:10.1021/ja039823p. PMID 15053580.
- ^ “RNase H-dependent PCR (rhPCR): improved specificity and single nucleotide polymorphism detection using blocked cleavable primers”. BMC Biotechnology 11: 80. (August 2011). doi:10.1186/1472-6750-11-80. PMC 3224242. PMID 21831278.
- ^ “Enzyme from calf thymus degrading the RNA moiety of DNA-RNA Hybrids: effect on DNA-dependent RNA polymerase”. Science 166 (3903): 393–5. (October 1969). Bibcode: 1969Sci...166..393S. doi:10.1126/science.166.3903.393. PMID 5812039.
- ^ “Ribonuclease H. An enzyme degrading the RNA moiety of DNA-RNA hybrids”. European Journal of Biochemistry 14 (2): 278–83. (June 1970). doi:10.1111/j.1432-1033.1970.tb00287.x. PMID 5506170.
- ^ “Ribonuclease H (hybrid) in Escherichia coli. Identification and characterization”. The Journal of Biological Chemistry 248 (7): 2621–4. (April 1973). doi:10.1016/S0021-9258(19)44152-5. PMID 4572736.
- ^ “Association of viral reverse transcriptase with an enzyme degrading the RNA moiety of RNA-DNA hybrids”. Nature 234 (51): 240–3. (December 1971). doi:10.1038/newbio234240a0. PMID 4331605.
- ^ “Ribonuclease H: a ubiquitous activity in virions of ribonucleic acid tumor viruses”. Journal of Virology 10 (6): 1136–42. (December 1972). doi:10.1128/jvi.10.6.1136-1142.1972. PMC 356594. PMID 4118867.
- ^ “Distinct ribonuclease H activities in calf thymus”. European Journal of Biochemistry 52 (1): 179–90. (March 1975). doi:10.1111/j.1432-1033.1975.tb03985.x. PMID 51794.
- ^ “DNA sequence of the gene coding for Escherichia coli ribonuclease H”. The Journal of Biological Chemistry 258 (2): 1276–81. (January 1983). doi:10.1016/S0021-9258(18)33189-2. PMID 6296074.
- ^ “Isolation and characterization of a second RNase H (RNase HII) of Escherichia coli K-12 encoded by the rnhB gene”. Proceedings of the National Academy of Sciences of the United States of America 87 (21): 8587–91. (November 1990). Bibcode: 1990PNAS...87.8587I. doi:10.1073/pnas.87.21.8587. PMC 55002. PMID 2172991.
- ^ “RNase H1 of Saccharomyces cerevisiae: methods and nomenclature”. Methods in Enzymology 341: 395–413. (2001-01-01). doi:10.1016/s0076-6879(01)41166-9. ISBN 978-0-12-182242-2. PMID 11582793.
- ^ “Cloning of the cDNA encoding the large subunit of human RNase HI, a homologue of the prokaryotic RNase HII”. Proceedings of the National Academy of Sciences of the United States of America 95 (22): 12872–7. (October 1998). Bibcode: 1998PNAS...9512872F. doi:10.1073/pnas.95.22.12872. PMC 23637. PMID 9789007.
- ^ “Yeast RNase H(35) is the counterpart of the mammalian RNase HI, and is evolutionarily related to prokaryotic RNase HII”. FEBS Letters 421 (1): 23–6. (January 1998). doi:10.1016/s0014-5793(97)01528-7. PMID 9462832.
- ^ “RNase H2 of Saccharomyces cerevisiae is a complex of three proteins”. Nucleic Acids Research 32 (2): 407–14. (2004-01-01). doi:10.1093/nar/gkh209. PMC 373335. PMID 14734815.

