O6-メチルグアニン-DNAメチルトランスフェラーゼ
O6-メチルグアニン-DNAメチルトランスフェラーゼ(英: O6-methylguanine DNA methyltransferase、略称: MGMT)またはO6-アルキルグアニン-DNAアルキルトランスフェラーゼ(英: O6-alkylguanine DNA alkyltransferase、略称: AGT、AGAT)は、ヒトではMGMT遺伝子にコードされるタンパク質である[5][6]。MGMTはゲノムの安定性に重要であり、自然発生する変異原性DNA損傷である6-O-メチルグアニンをグアニンに戻し、DNA修復や転写時のミスマッチやエラーを防ぐ役割を果たす。マウスでは、Mgmt遺伝子の喪失によってアルキル化試薬曝露後の発がんリスクが増大する[7]。細菌には2つのアイソザイムが存在し、Ada、Ogtと呼ばれている。
機能と機構
[編集]| O6-alkylguanine DNA alkyltransferase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 識別子 | |||||||||
| EC番号 | 2.1.1.63 | ||||||||
| CAS登録番号 | 77271-19-3 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
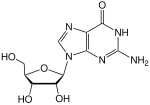
アルキル化変異原が選択的に修飾するのはグアニンのN7位であるが、DNAの主要な発がん性損傷となるのは6-O-アルキル化グアニンである。このDNA付加体は修復タンパク質MGMTによってSN2機構を介して除去される。このタンパク質は、化学量論的反応によって損傷部位からアルキル基を除去し、アルキル化後に活性型酵素が再生されることはないため、厳密な意味で酵素ではない(自殺酵素と呼ばれる)。タンパク質のメチル基受容残基はシステインである[8]。
- 6-O-メチルグアノシンからグアノシンへの脱メチル化反応
臨床的意義
[編集]悪性度の高い脳腫瘍である膠芽腫の患者の中でも、MGMT遺伝子のプロモーターがメチル化されている患者は抗がん剤テモゾロミドの効果が高い[9]。全体として、臨床予測モデルにおいてMGMT遺伝子のメチル化は患者の生存期間の長さと関係している[10]。臨床現場で行われるMGMTプロモーターのメチル化の検査としては、免疫組織学的アッセイやRNAベースのアッセイよりもメチル化特異的PCR(MS-PCR)やパイロシークエンシングといったDNAベースの手法が望ましい[11]。
また、MGMTは遺伝子治療の効果を高める有用なツールであることも示されている。目的の遺伝子とMGMTの2つのコンポーネントを含むベクターを用いることで、遺伝子導入に成功した細胞をin vivoで薬剤で選別することができる[12]。
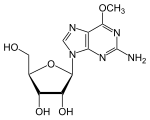
環境[13]、タバコの煙[14]、食品[15]、内在性代謝産物[16]中の変異原によって、DNAをアルキル化、より具体的にはメチル化する反応性の高い親電子種が生成され、6-O-メチルグアニン(m6G)が生成される。
1985年のYaroshによる先駆的業績によって、m6Gが最も変異原性と発がん性の高いアルキル化塩基であることが確立された[17]。1994年Rasouli-Niaらは、DNA中の対合していないm6G約8個につき1つの変異が導入されることを示した[18]。
がんにおける発現
[編集]| 種類 | 欠損の頻度 | 発がん素地における欠損の頻度 |
|---|---|---|
| 子宮頸癌[19] | 61% | 39% |
| 大腸癌 | 40–90%[20][21][22][23][24] | 11–34%[20][21] |
| マイクロサテライト不安定性を示す大腸癌[25] | 70% | 60% |
| 食道腺癌 | 71–79%[26][27] | 89%[27] |
| 食道扁平上皮癌 | 38–96%[26][28][29] | 65%[29] |
| プロモーターメチル化を伴う膠芽腫 | 44–59%[30][31] | |
| 頭頸部扁平上皮癌 | 54%[32] | |
| 肝細胞癌(C型肝炎ウイルス関連)[33] | 68% | 65% |
| 喉頭癌 | 54–61%[34][35] | 38%[35] |
| 胃癌 | 32–88%[36][37] | 17–78%[36][37] |
| 甲状腺癌[38] | 87% |
エピジェネティックな抑制
[編集]DNA修復に欠陥を有する散発性がんの中で、DNA修復遺伝子に変異を抱えているものは少数である。こうしたがんの大部分では、1つまたはそれ以上のエピジェネティックな変化によってDNA修復遺伝子の発現が低下もしくはサイレンシングされている。ある研究では、大腸がん試料113種類のうちDNA修復遺伝子MGMTにミスセンス変異が存在するものはわずか4つであったのに対し、大部分ではMGMTプロモーター領域のメチル化によってMGMTの発現が低下していた[39]。
MGMTのエピジェネティックな抑制はいくつかの方法で行われている[40]。がんでMGMTの発現が抑制されている場合、多くはプロモーター領域のメチル化によるものである[40]。一方で、ヒストンH3のリジン9番のジメチル化[41]やmiR-181d、miR-767-3p、miR-603などのmiRNAの過剰発現によって抑制が行われている場合もある[40][42][43]。
発がん素地における欠損
[編集]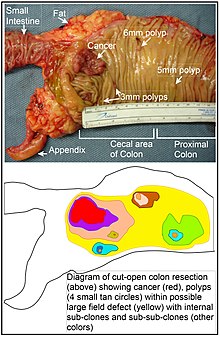
発がん素地(field defect)は、エピジェネティックな変化や変異によって、がん発生の素因となるような状態が整えられた領域である。Rubinによって指摘されているように、がん研究の大部分はin vivoでは明確に定義された腫瘍を、in vitroでは個別の腫瘍病巣を対象として行われたものである[44]。しかしながら、mutator phenotypeのヒト大腸がんでみられる体細胞変異の80%以上がクローン性増殖の開始前に生じていることを示す証拠が得られている[45]。同様にVogelsteinらは、腫瘍で同定されている体細胞変異の半数以上は新生物発生前の段階、見かけ上正常な細胞の成長時に生じたものであることを指摘している[46]。
上の表では、がん周囲の発がん素地(組織学的には正常な組織)でのMGMTの欠損が記載されている。MGMTのエピジェネティックな抑制やサイレンシング自体は、幹細胞の選択上の利点をもたらすものではないと考えられる。しかし、MGMTの発現の低下また欠損は変異率の上昇を引き起こすと考えられ、変異した遺伝子のうちの1つまたは複数が細胞に選択上の利点をもたらす可能性がある。その後、変異した幹細胞が増殖クローンを生み出す際に、発現が不十分なMGMT遺伝子は選択的に中立もしくはわずかに有害なパッセンジャー遺伝子(ヒッチハイカー遺伝子)として保有される可能性がある。MGMTをがエピジェネティックに抑制されたクローンが存在し続けることでさらなる変異が生じ続け、そのうちのいくつかから腫瘍が生じる可能性がある。
外因性損傷と欠損
[編集]MGMTの欠損だけでは、がんへの進行を引き起こすには不十分である可能性がある。Mgmtのホモ接合型変異マウスは、ストレスがない状態で生育された場合には野生型マウスより多くのがんが発生することはない[47]。一方、アゾキシメタンとデキストラン硫酸によるストレス処理を行うと、Mgmt変異マウスでは4コロニー以上の腫瘍が生じるのに対し、野生型マウスでは1以下である[48]。
他のDNA修復遺伝子との協調的な抑制
[編集]がんでは、複数のDNA修復遺伝子が同時に抑制されていることが多い[49]。MGMTが関係する例として、40種類の星細胞腫試料で27種類のDNA修復遺伝子のmRNAの発現を非患者由来の正常な脳組織と比較した研究では、星細胞腫の全てのグレード(II、III、IV)で13種類のDNA修復遺伝子(MGMT、NTHL1、OGG1、SMUG1、ERCC1、ERCC2、ERCC3、ERCC4、MLH1、MLH3、RAD50、XRCC4、XRCC5)が有意にダウンレギュレーションされていた。これら13遺伝子が高グレードだけでなく低グレードでも抑制されていることは、これらが後期段階だけでなく初期段階にも重要である可能性を示唆している[50]。他の例では、胃がんの135試料でMGMTとMLH1に対する免疫反応性は密接に相関しており、腫瘍のプログレッションの過程でMGMTとMLH1の喪失は協調的に加速しているようである[51]。
相互作用
[編集]MGMTはエストロゲン受容体αと相互作用することが示されている[52]。
出典
[編集]- ^ a b c GRCh38: Ensembl release 89: ENSG00000170430 - Ensembl, May 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000054612 - Ensembl, May 2017
- ^ Human PubMed Reference:
- ^ Mouse PubMed Reference:
- ^ “Isolation and structural characterization of a cDNA clone encoding the human DNA repair protein for O6-alkylguanine”. Proc. Natl. Acad. Sci. U.S.A. 87 (2): 686–90. (January 1990). Bibcode: 1990PNAS...87..686T. doi:10.1073/pnas.87.2.686. PMC 53330. PMID 2405387.
- ^ “Chromosomal localization of human O6-methylguanine-DNA methyltransferase (MGMT) gene by in situ hybridization”. Mutagenesis 7 (1): 83–5. (January 1992). doi:10.1093/mutage/7.1.83. PMID 1635460.
- ^ “Increased susceptibility to chemotherapeutic alkylating agents of mice deficient in DNA repair methyltransferase”. Carcinogenesis 21 (10): 1879–83. (October 2000). doi:10.1093/carcin/21.10.1879. PMID 11023546.
- ^ “MGMT: key node in the battle against genotoxicity, carcinogenicity and apoptosis induced by alkylating agents”. DNA Repair (Amst.) 6 (8): 1079–99. (August 2007). doi:10.1016/j.dnarep.2007.03.008. PMID 17485253.
- ^ “MGMT gene silencing and benefit from temozolomide in glioblastoma”. N. Engl. J. Med. 352 (10): 997–1003. (2005). doi:10.1056/NEJMoa043331. PMID 15758010.
- ^ “The combination of IDH1 mutations and MGMT methylation status predicts survival in glioblastoma better than either IDH1 or MGMT alone”. Neuro-Oncology 16 (9): 1263–73. (2014). doi:10.1093/neuonc/nou005. PMC 4136888. PMID 24510240.
- ^ Preusser, M.; Janzer, Charles R.; Felsberg, J.; Reifenberger, G.; Hamou, M. F.; Diserens, A. C.; Stupp, R.; Gorlia, T. et al. (Oct 2008). “Anti-O6-methylguanine-methyltransferase (MGMT) immunohistochemistry in glioblastoma multiforme: observer variability and lack of association with patient survival impede its use as clinical biomarker”. Brain Pathol 18 (4): 520–532. doi:10.1111/j.1750-3639.2008.00153.x. PMC 8095504. PMID 18400046.
- ^ “Erythroid-specific human factor IX delivery from in vivo selected hematopoietic stem cells following nonmyeloablative conditioning in hemophilia B mice”. Mol. Ther. 16 (10): 1745–52. (2008). doi:10.1038/mt.2008.161. PMC 2658893. PMID 18682698.
- ^ “Relevance of nitrosamines to human cancer”. Carcinogenesis 5 (11): 1381–93. (1984). doi:10.1093/carcin/5.11.1381. PMID 6386215.
- ^ “O(6)-methylguanine-DNA methyltransferase (MGMT): impact on cancer risk in response to tobacco smoke”. Mutat. Res. 736 (1–2): 64–74. (2012). doi:10.1016/j.mrfmmm.2011.06.004. PMID 21708177.
- ^ “O6-methylguanine-DNA methyltransferase in the defense against N-nitroso compounds and colorectal cancer”. Carcinogenesis 34 (11): 2435–42. (2013). doi:10.1093/carcin/bgt275. PMID 23929436.
- ^ “Endogenous DNA damage in humans: a review of quantitative data”. Mutagenesis 19 (3): 169–85. (2004). doi:10.1093/mutage/geh025. PMID 15123782.
- ^ “The role of O6-methylguanine-DNA methyltransferase in cell survival, mutagenesis and carcinogenesis”. Mutat. Res. 145 (1–2): 1–16. (1985). doi:10.1016/0167-8817(85)90034-3. PMID 3883145.
- ^ “On the quantitative relationship between O6-methylguanine residues in genomic DNA and production of sister-chromatid exchanges, mutations and lethal events in a Mer- human tumor cell line”. Mutat. Res. 314 (2): 99–113. (1994). doi:10.1016/0921-8777(94)90074-4. PMID 7510369.
- ^ “Correlation of promoter hypermethylation in hTERT, DAPK and MGMT genes with cervical oncogenesis progression”. Oncol. Rep. 22 (1): 199–204. (2009). doi:10.3892/or_00000425. PMID 19513524.
- ^ a b “MGMT promoter methylation and field defect in sporadic colorectal cancer”. J. Natl. Cancer Inst. 97 (18): 1330–8. (2005). doi:10.1093/jnci/dji275. PMID 16174854.
- ^ a b “Promoter methylation status of hMLH1, hMSH2, and MGMT genes in colorectal cancer associated with adenoma-carcinoma sequence”. Langenbecks Arch Surg 396 (7): 1017–26. (2011). doi:10.1007/s00423-011-0812-9. PMID 21706233.
- ^ “Promoter methylation status of hMLH1, MGMT, and CDKN2A/p16 in colorectal adenomas”. World J. Gastroenterol. 16 (28): 3553–60. (2010). doi:10.3748/wjg.v16.i28.3553. PMC 2909555. PMID 20653064.
- ^ “Promoter CpG island hypermethylation of the DNA repair enzyme MGMT predicts clinical response to dacarbazine in a phase II study for metastatic colorectal cancer”. Clin. Cancer Res. 19 (8): 2265–72. (2013). doi:10.1158/1078-0432.CCR-12-3518. PMID 23422094.
- ^ “Different patterns of DNA methylation of the two distinct O6-methylguanine-DNA methyltransferase (O6-MGMT) promoter regions in colorectal cancer”. Mol. Biol. Rep. 40 (5): 3851–7. (2013). doi:10.1007/s11033-012-2465-3. PMID 23271133.
- ^ “Methylation tolerance due to an O6-methylguanine DNA methyltransferase (MGMT) field defect in the colonic mucosa: an initiating step in the development of mismatch repair-deficient colorectal cancers”. Gut 59 (11): 1516–26. (2010). doi:10.1136/gut.2009.194787. PMID 20947886.
- ^ a b “O-6-methylguanine-deoxyribonucleic acid methyltransferase methylation enhances response to temozolomide treatment in esophageal cancer”. J Carcinog 12: 20. (2013). doi:10.4103/1477-3163.120632. PMC 3853796. PMID 24319345.
- ^ a b “Silencing of MGMT expression by promoter hypermethylation in the metaplasia-dysplasia-carcinoma sequence of Barrett's esophagus”. Cancer Lett. 275 (1): 117–26. (2009). doi:10.1016/j.canlet.2008.10.009. PMC 4028828. PMID 19027227.
- ^ “Aberrant methylation of different DNA repair genes demonstrates distinct prognostic value for esophageal cancer”. Dig. Dis. Sci. 56 (10): 2992–3004. (2011). doi:10.1007/s10620-011-1774-z. PMID 21674174.
- ^ a b “Promoter methylation status of MGMT, hMSH2, and hMLH1 and its relationship to corresponding protein expression and TP53 mutations in human esophageal squamous cell carcinoma”. Med. Oncol. 31 (2): 784. (2014). doi:10.1007/s12032-013-0784-4. PMID 24366688.
- ^ “Promoter methylation analysis of O6-methylguanine-DNA methyltransferase in glioblastoma: detection by locked nucleic acid based quantitative PCR using an imprinted gene (SNURF) as a reference”. BMC Cancer 10: 48. (2010). doi:10.1186/1471-2407-10-48. PMC 2843669. PMID 20167086.
- ^ “Comparative assessment of 5 methods (methylation-specific polymerase chain reaction, MethyLight, pyrosequencing, methylation-sensitive high-resolution melting, and immunohistochemistry) to analyze O6-methylguanine-DNA-methyltranferase in a series of 100 glioblastoma patients”. Cancer 118 (17): 4201–11. (2012). doi:10.1002/cncr.27392. PMID 22294349.
- ^ “Promoter methylation of MGMT, MLH1 and RASSF1A tumor suppressor genes in head and neck squamous cell carcinoma: pharmacological genome demethylation reduces proliferation of head and neck squamous carcinoma cells”. Oncol. Rep. 27 (4): 1135–41. (2012). doi:10.3892/or.2012.1624. PMC 3583513. PMID 22246327.
- ^ “Methylation of multiple genes in hepatitis C virus associated hepatocellular carcinoma”. J Adv Res 5 (1): 27–40. (2014). doi:10.1016/j.jare.2012.11.002. PMC 4294722. PMID 25685469.
- ^ “Promoter hypermethylation of CDKN2A, MGMT, MLH1, and DAPK genes in laryngeal squamous cell carcinoma and their associations with clinical profiles of the patients”. Head Neck 36 (8): 1103–8. (2014). doi:10.1002/hed.23413. PMID 23804521.
- ^ a b “Frequent hypermethylation of DAPK, RARbeta, MGMT, RASSF1A and FHIT in laryngeal squamous cell carcinomas and adjacent normal mucosa”. Oral Oncol. 47 (2): 104–7. (2011). doi:10.1016/j.oraloncology.2010.11.006. PMID 21147548.
- ^ a b “Aberrant DNA methylation of MGMT and hMLH1 genes in prediction of gastric cancer”. Genet. Mol. Res. 13 (2): 4140–5. (2014). doi:10.4238/2014.May.30.9. PMID 24938706.
- ^ a b “Promoter hypermethylation of multiple genes in early gastric adenocarcinoma and precancerous lesions”. Hum. Pathol. 40 (11): 1534–42. (2009). doi:10.1016/j.humpath.2009.01.029. PMID 19695681.
- ^ “Methylation and expression profiles of MGMT gene in thymic epithelial tumors”. Lung Cancer 83 (2): 279–87. (2014). doi:10.1016/j.lungcan.2013.12.004. PMID 24388682.
- ^ “O(6)-methylguanine methyltransferase in colorectal cancers: detection of mutations, loss of expression, and weak association with G:C>A:T transitions”. Gut 54 (6): 797–802. (June 2005). doi:10.1136/gut.2004.059535. PMC 1774551. PMID 15888787.
- ^ a b c “Regulation of expression of O6-methylguanine-DNA methyltransferase and the treatment of glioblastoma (Review)”. Int. J. Oncol. 47 (2): 417–28. (2015). doi:10.3892/ijo.2015.3026. PMC 4501657. PMID 26035292.
- ^ “Silencing effect of CpG island hypermethylation and histone modifications on O6-methylguanine-DNA methyltransferase (MGMT) gene expression in human cancer”. Oncogene 22 (55): 8835–44. (2003). doi:10.1038/sj.onc.1207183. PMID 14647440.
- ^ “A genome-wide miRNA screen revealed miR-603 as a MGMT-regulating miRNA in glioblastomas”. Oncotarget 5 (12): 4026–39. (2014). doi:10.18632/oncotarget.1974. PMC 4147303. PMID 24994119.
- ^ “miR-181d: a predictive glioblastoma biomarker that downregulates MGMT expression”. Neuro-Oncology 14 (6): 712–9. (2012). doi:10.1093/neuonc/nos089. PMC 3367855. PMID 22570426.
- ^ Rubin H (March 2011). “Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture”. BioEssays 33 (3): 224–31. doi:10.1002/bies.201000067. PMID 21254148.
- ^ “Genetic reconstruction of individual colorectal tumor histories”. Proc. Natl. Acad. Sci. U.S.A. 97 (3): 1236–41. (February 2000). Bibcode: 2000PNAS...97.1236T. doi:10.1073/pnas.97.3.1236. PMC 15581. PMID 10655514.
- ^ “Cancer genome landscapes”. Science 339 (6127): 1546–58. (March 2013). Bibcode: 2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ “Repair of endogenous DNA base lesions modulate lifespan in mice”. DNA Repair (Amst.) 21: 78–86. (2014). doi:10.1016/j.dnarep.2014.05.012. PMC 4125484. PMID 24994062.
- ^ “Both base excision repair and O6-methylguanine-DNA methyltransferase protect against methylation-induced colon carcinogenesis”. Carcinogenesis 31 (12): 2111–7. (2010). doi:10.1093/carcin/bgq174. PMC 2994278. PMID 20732909.
- ^ “Epigenetic reduction of DNA repair in progression to gastrointestinal cancer”. World J Gastrointest Oncol 7 (5): 30–46. (2015). doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ “Expression analyses of 27 DNA repair genes in astrocytoma by TaqMan low-density array”. Neurosci. Lett. 409 (2): 112–7. (2006). doi:10.1016/j.neulet.2006.09.038. PMID 17034947.
- ^ “Loss of expression of DNA repair enzymes MGMT, hMLH1, and hMSH2 during tumor progression in gastric cancer”. Gastric Cancer 6 (2): 86–95. (2003). doi:10.1007/s10120-003-0213-z. PMID 12861399.
- ^ “The modified human DNA repair enzyme O(6)-methylguanine-DNA methyltransferase is a negative regulator of estrogen receptor-mediated transcription upon alkylation DNA damage”. Mol. Cell. Biol. 21 (20): 7105–14. (October 2001). doi:10.1128/MCB.21.20.7105-7114.2001. PMC 99886. PMID 11564893.
関連文献
[編集]- “Variability and regulation of O6-alkylguanine-DNA alkyltransferase”. Carcinogenesis 24 (4): 625–35. (2003). doi:10.1093/carcin/bgg005. PMID 12727789.