「リボソーム」の版間の差分
Smilesworth (会話 | 投稿記録) m →分布・構造: リンクの追加 |
|||
| (3人の利用者による、間の5版が非表示) | |||
| 1行目: | 1行目: | ||
{{混同|リポソーム|x1=脂質二重層でできた小胞の}} |
{{混同|リポソーム|x1=脂質二重層でできた小胞の}} |
||
[[ファイル:Biological cell.svg|thumb|400px|典型的な動物細胞の模式図: (1) [[核小体]](仁)、(2) [[細胞核]]、(3) '''リボソーム'''、(4) [[小胞]]、(5) [[粗面小胞体]]、(6) [[ゴルジ体]]、(7) [[微小管]]、(8) [[滑面小胞体]]、(9) [[ミトコンドリア]]、(10) [[液胞]]、(11) [[細胞質基質]]、(12) [[リソソーム]]、(13) [[中心体]]]] |
|||
'''リボソーム'''または'''リボゾーム'''({{Lang-en-short|ribosome}}; ライボソーム)は、あらゆる[[生物]]の[[細胞]]内に存在する構造であり、[[粗面小胞体]] (rER) に付着している膜結合リボソームと細胞質中に存在する遊離リボソームがある。[[mRNA]]の[[遺伝情報]]を読み取って[[タンパク質]]へと変換する機構である[[翻訳 (生物学)|翻訳]]が行われる場である。大小2つのサブユニットから成り、これらは[[タンパク質]](リボソームタンパク、ribosomal protein)と[[リボ核酸|RNA]]([[リボソームRNA]]、rRNA; ribosomal RNA)の複合体である。[[細胞小器官]]に分類される場合もある。[[2000年]]、[[X線構造解析]]により[[立体構造]]が決定された。 |
|||
{{Organelle diagram}} |
|||
== 分布・構造 == |
|||
[[File:010 small subunit-1FKA.gif|thumb|350px|right|真正細菌''[[サーマス・サーモフィルス|Thermus thermophilus]]''の30Sサブユニットの構造。タンパク質は青色で、rRNAはオレンジ色で表されている<ref name=Schluenzen>{{cite journal |author=Schluenzen F, Tocilj A, Zarivach R, Harms J, Gluehmann M, Janell D, Bashan A, Bartels H, Agmon I, Franceschi F, Yonath A |title=Structure of functionally activated small ribosomal subunit at 3.3 angstroms resolution |journal=Cell |volume=102 |issue=5 |pages=615-23 |year=2000 |pmid=11007480 | doi = 10.1016/S0092-8674(00)00084-2 <!--Retrieved from CrossRef by DOI bot-->}}</ref>。]] |
|||
[[File:10 large subunit.gif|thumb|350px|right|古細菌(好塩菌)''[[Haloarcula marismortui]]''の50Sサブユニットの構造。タンパク質は青色で、二つのrRNAは黄色とオレンジ色で表されている<ref name=Ban>{{cite journal |author=Ban N, Nissen P, Hansen J, Moore P, Steitz T |title=The complete atomic structure of the large ribosomal subunit at 2.4 [[Ångström|A]] resolution |journal=Science |volume=289 |issue=5481 |pages=905–20 |year=2000 |pmid=10937989 | doi = 10.1126/science.289.5481.905 <!--Retrieved from CrossRef by DOI bot-->}}</ref>。中心にある小さな緑色の部分は、サブユニットの活性部位である。]] |
|||
'''リボソーム'''({{Lang-en-short|ribosome}} {{Lang-de-short|Ribosom}}、'''リボゾーム''')は、すべての[[細胞]]に存在する[[翻訳 (生物学)|生体タンパク質合成]](mRNAの翻訳)を行う[[分子機械]]である。リボソームは、[[メッセンジャーRNA|伝令RNA]](mRNA)分子の[[コドン]]によって指定された順序で[[アミノ酸]]をつなぎ合わせ、[[ポリペプチド]]鎖を形成する。リボソームは、リボソーム小サブユニットとリボソーム大サブユニットという2つの主要な構成要素からなる。それぞれのサブユニットは、1つまたは複数の[[リボソームRNA]](rRNA)分子と多数の{{Ill2|リボソームタンパク質|en|Ribosomal protein}}(RPまたはr-タンパク質)から構成されている<ref>{{cite thesis | vauthors = Konikkat S | url = http://repository.cmu.edu/dissertations/608/ | title = Dynamic Remodeling Events Drive the Removal of the ITS2 Spacer Sequence During Assembly of 60S Ribosomal Subunits in S. cerevisiae | archive-url = https://web.archive.org/web/20170803051524/http://repository.cmu.edu/dissertations/608/ | archive-date = 3 August 2017 | publisher = Carnegie Mellon University | date = February 2016 | degree = Ph.D. }}</ref><ref>{{cite book |url=https://www.google.co.in/books/edition/Allgemeine_und_molekulare_Botanik/TA9rjr034h8C?hl=en |title=Allgemeine und Molekulare Botanik |vauthors=Weiler EW, Nover L |date=2008 |publisher=Georg Thieme Verlag |isbn=9783131527912 |location=Stuttgart |pages=532 |language=de}}</ref><ref>{{cite journal | vauthors = de la Cruz J, Karbstein K, Woolford JL | title = Functions of ribosomal proteins in assembly of eukaryotic ribosomes in vivo | journal = Annual Review of Biochemistry | volume = 84 | pages = 93–129 | date = 2015 | pmid = 25706898 | pmc = 4772166 | doi = 10.1146/annurev-biochem-060614-033917 }}</ref>。リボソームとそれらが会合する分子を合わせて翻訳装置(''translational apparatus'')とも呼ぶ。 |
|||
リボソームは、RNAの情報からタンパク質を合成するという容易ならざる作業を正確に行うため、大きく複雑な構造体となっている。リボソームは50種類以上のタンパク質と、少なくとも3種類のRNA分子から構成され、[[分子量]]としては[[大腸菌]]では2.7 M[[原子質量単位|Da]]、[[哺乳類]]では4.6 MDaにもなる。また[[ミトコンドリア]]や[[葉緑体]]も独自に[[真正細菌]]のものと類似したリボソームをもつ。真正細菌と[[古細菌]]には[[細胞核]]がない。つまり[[転写 (生物学)|転写]]と翻訳が同区画で行われるため、リボソームは転写されている [[伝令RNA|mRNA]] に速やかに集まり翻訳を開始する。真正細菌や古細菌の平均的な翻訳速度は毎秒20アミノ酸で、mRNAにおける60[[ヌクレオチド]]である。この値はRNAポリメラーゼによる合成速度である毎秒50〜100ヌクレオチドに近い。真核生物では核と[[細胞質]]が[[核膜]]によって隔てられているため、mRNA は様々な修飾を受けた後、リボソームのある細胞質へと移行する必要がある。真核生物の翻訳は毎秒2〜4アミノ酸というゆっくりした速度で進む<ref name="watson">James D. Watson, T. A. Baker, S. P. Bell他 『ワトソン 遺伝子の分子生物学【第5版】』 [[中村桂子]] 監訳、[[東京電機大学]]出版局、2006年3月、p.423-430</ref>。 |
|||
== 概要 == |
|||
一本の mRNA に複数のリボソームが連結した状態をポリリボソーム(polyribosome)または[[ポリソーム]](polysome)と呼ぶ。[[小胞体]]のうち、粗面小胞体上にはリボソームが大量に結合している。 |
|||
[[File:Peptide syn.svg|thumb|200px| '''図1''': リボソームは、[[メッセンジャーRNA]]分子の配列によって制御される[[重合体|高分子]][[タンパク質]][[分子]]を組み立てる。これは、すべての生細胞や関連するウイルスにとって必要である。]] |
|||
[[タンパク質]]を構成する[[アミノ酸]]の配列を[[遺伝暗号|コード]]する[[デオキシリボ核酸|デオキシリボ核酸(DNA)]]の配列は、mRNA鎖に[[転写 (生物学)|転写]]される。リボソームはmRNAに結合し、その塩基配列から、特定のタンパク質を生成するための正しいアミノ酸の配列を決定する。アミノ酸は、[[トランスファーRNA|転移RNA]](tRNA)分子によって選択され、リボソームへと運ばれ、[[アンチコドン]]の[[ステムループ]]を介してmRNA鎖に結合する。mRNAの各コーディングトリプレット(三連符、[[コドン]])に対して、アンチコドンが正確に一致した固有のtRNAがあり、成長する[[ポリペプチド]]鎖に組み込むために正しいアミノ酸を運ぶ。タンパク質が生成されると、[[タンパク質フォールディング|折り重なって]]機能的な[[三次構造|三次元構造]]を形成することができる。 |
|||
リボソームはリボソームRNAとリボソームタンパク質の複合体である大小2つのサブユニットからなり、それぞれのサブユニットは遠心力をかけたときの沈降速度によって名づけられている。沈降速度に使われる単位は[[スベドベリ]](Svedberg、略してS)で、Sが大きいほど沈降速度が速い。真正細菌と古細菌では小さいサブユニットは30[[沈降係数|S]]サブユニット、大きいサブユニットは50Sサブユニットと呼ばれ、この2つからなる完全なリボソームは70Sリボソームと呼ばれる。30Sと50Sからなるリボソームがなぜ80Sではなく70Sなのかというと、沈降速度は質量と形態の両方で決まるためである。真核生物のリボソームは少し大きく、40Sサブユニットと60Sサブユニットからなり、あわせて80Sリボソームとなる。スベドベリ単位はrRNAを区別するのにも使われる。たとえば真核生物の60Sサブユニットには160ヌクレオチド、120ヌクレオチド、4700ヌクレオチドの3種のrRNAが含まれるが、それぞれ[[5.8S rRNA]]、[[5S rRNA]]、28S rRNAと呼ばれる<ref name=watson/>。 |
|||
リボソームは、[[リボ核酸|リボ核酸(RNA)]]とタンパク質の{{Ill2|高分子集合|en|Macromolecular assembly|label=複合体}}からなる{{Ill2|リボ核タンパク質粒子|en|Ribonucleoprotein particle|label=リボ核タンパク質複合体}}である。各リボソームは、サブユニットと呼ばれる小サブユニット(30[[スヴェドベリ (単位)|S]])と大サブユニット(50[[スヴェドベリ (単位)|S]])が互いに結合して構成される。 |
|||
リボソームの基本的な機能は全生物でおおむね共通するが、構造は各ドメインや界ごとに少しずつ異なる。例えば古細菌や真正細菌で23S rRNAと呼ばれるRNAは、真核生物では二つに分かれており、28S rRNA、5.8S rRNAと呼ばれる。タンパク質は真正細菌が57、[[ユーリ古細菌]]が63、[[クレン古細菌]]が68、真核生物が78。真正細菌のものが他の生物とやや異なる。真核生物は古細菌のものをほぼ引き継ぐ(67のタンパク質が共通する)が、新たに11のタンパク質が追加されている。 |
|||
* 小サブユニット(30S)は主に解読機能を担っており、mRNAとも結合する。 |
|||
* 大サブユニット(50S)は主に触媒機能を担っており、[[アミノアシルtRNA]]とも結合する。 |
|||
タンパク質が構成要素から合成される過程は、開始、伸長、終結、リサイクルの4つの段階で行われる。すべてのmRNA分子の[[開始コドン]]はAUGという配列を持っている。[[終止コドン]]はUAA、UAG、UGAのいずれかであり、これらのコドンを認識するtRNA分子が存在しないため、リボソームは翻訳の完了を認識する<ref>{{cite web |url=https://nature.com/scitable/definition/translation-rna-translation-173 |title=Scitable by nature translation / RNA translation |access-date=2022-11-28}}</ref>。リボソームがmRNA分子の読み取りを終えると、2つのサブユニットが分離し、通常は分解されるが、再利用されることもある。リボソームは[[リボザイム]]の一種であり、リボソームRNAは[[触媒|触媒的]]アミノ酸をに結合する{{Ill2|ペプチジルトランスフェラーゼ|en|Peptidyl transferase|label=ペプチジルトランスフェラーゼ}}活性を担っているからである<ref name="PTC">{{cite journal | vauthors = Tirumalai MR, Rivas M, Tran Q, Fox GE | title = The Peptidyl Transferase Center: a Window to the Past | journal = Microbiol Mol Biol Rev | volume = 85 | issue = 4 | pages = e0010421 | date = November 2021 | pmid = 34756086 | pmc = 8579967 | doi = 10.1128/MMBR.00104-21}}</ref>。 |
|||
リボソームは、多くの場合、[[粗面小胞体]]を構成する細胞内膜と関連している。 |
|||
[[細菌]]、[[古細菌]]、[[真核生物]]の[[3ドメイン説|3ドメイン系]]のリボソームは、互いに驚くほど似ていることから、共通の起源を示す証拠と考えられている。しかし、これらの大きさ、配列、構造、およびタンパク質とRNAの比率は異なっている。この構造の違いにより、ある種の[[抗生物質]]は細菌のリボソームを阻害して細菌を殺滅させるが、ヒトのリボソームは影響を受けない。すべての[[生物種]]において、複数のリボソームが1本のmRNA鎖に沿って同時に移動し([[ポリソーム]]と呼ばれる)、それぞれが特定の配列を「読み取り」、対応するタンパク質分子を生成することがある。 |
|||
真核細胞の{{Ill2|ミトコンドリアリボソーム|en|Mitochondrial ribosome}}は、機能的には細菌のリボソームと多くの特徴が類似しており、[[ミトコンドリア]]の進化的起源を反映しているものと考えられる<ref>{{cite journal | vauthors = Benne R, Sloof P | title = Evolution of the mitochondrial protein synthetic machinery | journal = Bio Systems | volume = 21 | issue = 1 | pages = 51–68 | year = 1987 | pmid = 2446672 | doi = 10.1016/0303-2647(87)90006-2 }}</ref><ref>{{cite web|url=http://www.cs.stedwards.edu/chem/Chemistry/CHEM43/CHEM43/Ribosomes/Ribosome.HTML|title=Ribosomes|access-date=2011-04-28|url-status=dead|archive-url=https://web.archive.org/web/20090320163538/http://www.cs.stedwards.edu/chem/Chemistry/CHEM43/CHEM43/Ribosomes/Ribosome.HTML|archive-date=2009-03-20}}</ref>。 |
|||
== 発見 == |
|||
リボソームは、1950年代半ばに{{Ill2|ルーマニア系アメリカ人|en|Romanian Americans}}の細胞生物学者[[ジョージ・エミール・パラーデ]]によって、[[電子顕微鏡]]を使って高密度の粒子または顆粒として初めて観察された<ref>{{cite journal | vauthors = Palade GE | title = A small particulate component of the cytoplasm | journal = The Journal of Biophysical and Biochemical Cytology | volume = 1 | issue = 1 | pages = 59–68 | date = January 1955 | pmid = 14381428 | pmc = 2223592 | doi = 10.1083/jcb.1.1.59 }}</ref>。その粒状の構造から、当初は'''[[パラーデ顆粒]]'''(Palade granules)と呼ばれていた。「リボソーム」という用語は、1958年末に科学者アグノーによって提案された。{{Quotation|シンポジウムの途中で、意味上の問題が明らかになった。ある参加者は、「ミクロソーム」とは、他のタンパク質や脂質の物質によって汚染されたミクロソーム画分のリボ核タンパク質粒子を意味すると考え、別の参加者は、ミクロソームは粒子によって汚染されたタンパク質と脂質から構成されていると考えた。「ミクロソーム粒子」という表現は適切とは思えず、「ミクロソーム画分のリボ核タンパク質粒子」という表現はあまりにも不自然であった。会議中に「リボソーム」という用語が提案され、これはとても納得のゆく名前であり、響きもよい。もし「リボソーム」が35-100Sサイズのリボ核タンパク質粒子を表すのに採用されれば、現在の混乱は解決するだろう。|Albert|Microsomal Particles and Protein Synthesis<ref>{{cite book | veditors = Roberts RB | date = 1958 | chapter = Introduction | title = Microsomal Particles and Protein Synthesis | url = https://archive.org/details/microsomalpartic00biop | location = New York | publisher = Pergamon Press, Inc. }}</ref>}} |
|||
1974年、[[アルベルト・クラウデ]]、[[クリスチャン・ド・デューブ]]、[[ジョージ・エミール・パラーデ]]が、リボソームの発見により[[ノーベル生理学・医学賞]]を共同受賞した<ref>{{cite web |url=https://www.nobelprize.org/nobel_prizes/medicine/laureates/1974/ |title=The Nobel Prize in Physiology or Medicine 1974 |work=Nobelprize.org |publisher=The Nobel Foundation |access-date=10 December 2012 |url-status=live |archive-url=https://web.archive.org/web/20130126131649/http://www.nobelprize.org/nobel_prizes/medicine/laureates/1974/ |archive-date=26 January 2013 }}</ref>。2009年の[[ノーベル化学賞]]は、リボソームの詳細な構造と機構の解明により、[[ヴェンカトラマン・ラマクリシュナン]]、[[トマス・A・スタイツ]]、[[アダ・ヨナス]]に贈られた<ref>{{cite web|url=http://nobelprize.org/nobel_prizes/chemistry/laureates/2009/|title=2009 Nobel Prize in Chemistry|publisher=The Nobel Foundation|access-date=10 December 2012|url-status=live|archive-url=https://web.archive.org/web/20120428061419/http://www.nobelprize.org/nobel_prizes/chemistry/laureates/2009/|archive-date=28 April 2012}}</ref>。 |
|||
== 構造 == |
|||
<!-- [[File:Ribosome Structure.png|thumb|left|原核生物と真核生物でリボソームrRNAの構成を比較した図]] --><!-- 表で代替可能のため削除する。 --> |
|||
[[Image:ribosome shape.png|thumb|'''図2''': 大腸菌 (''E.coli'') 70Sリボソームの構造と形状を異なる位置から描画した。50Sリボソーム大サブユニット(赤系)と30Sリボソーム小サブユニット(青系)を200オングストローム (20 nm) のスケールバーと共に示した。50Sサブユニットについては、23S(濃赤)、5S(橙赤色)、rRNAとリボソームタンパク質(薄赤)が見える。30Sサブユニットについては、16S rRNA(濃青)とリボソームタンパク質(薄青)が見える。]] |
|||
リボソームは、複雑な細胞内機械(''cellular machine'')である。リボソームの大部分は、[[リボソームRNA]](rRNA)と呼ばれる特殊なRNAと、数十種類の異なるタンパク質(正確な数は生物種によって多少異なる)で構成されている。リボソームタンパク質とrRNAは一般に、大サブユニットと小サブユニットと呼ばれる大きさが異なる2つのリボソーム断片の中にある。リボソームは2つのサブユニットが組み合わさって構成され(図2)、タンパク質合成の際に一体となって、協働してmRNAをポリペプチド鎖に翻訳する(図1)。大きさの異なる2つのサブユニットから形成されるため、直径よりも軸長の方がわずかに長くなっている。[[分子量]]としては、[[大腸菌]]では2.7 M[[原子質量単位|Da]]、[[哺乳類]]では4.6 MDaに達する。 |
|||
=== 原核生物のリボソーム === |
|||
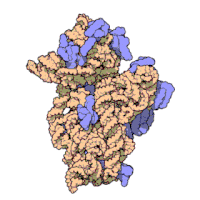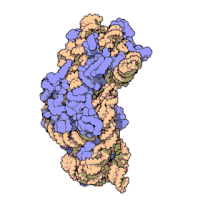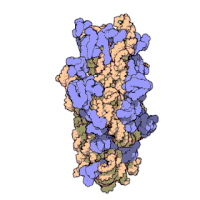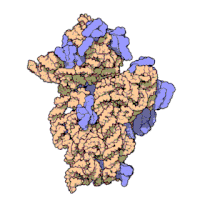
[[原核生物]]のリボソームは直径が約20 nm(200 Å)、65%のrRNAと35%の{{Ill2|リボソームタンパク質|en|Ribosomal protein}}からなる<ref>{{cite journal|author1-link=Charles Kurland| vauthors = Kurland CG |title=Molecular characterization of ribonucleic acid from Escherichia coli ribosomes|journal=Journal of Molecular Biology|volume=2|issue=2|pages=83–91|doi=10.1016/s0022-2836(60)80029-0|year=1960}}</ref>。真核生物のリボソームは直径が25-30 [[ナノメートル|nm]](250-300 Å)、rRNAとタンパク質の比率は1に近い<ref>{{cite journal | vauthors = Wilson DN, Doudna Cate JH | title = The structure and function of the eukaryotic ribosome | journal = Cold Spring Harbor Perspectives in Biology | volume = 4 | issue = 5 | pages = a011536 | date = May 2012 | pmid = 22550233 | pmc = 3331703 | doi = 10.1101/cshperspect.a011536 }}</ref>。[[結晶学|結晶学的研究]]により<ref>{{cite journal | vauthors = Nissen P, Hansen J, Ban N, Moore PB, Steitz TA | title = The structural basis of ribosome activity in peptide bond synthesis | journal = Science | volume = 289 | issue = 5481 | pages = 920–30 | date = August 2000 | pmid = 10937990 | doi = 10.1126/science.289.5481.920 | bibcode = 2000Sci...289..920N | s2cid = 8370119 | url = http://pdfs.semanticscholar.org/0baa/9ba2a4d098674984113142bbf1ffca35700c.pdf | archive-url = https://web.archive.org/web/20201130102727/http://pdfs.semanticscholar.org/0baa/9ba2a4d098674984113142bbf1ffca35700c.pdf | url-status = dead | archive-date = 2020-11-30 }}</ref>、ポリペプチド合成の[[活性部位|反応部位]]近傍にリボソームタンパク質が存在しないことが明らかになった。このことから、リボソームのタンパク質成分は、[[ペプチド結合]]形成の触媒作用には直接関与せず、むしろrRNAのタンパク質合成能力を高める足場として機能している可能性が考えられる([[リボザイム]]を参照)。[[Image:010 small subunit-1FKA.gif|thumb|200px|right|'''図3''': 細菌[[サーマス・サーモフィルス]] (''Thermus thermophilus'') 由来の30Sサブユニットの分子構造<ref name="Venki" />。タンパク質を青色で、単一のRNA鎖を茶色で示す。]] |
|||
原核生物のリボソームサブユニットと真核生物のリボソームサブユニットは非常によく似ている<ref name="Alberts_2002">{{cite book | vauthors = Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P | chapter = Membrane-bound Ribosomes Define the Rough ER | chapter-url = https://www.ncbi.nlm.nih.gov/books/NBK26841/#A2204 | title = Molecular Biology of the Cell | edition = 4th | location = New York | publisher = Garland Science | date = 2002 | isbn = 978-0-8153-4072-0 | pages = 342 }}</ref>。 |
|||
リボソームサブユニットやrRNA断片の表記に用いられる単位は[[スヴェドベリ (単位)|スヴェドベリ]](S)で、サイズではなく[[遠心分離]]における[[沈降係数|沈降速度]]を表している。たとえば細菌の70Sリボソームは50Sと30Sのサブユニットで構成されるなど、断片の名称が一致しないのはこのためである。 |
|||
原核生物は70Sリボソームを持っており、小サブユニット({{Ill2|原核生物のリボソーム小サブユニット|en|Prokaryotic small ribosomal subunit|label=30S}})と大サブユニット({{Ill2|原核生物のリボソーム大サブユニット|en|Prokaryotic large ribosomal subunit|label=50S}})のそれぞれから構成されている。 たとえば、大腸菌(''E. coli'')の小サブユニットは、[[16SリボソームRNA|16S]] RNAサブユニット(1,540[[ヌクレオチド]]からなる)を持ち、21個のタンパク質と結合している。大サブユニットは、[[5SリボソームRNA|5S]] RNAサブユニット(120ヌクレオチド)、23S RNAサブユニット(2,900ヌクレオチド)、および31個の[[タンパク質]]から構成されている<ref name="Alberts_2002" />。 |
|||
:{| class="wikitable float-right" style="text-align:center" |
|||
|+ 大腸菌 (細菌) のリボソーム<ref name="Garrett_2009" />{{rp|962}} |
|||
|- |
|||
! width="25%"| リボソーム |
|||
! width="25%"| サブユニット |
|||
! width="25%"| rRNA |
|||
! width="25%"| r-タンパク質 |
|||
|- |
|||
| rowspan="3" | 70S || rowspan="2" | 50S || 23S (2904 [[:en:Nucleotide|nt]]) || rowspan="2" | 31 |
|||
|- |
|||
| 5S (120 nt) |
|||
|- |
|||
| 30S || 16S (1542 nt) || 21 |
|||
|} |
|||
大腸菌リボソームのtRNA結合部位の{{Ill2|アフィニティーラベル|en|Affinity label|label=アフィニティーラベリング}}により、ペプチジルトランスフェラーゼ活性に関連する可能性が高いA部位およびP部位のタンパク質が同定された<ref name="PTC" />。標識されたタンパク質は、L27、L14、L15、L16、L2であり、少なくともL27は[[活性部位|ドナー部位]]に位置していることが、E. CollatzとA. P. Czernilofskyによって示された<ref>{{cite journal | vauthors = Collatz E, Küchler E, Stöffler G, Czernilofsky AP | title = The site of reaction on ribosomal protein L27 with an affinity label derivative of tRNA Met f | journal = FEBS Letters | volume = 63 | issue = 2 | pages = 283–6 | date = April 1976 | pmid = 770196 | doi = 10.1016/0014-5793(76)80112-3 | doi-access = free }}</ref><ref>{{cite journal | vauthors = Czernilofsky AP, Collatz EE, Stöffler G, Kuechler E | title = Proteins at the tRNA binding sites of Escherichia coli ribosomes | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 71 | issue = 1 | pages = 230–4 | date = January 1974 | pmid = 4589893 | pmc = 387971 | doi = 10.1073/pnas.71.1.230 | bibcode = 1974PNAS...71..230C | doi-access = free }}</ref>。さらなる研究によって、S1およびS21タンパク質が16SリボソームRNAの3′末端と関連して、翻訳開始に関与していることが確認された<ref>{{cite journal | vauthors = Czernilofsky AP, Kurland CG, Stöffler G | title = 30S ribosomal proteins associated with the 3'-terminus of 16S RNA | journal = FEBS Letters | volume = 58 | issue = 1 | pages = 281–4 | date = October 1975 | pmid = 1225593 | doi = 10.1016/0014-5793(75)80279-1 | doi-access = free }}</ref>。 |
|||
=== 古細菌のリボソーム === |
|||
古細菌のリボソームは、50S大サブユニットと30S小サブユニットからなる70Sリボソームであり、3本のrRNA鎖を含むという点で細菌と共通の側面を呈している。しかし、配列の観点では、細菌のものよりも真核生物のものにはるかに近い。古細菌が細菌に比べて持っている余分なリボソームタンパク質はすべて真核生物のものに対応するが、古細菌と細菌の間にはそのような関係はない<ref>{{cite book | vauthors = Cullen KE |title=Encyclopedia of Life Science |date=2009 |publisher=Facts On File |location=New York |isbn=9780470015902 |chapter=Archaeal Ribosomes|pages=1–5 |doi=10.1002/9780470015902.a0000293.pub3|s2cid=243730576 }}</ref><ref name="Rps">{{cite journal | vauthors = Tirumalai MR, Anane-Bediakoh D, Rajesh R, Fox GE | title = Net Charges of the Ribosomal Proteins of the ''S10'' and ''spc'' Clusters of Halophiles Are Inversely Related to the Degree of Halotolerance | journal = Microbiol. Spectrum | volume = 9 | issue = 3 | pages = e0178221 | date = November 2021 | pmid = 34908470 | pmc = 8672879 | doi = 10.1128/spectrum.01782-21}}</ref><ref>{{cite journal | vauthors = Wang J, Dasgupta I, Fox GE | title = Many nonuniversal archaeal ribosomal proteins are found in conserved gene clusters | journal = Archaea | volume = 2 | issue = 4 | pages = 241–51 | date = 28 April 2009 | pmid = 19478915 | pmc =2686390 | doi = 10.1155/2009/971494 | doi-access = free }}</ref>。 |
|||
=== 真核生物のリボソーム === |
|||
{{main|{{ill2|真核細胞のリボソーム|en|Eukaryotic ribosome}}}} |
|||
[[真核生物]]は、[[細胞質]]内に80Sリボソームを持ち、{{Ill2|真核生物のリボソーム小サブユニット (40S)|en|Eukaryotic small ribosomal subunit (40S)|label=小サブユニット (40S)}}と{{Ill2|真核生物のリボソーム大サブユニット (60S)|en|Eukaryotic small ribosomal subunit (60S)|label=大サブユニット (60S)}}のそれぞれから構成されている。小サブユニットは、{{Ill2|18SリボソームRNA|en|18S ribosomal RNA|label=18S RNA}}(1,900ヌクレオチド)と33個のタンパク質から構成されている<ref name="Ben-Shem A, Garreau de Loubresse N, Melnikov S, Jenner L, Yusupova G, Yusupov M.">{{cite journal | vauthors = Ben-Shem A, Garreau de Loubresse N, Melnikov S, Jenner L, Yusupova G, Yusupov M | title = The structure of the eukaryotic ribosome at 3.0 Å resolution | journal = Science | volume = 334 | issue = 6062 | pages = 1524–9 | date = December 2011 | pmid = 22096102 | doi = 10.1126/science.1212642 | bibcode = 2011Sci...334.1524B | s2cid = 9099683 }}</ref><ref name="Rabl, Leibundgut, Ataide, Haag, Ban 2010 730–736">{{cite journal | vauthors = Rabl J, Leibundgut M, Ataide SF, Haag A, Ban N | title = Crystal structure of the eukaryotic 40S ribosomal subunit in complex with initiation factor 1 | journal = Science | volume = 331 | issue = 6018 | pages = 730–6 | date = February 2011 | pmid = 21205638 | doi = 10.1126/science.1198308 | hdl = 20.500.11850/153130 | bibcode = 2011Sci...331..730R | s2cid = 24771575 | url = https://www.research-collection.ethz.ch/bitstream/20.500.11850/153130/1/eth-5105-01.pdf | hdl-access = free }}</ref>。大サブユニットは、[[5SリボソームRNA|5S RNA]](120ヌクレオチド)、{{Ill2|28SリボソームRNA|en|28S ribosomal RNA|label=28S RNA}}(4,700ヌクレオチド)、[[5.8SリボソームRNA|5.8S RNA]]サブユニット(160ヌクレオチド)および49個のタンパク質で構成されている<ref name="Alberts_2002" /><ref name="Ben-Shem A, Garreau de Loubresse N, Melnikov S, Jenner L, Yusupova G, Yusupov M." /><ref name="Klinge, Voigts-Hoffmann, Leibundgut, Arpagaus, Ban 2011 941–948">{{cite journal | vauthors = Klinge S, Voigts-Hoffmann F, Leibundgut M, Arpagaus S, Ban N | title = Crystal structure of the eukaryotic 60S ribosomal subunit in complex with initiation factor 6 | journal = Science | volume = 334 | issue = 6058 | pages = 941–8 | date = November 2011 | pmid = 22052974 | doi = 10.1126/science.1211204 | bibcode = 2011Sci...334..941K | s2cid = 206536444 }}</ref>。 |
|||
:{| class="wikitable float-right" style="text-align:center" |
|||
|+ 真核生物の細胞質リボソーム (ドブネズミ、''R. norvegicus'')<ref name="Garrett_2009">{{cite book | vauthors = Garrett R, Grisham CM | title = Biochemistry | edition = 4th | publisher = Cengage Learning Services | date = 2009 | isbn = 978-0-495-11464-2 }}</ref>{{rp|65}} |
|||
|- |
|||
! width="25%"| リボソーム |
|||
! width="25%"| サブユニット |
|||
! width="25%"| rRNA |
|||
! width="25%"| r-タンパク質 |
|||
|- |
|||
| rowspan="4" | 80S || rowspan="3" | 60S || 28S (4718 nt) || rowspan="3" | 49 |
|||
|- |
|||
| 5.8S (160 nt) |
|||
|- |
|||
| 5S (120 nt) |
|||
|- |
|||
| 40S || 18S (1874 nt) || 33 |
|||
|} |
|||
1977年、Czernilofskyは、{{Ill2|アフィニティーラベル|en|Affinity label|label=アフィニティーラベリング}}を用いて、ラット肝リボソームのtRNA結合部位を特定する研究を発表した。L32/33、L36、L21、L23、L28/29、L13などのいくつかのタンパク質が{{Ill2|ペプチジルトランスフェラーゼ|en|Peptidyl transferase|label=}}中心またはその近傍にあることが示唆された<ref>{{cite journal | vauthors = Fabijanski S, Pellegrini M | title = Identification of proteins at the peptidyl-tRNA binding site of rat liver ribosomes | journal = Molecular & General Genetics | volume = 184 | issue = 3 | pages = 551–6 | year = 1977 | pmid = 6950200 | doi = 10.1007/BF00431588 | s2cid = 9751945 }}</ref>。 |
|||
=== プラストリボソームとミトコンドリアリボソーム === |
|||
{{main|{{ill2|ミトコンドリアリボソーム|en|Mitochondrial ribosome}}|[[:en:Chloroplast#Chloroplast ribosomes|葉緑体#プラストリボソーム <small>(英語版)</small>]]}} |
|||
真核生物では、リボソームは[[ミトコンドリア]]({{Ill2|ミトコンドリアリボソーム|en|Mitochondrial ribosome|label=ミトリボソーム}}ともいう)や[[葉緑体]]などの[[プラスチド]](プラストリボソーム<!-- plastoribosomes -->ともいう)に存在する。また、それらは大小のサブユニットが複数の[[タンパク質]]によって結合された1つの70S粒子を構成している<ref name="Alberts_2002" />。これらのリボソームは細菌のリボソームと類似しており、これらの[[オルガネラ]]は{{Ill2|共生細菌|en|Symbiotic bacteria}}に由来すると考えられている<ref name="Alberts_2002" />。中でも葉緑体リボソームは、ミトコンドリアのものよりも細菌のものに近い。ミトコンドリア内では、リボソームRNAの多くの断片が短縮されており、動物や菌類では、[[5SリボソームRNA|5S rRNA]]が別の構造に置き換わっている<ref>{{cite journal | vauthors = Agrawal RK, Sharma MR | title = Structural aspects of mitochondrial translational apparatus | journal = Current Opinion in Structural Biology | volume = 22 | issue = 6 | pages = 797–803 | date = December 2012 | pmid = 22959417 | pmc = 3513651 | doi = 10.1016/j.sbi.2012.08.003 }}</ref>。特に原生生物である[[リーシュマニア]]・タレントラ(''Leishmania tarentolae'')は、ミトコンドリアrRNAの集合体を最小限に抑えている<ref>{{cite journal | vauthors = Sharma MR, Booth TM, Simpson L, Maslov DA, Agrawal RK | title = Structure of a mitochondrial ribosome with minimal RNA | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 106 | issue = 24 | pages = 9637–42 | date = June 2009 | pmid = 19497863 | pmc = 2700991 | doi = 10.1073/pnas.0901631106 | bibcode = 2009PNAS..106.9637S | doi-access = free }}</ref>。一方、植物のミトコンドリアは、細菌と比較して、拡張したrRNAと追加のタンパク質、特に多くの{{Ill2|ペンタトリコペプチドリピート|en|Pentatricopeptide repeat}}タンパク質の両方を持っている<ref>{{cite journal | vauthors = Waltz F, Nguyen TT, Arrivé M, Bochler A, Chicher J, Hammann P, Kuhn L, Quadrado M, Mireau H, Hashem Y, Giegé P | title = Small is big in Arabidopsis mitochondrial ribosome | journal = Nature Plants | volume = 5 | pages = 106–117 | date = January 2019 | issue = 1 | doi = 10.1038/s41477-018-0339-y | pmid = 30626926 | s2cid = 58004990 }}</ref>。 |
|||
[[クリプト藻]]や[[クロララクニオン藻]]は、真核生物の痕跡核<!-- vestigial eukaryotic nucleus -->に似た[[ヌクレオモルフ]](退化した共生体の核)を持っている可能性がある<ref name="archibald1">{{cite journal | vauthors = Archibald JM, Lane CE | title = Going, going, not quite gone: nucleomorphs as a case study in nuclear genome reduction | journal = The Journal of Heredity | volume = 100 | issue = 5 | pages = 582–90 | year = 2009 | pmid = 19617523 | doi = 10.1093/jhered/esp055 | doi-access = free }}</ref>。真核生物の80Sリボソームは、ヌクレオモルフが含まれる細胞内区画に存在する可能性がある<ref>{{Cite web|title=Specialized Internal Structures of Prokaryotes {{!}} Boundless Microbiology|url=https://courses.lumenlearning.com/boundless-microbiology/chapter/specialized-internal-structures-of-prokaryotes/|access-date=2021-09-24|website=courses.lumenlearning.com}}</ref>。 |
|||
=== 相違点の利用 === |
|||
細菌のリボソームと真核生物のリボソームの違いを利用して、感染者の細胞に害を与えずに感染細菌だけを破壊できる[[抗生物質]]が、[[医薬品化学|創薬化学者]]によって開発されている。その構造の違いから、細菌の70Sリボソームはこれらの抗生物質に対して脆弱であるが、真核生物の80Sリボソームは脆弱ではない<ref name="Recht">{{cite journal | vauthors = Recht MI, Douthwaite S, Puglisi JD | title = Basis for prokaryotic specificity of action of aminoglycoside antibiotics | journal = The EMBO Journal | volume = 18 | issue = 11 | pages = 3133–8 | date = June 1999 | pmid = 10357824 | pmc = 1171394 | doi = 10.1093/emboj/18.11.3133 }}</ref>。真核生物の[[ミトコンドリア]]は、細菌と同様のリボソームを持っているが、[[生体膜|二重膜]]に囲まれているため、これらの抗生物質がオルガネラに容易に侵入できないからである<ref>{{cite journal | vauthors = O'Brien TW | title = The general occurrence of 55 S ribosomes in mammalian liver mitochondria | journal = The Journal of Biological Chemistry | volume = 246 | issue = 10 | pages = 3409–17 | date = May 1971 | doi = 10.1016/S0021-9258(18)62239-2 | pmid = 4930061 | doi-access = free }}</ref>。注目すべき反例として、抗悪性腫瘍剤の抗生物質[[クロラムフェニコール]]は、細菌だけでなく、真核生物でもミトコンドリアの50Sリボソームを阻害することが知られている<ref>{{Cite journal|date=1970-08-17|title=Chloramphenicol-lnduced Bone Marrow Suppression|url=https://jamanetwork.com/journals/jama/fullarticle/356164|journal=JAMA|language=en|volume=213|issue=7|pages=1183–1184|doi=10.1001/jama.1970.03170330063011|pmid=5468266|issn=0098-7484}}</ref>。しかし、葉緑体のリボソームは異なり、葉緑体リボソームタンパク質に抗生物質耐性という形質を持たせるためには、[[遺伝子工学]]を利用してマーカーとして導入する必要がある<ref>{{cite journal | vauthors = Newman SM, Boynton JE, Gillham NW, Randolph-Anderson BL, Johnson AM, Harris EH | title = Transformation of chloroplast ribosomal RNA genes in Chlamydomonas: molecular and genetic characterization of integration events | journal = Genetics | volume = 126 | issue = 4 | pages = 875–88 | date = December 1990 | doi = 10.1093/genetics/126.4.875 | pmid = 1981764 | pmc = 1204285 }}</ref>。 |
|||
=== 共通の特性 === |
|||
さまざまなリボソームでは、サイズこそ大きく異なるものの、コア構造は非常によく似ている。多くのRNAは、たとえば[[同軸的スタッキング|同軸スタッキング]]を示す[[シュードノット]]のように、さまざまな[[核酸の三次構造|三次構造モチーフ]]に高度に組織化されている。大型のリボソームの余分なRNAは、いくつかの長く連続した挿入部分に含まれ<ref>{{cite journal | vauthors = Penev PI, Fakhretaha-Aval S, Patel VJ, Cannone JJ, Gutell RR, Petrov AS, Williams LD, Glass JB| title = Supersized ribosomal RNA expansion segments in Asgard archaea | journal = Genome Biology and Evolution | date = August 2020 | volume = 12 | issue = 10 | pages = 1694–1710 | pmid = 32785681 | doi = 10.1093/gbe/evaa170 | pmc = 7594248 | doi-access = free }}</ref>、コア構造を破壊したり変化させることなくループを形成している<ref name="Alberts_2002" />。リボソームの触媒活性はすべて[[リボザイム]]であるRNAによって担われており、{{Ill2|リボソームタンパク質|en|Ribosomal protein}}は表面に存在して構造を安定化させているようである<ref name="Alberts_2002" />。また、タンパク質は翻訳の開始・終結点の決定、翻訳の制御と維持も行っている<ref name="essential">Bruce Alberts, Dennis Bray, Karen Hopkin他 『Essential 細胞生物学(原書第2版)』 中村桂子・松原謙一 監訳、[[南江堂]]、2005年9月、p.251-252</ref>。 |
|||
=== 高分解能での構造 === |
|||
[[Image:10 large subunit.gif|thumb|200px|right| '''図4''': 古細菌{{Ill2|ハロアーキュラ|en|Haloarcula|label=ハロアーキュラ・マリスモルツイ}}(''Haloarcula marismortui'')の50Sサブユニットの原子構造。タンパク質は青色で、2本のRNA鎖は茶色と黄色で示されている<ref name="Ban">{{cite journal|date=August 2000|title=The complete atomic structure of the large ribosomal subunit at 2.4 A resolution|url=https://www.researchgate.net/publication/12382734_The_Complete_Atomic_Structure_of_the_Large_Ribosomal_Subunit_at_24_A_Resolution|journal=Science|volume=289|issue=5481|pages=905–20|bibcode=2000Sci...289..905B|doi=10.1126/science.289.5481.905|pmid=10937989|vauthors=Ban N, Nissen P, Hansen J, Moore PB, Steitz TA|citeseerx=10.1.1.58.2271}}</ref>。サブユニットの中央にある緑色の小片が[[活性部位]]である。]] |
|||
リボソームの一般的な分子構造は、1970年代初頭から知られていたが、2000年代初頭、その構造が数[[オングストローム]]の高分解能で明らかにされた。 |
|||
リボソームの構造を原子レベルの分解能で示した最初の論文は、2000年後半にほぼ同時に発表された。原核生物の50S大サブユニットの構造は、古細菌の{{Ill2|ハロアーキュラ|en|Haloarcula|label=ハロアーキュラ・マリスモルツイ}}(''Haloarcula marismortui'')(図4)<ref name="Ban" />と細菌の[[デイノコッカス・ラディオデュランス]](''Deinococcus radiodurans'')<ref name="Schluenzen">{{cite journal | vauthors = Schluenzen F, Tocilj A, Zarivach R, Harms J, Gluehmann M, Janell D, Bashan A, Bartels H, Agmon I, Franceschi F, Yonath A | title = Structure of functionally activated small ribosomal subunit at 3.3 angstroms resolution | journal = Cell | volume = 102 | issue = 5 | pages = 615–23 | date = September 2000 | pmid = 11007480 | doi = 10.1016/S0092-8674(00)00084-2 | s2cid = 1024446 | doi-access = free }}</ref>から、30Sサブユニットの構造は高度好熱菌の[[サーマス・サーモフィルス]](''Thermus thermophilus'')から決定された<ref name="Venki">{{cite journal | vauthors = Wimberly BT, Brodersen DE, Clemons WM, Morgan-Warren RJ, Carter AP, Vonrhein C, Hartsch T, Ramakrishnan V | title = Structure of the 30S ribosomal subunit | journal = Nature | volume = 407 | issue = 6802 | pages = 327–39 | date = September 2000 | pmid = 11014182 | doi = 10.1038/35030006 | bibcode = 2000Natur.407..327W | s2cid = 4419944 }}</ref>。これらの構造研究は2009年のノーベル化学賞を受賞した。2001年5月には、この座標を用いて、サーマス・サーモフィルスの70S粒子全体を5.5 Åの分解能で再現した(図3)<ref>{{cite journal | vauthors = Yusupov MM, Yusupova GZ, Baucom A, Lieberman K, Earnest TN, Cate JH, Noller HF | title = Crystal structure of the ribosome at 5.5 A resolution | journal = Science | volume = 292 | issue = 5518 | pages = 883–96 | date = May 2001 | pmid = 11283358 | doi = 10.1126/science.1060089 | bibcode = 2001Sci...292..883Y | s2cid = 39505192 }}</ref>。 |
|||
2005年11月、[[大腸菌]](''Escherichia coli'')の70Sリボソームの構造について、2つの論文が発表された。空のリボソームの構造は、[[X線結晶構造解析]]により3.5 Åの分解能で決定された<ref>{{cite journal | vauthors = Schuwirth BS, Borovinskaya MA, Hau CW, Zhang W, Vila-Sanjurjo A, Holton JM, Cate JH | title = Structures of the bacterial ribosome at 3.5 A resolution | journal = Science | volume = 310 | issue = 5749 | pages = 827–34 | date = November 2005 | pmid = 16272117 | doi = 10.1126/science.1117230 | bibcode = 2005Sci...310..827S | s2cid = 37382005 }}</ref>。その2週間後、新たに合成されたタンパク質鎖がタンパク質伝導路<!-- protein-conducting channel -->を通過する際のリボソームの構造を、[[低温電子顕微鏡法|低温電子顕微鏡]](クライオ電子顕微鏡)により11-15 Åの分解能で描写した論文が発表された<ref>{{cite journal | vauthors = Mitra K, Schaffitzel C, Shaikh T, Tama F, Jenni S, Brooks CL, Ban N, Frank J | title = Structure of the E. coli protein-conducting channel bound to a translating ribosome | journal = Nature | volume = 438 | issue = 7066 | pages = 318–24 | date = November 2005 | pmid = 16292303 | pmc = 1351281 | doi = 10.1038/nature04133 | bibcode = 2005Natur.438..318M }}</ref>。 |
|||
また、リボソームがtRNAやmRNAと形成する複合体の原子構造が、独立した2つのX線結晶構造解析のグループによって、2.8 Å<ref>{{cite journal | vauthors = Selmer M, Dunham CM, Murphy FV, Weixlbaumer A, Petry S, Kelley AC, Weir JR, Ramakrishnan V | title = Structure of the 70S ribosome complexed with mRNA and tRNA | journal = Science | volume = 313 | issue = 5795 | pages = 1935–42 | date = September 2006 | pmid = 16959973 | doi = 10.1126/science.1131127 | bibcode = 2006Sci...313.1935S | s2cid = 9737925 }}</ref>および3.7 Å<ref>{{cite journal | vauthors = Korostelev A, Trakhanov S, Laurberg M, Noller HF | title = Crystal structure of a 70S ribosome-tRNA complex reveals functional interactions and rearrangements | journal = Cell | volume = 126 | issue = 6 | pages = 1065–77 | date = September 2006 | pmid = 16962654 | doi = 10.1016/j.cell.2006.08.032 | s2cid = 13452915 | doi-access = free }}</ref>の分解能で初めて明らかにされた。これらの構造から、サーマス・サーモフィルスのリボソームとmRNA、および古典的なリボソーム部位に結合したtRNAとの相互作用の詳細が確認された。その後まもなく、[[シャイン・ダルガノ配列]]を含む長いmRNAとリボソームとが相互作用が、4.5-5.5 Åの分解能で可視化された<ref>{{cite journal | vauthors = Yusupova G, Jenner L, Rees B, Moras D, Yusupov M | title = Structural basis for messenger RNA movement on the ribosome | journal = Nature | volume = 444 | issue = 7117 | pages = 391–4 | date = November 2006 | pmid = 17051149 | doi = 10.1038/nature05281 | bibcode = 2006Natur.444..391Y | s2cid = 4419198 }}</ref>。 |
|||
2011年、[[出芽酵母]](''Saccharomyces cerevisiae'')の真核生物80Sリボソームの完全な原子構造が、結晶構造解析によって初めて明らかにされた<ref name="Ben-Shem A, Garreau de Loubresse N, Melnikov S, Jenner L, Yusupova G, Yusupov M." />。このモデルにより、真核生物に特有な要素の構造と、[[保存配列#普遍的に保存された遺伝子|普遍的に保存された]]コアとの相互作用が明らかになった。同時に、{{Ill2|テトラヒメナ・サーモフィラ|en|Tetrahymena thermophila}}(''Tetrahymena thermophila'')の真核生物40Sリボソームの完全な構造モデルが示され、{{Ill2|真核生物のリボソーム小サブユニット (40S)|en|Eukaryotic small ribosomal subunit (40S)|label=40Sサブユニット}}の構造と、[[翻訳 (生物学)|翻訳開始時]]における{{Ill2|EIF1|en|EIF1|label=eIF1}}と40Sサブユニットの相互作用の多くが説明された<ref name="Rabl, Leibundgut, Ataide, Haag, Ban 2010 730–736" />。同様に、真核生物の60Sサブユニットについても、テトラヒメナ・サーモフィラから{{Ill2|EIF6|en|EIF6|label=eIF6}}との複合体としてその構造が決定された<ref name="Klinge, Voigts-Hoffmann, Leibundgut, Arpagaus, Ban 2011 941–948" />。 |
|||
== 機能 == |
== 機能 == |
||
リボソームは、RNAと関連タンパク質からなる微粒子で、[[タンパク質生合成|タンパク質を合成]]する機能を持っている。これらのタンパク質は、損傷の修復や化学プロセスの制御など、多くの細胞機能に必要である。リボソームは、細胞質内を浮遊しているか、[[小胞体]]に付着しているかのどちらかである。リボソームの主な機能は、遺伝暗号をアミノ酸配列に変換し、アミノ酸[[モノマー]]からタンパク質[[ポリマー]]を構築することである。 |
|||
[[File:Peptide syn.png|thumb|left|400px|'''リボソーム'''は、一連の[[伝令RNA]]([[:en: Messenger RNA]])を読み取り、[[転移RNA]]([[:en: Transfer RNA]] (TRNA))に結びついた[[アミノ酸]]から所定の[[タンパク質]]を組み立てる。]] |
|||
[[File:Protein translation.gif|thumb|left|300px|[[小胞体]]に分泌されるタンパク質を組み立てている'''リボソーム''']] |
|||
リボソームは[[コドン]]に応じて[[tRNA]]が運んでくる[[アミノ酸]]を連結させ[[ペプチド]]鎖を作る反応を触媒する。小サブユニットには復号センター(decoding center)があり、mRNAのコドンを1つ1つ解読してtRNA と結合させる役割をもつ。大サブユニットにはペプチジル転移酵素中心(peptidyl transferanse center)があり[[ペプチド結合]]の形成に働く<ref name="watson" />。ペプチド結合形成の触媒作用の中心的な働きは、タンパク質ではなく厳密に折りたたまれたrRNAが担っている。rRNAはリボソーム内部でコアを形成し、リボソームタンパク質は通常リボソーム表面に存在して折りたたまれたrRNAの隙間を埋めている。リボソームタンパク質の主な役割はRNAコアの安定化である<ref name="essential">Bruce Alberts, Dennis Bray, Karen Hopkin他 『Essential 細胞生物学(原書第2版)』 中村桂子・松原謙一 監訳、[[南江堂]]、2005年9月、p.251-252</ref>。この他、翻訳の開始・終結地点の決定、翻訳の制御・維持などもタンパク質が行っている。 |
|||
リボソームは、ペプチジル転移とペプチジル加水分解という2つの極めて重要な生物学的プロセスにおいて触媒として機能している<ref name="PTC" /><ref name="courses.lumenlearning.com">{{cite web |title=Specialized Internal Structures of Prokaryotes |series=Boundless Microbiology |website=courses.lumenlearning.com |url=https://courses.lumenlearning.com/boundless-microbiology/chapter/specialized-internal-structures-of-prokaryotes/ |access-date=2018-09-27}}</ref>。ペプチジルトランスフェラーゼ(ペプチド転移酵素)中心は、タンパク質の伸長時にタンパク質結合を生成する役割を担っている<ref name="courses.lumenlearning.com" />。 |
|||
== リボソーム阻害剤 == |
|||
リボソームや翻訳を阻害する薬剤は生物のタンパク質の合成を停止させるために毒性を示す。例えば、毒物の[[リシン_(毒物)|リシン]]はリボソームを不活性化することで毒性を発揮する。ただしその一方で、リボソーム阻害剤は病原細菌の増殖停止を目的にした感染症の化学療法薬にも利用されている。真正細菌とヒトなどの真核生物ではリボソームの構造が異なるため、真正細菌のリボソームにのみ特異的な阻害剤は、病原細菌に対する毒性は高いがヒトに対する毒性が低い、選択的治療薬として働くためである。このような薬として、[[抗生物質]]である[[アミノグリコシド]]系化合物([[ストレプトマイシン]]、[[ネオマイシン]]、[[カナマイシン]])や[[テトラサイクリン]]、[[クロラムフェニコール]]、[[マクロライド]]系化合物などが挙げられる。 |
|||
まとめると、リボソームにはメッセージの解読とペプチド結合の生成という2つの大きな機能がある。この2つの機能は、リボソームのサブユニットに存在する。各サブユニットは、1つまたは複数のrRNAと、いくつかのr-タンパク質から構成されている。小サブユニット(細菌と古細菌は30S、真核生物は40S)は解読機能を担っているのに対し、大サブユニット(細菌と古細菌は50S、真核生物は60S)はペプチド結合の形成を触媒し、これをペプチジルトランスフェラーゼ活性(ペプチド転移酵素活性)という。細菌と古細菌の小サブユニットは16S rRNAと21個のr-タンパク質(大腸菌)を含み、真核生物の小サブユニットは18S rRNAと32個のr-タンパク質(出芽酵母、数は種によって異なる)を含む。細菌の大サブユニットには5Sおよび23S rRNAと34個のr-タンパク質(大腸菌)を含み、真核生物の大サブユニットは5S、5.8Sおよび25S/28S rRNAと46個のr-タンパク質(出芽酵母の場合、正確な数は種によって異なる)を含む<ref>{{cite journal |author1=Lafontaine, D. |author2=Tollervey, D. |year=2001 |title=The function and synthesis of ribosomes |journal=Nat Rev Mol Cell Biol |volume=2 |issue=7 |pages=514–520 |doi=10.1038/35080045|pmid=11433365 |hdl=1842/729 |s2cid=2637106 }}</ref>。 |
|||
== 脚注 == |
|||
{{脚注ヘルプ}} |
|||
=== 翻訳 === |
|||
{{Reflist}} |
|||
{{Main|翻訳 (生物学)}} |
|||
<!-- == 参考文献 == --> |
|||
リボソームは、[[伝令RNA|mRNA]]を[[タンパク質]]に翻訳する過程「[[タンパク質生合成]]」の場である。mRNAは一連の[[コドン]]からなり、リボソームによって解読され、タンパク質が作られる。リボソームは、mRNAを鋳型として、mRNAの各コドン(3連の[[ヌクレオチド]])を走査して、[[アミノアシルtRNA]]が運ぶ適切なアミノ酸と対になるようにする。アミノアシルtRNAは、片方の端にコドンと相補的な[[アンチコドン]]、もう片方の端に適切なアミノ酸を持っている。コドンは、コドンとアンチコドンの対合と、小サブユニットの復号中心(decoding center)との相互作用によって認識される'''<ref name="watson">James D. Watson, T. A. Baker, S. P. Bell他 『ワトソン 遺伝子の分子生物学【第5版】』 [[中村桂子]] 監訳、[[東京電機大学]]出版局、2006年3月、p.423-430</ref>'''。大規模な立体構造変化([[立体配座選択]]という)を利用して、適切なtRNAを迅速かつ正確に同定する<ref name="pmid23582332">{{cite journal | vauthors = Savir Y, Tlusty T | date = April 2013 | title = The ribosome as an optimal decoder: A lesson in molecular recognition | journal = Cell | volume = 153 | issue = 2 | pages = 471–479 | pmid = 23582332 | doi = 10.1016/j.cell.2013.03.032 | doi-access = free | bibcode = 2013APS..MARY46006T }}</ref>。リボソーム小サブユニットは、通常、最初のアミノ酸である[[メチオニン]]を運ぶアミノアシルtRNAに結合し、mRNA上のAUGコドンに結合してリボソーム大サブユニットを動員する。リボソームにはA、P、Eと呼ばれる3つのRNA結合部位があり、{{Ill2|A部位|en|A-site}}はアミノアシルtRNAまたは終結因子<ref>{{cite journal | vauthors = Korkmaz G, Sanyal S | date = September 2017 | title = ''Escherichia coli'' | journal = The Journal of Biological Chemistry | volume = 292 | issue = 36 | pages = 15134–15142 | pmid = 28743745 | pmc = 5592688 | doi = 10.1074/jbc.M117.785238 | doi-access = free }}</ref><ref name="pmid14681588">{{cite journal | vauthors = Konevega AL, Soboleva NG, Makhno VI, Semenkov YP, Wintermeyer W, Rodnina MV, Katunin VI | date = January 2004 | title = Purine bases at position 37 of tRNA stabilize codon-anticodon interaction in the ribosomal A site by stacking and {{nobr|Mg{{sup|2+}}-dependent}} interactions | journal = RNA | volume = 10 | issue = 1 | pages = 90–101 | pmid = 14681588 | pmc = 1370521 | doi = 10.1261/rna.5142404 }}</ref>、{{Ill2|P部位|en|P-site}}はペプチジルtRNA(ポリペプチド鎖に結合したtRNA)、{{Ill2|E部位|en|E-site}}(出口)は遊離tRNAをそれぞれ結合させる。タンパク質合成は、mRNAの5'末端付近にある開始コドンAUGから始まる。mRNAは、最初にリボソームのP部位に結合する。リボソームは、原核生物では[[シャイン・ダルガノ配列]]を、真核生物では[[コザック配列]]を用いて、mRNAの開始コドンを認識する。 |
|||
[[ペプチド結合]]の触媒作用には、RNAのP部位[[アデノシン]]のC2[[ヒドロキシル基|ヒドロキシル]]がプロトンシャトル機構で関与し、タンパク質合成の他の段階(転移など)は、タンパク質の[[立体構造]]の変化によって引き起こされる。リボソームは、[[活性部位|触媒コア]]がRNAでできていることから「[[リボザイム]]」に分類され<ref>{{cite journal | vauthors = Rodnina MV, Beringer M, Wintermeyer W | date = January 2007 | title = How ribosomes make peptide bonds | journal = Trends in Biochemical Sciences | volume = 32 | issue = 1 | pages = 20–26 | pmid = 17157507 | doi = 10.1016/j.tibs.2006.11.007 }}</ref>、[[RNAワールド]]の名残の可能性があると考えられている<ref>{{cite journal | author = Cech, T.R. | date = August 2000 | title = Structural biology. The ribosome is a ribozyme | journal = Science | volume = 289 | issue = 5481 | pages = 878–879 | pmid = 10960319 | doi = 10.1126/science.289.5481.878 | s2cid = 24172338 }}</ref>。 |
|||
[[File:Ribosomer i arbete.png|frame|'''図5''': リボソーム(2)によりmRNA(1)が<span style="color:#AA00AA;">ポリペプチド鎖</span>(3)に翻訳される様子を表す模式図(<span style="color:#0000AA;">小サブユニット</span>と<span style="color:#AA0000;">大サブユニット</span>で表示)。翻訳はRNAの[[開始コドン]](<span style="color:#00AA00;">AUG</span>)から始まり、[[終止コドン]](<span style="color:#00AA00;">UAG</span>)で終了する。]] |
|||
図5では、リボソームの<span style="color:#0000AA;">小サブユニット</span>と<span style="color:#AA0000;">大サブユニット</span>の両方が開始コドン(mRNAの5'末端側)に集まっている。リボソームは、mRNA上の現在のコドン(トリプレット、三連符)に一致するtRNAを用いて、ポリペプチド鎖にアミノ酸を付加する。この作業は、リボソームがmRNAの3'末端に向かって移動する間に、mRNA上の各トリプレットに対して行われる。通常、細菌の細胞では、1つのmRNAに対して複数のリボソームが並行して働き、ポリリボソーム(''polyribosome'')または[[ポリソーム]]と呼ばれるものを形成している。 |
|||
細菌や古細菌では、翻訳速度は平均して1秒間に20アミノ酸、つまりmRNA上の60[[ヌクレオチド]]であり、これはRNAポリメラーゼによる1秒間に50-100ヌクレオチドに近い合成速度である。真核生物では、翻訳速度は1秒間に2-4アミノ酸と遅い速度で進行する<ref name="essential">Bruce Alberts, Dennis Bray, Karen Hopkin他 『Essential 細胞生物学(原書第2版)』 中村桂子・松原謙一 監訳、[[南江堂]]、2005年9月、p.251-252</ref>。 |
|||
==== 共翻訳フォールディング ==== |
|||
リボソームは[[タンパク質フォールディング|タンパク質のフォールディング]]に積極的に関与していることが知られている<ref>{{cite journal | vauthors = Banerjee D, Sanyal S | date = October 2014 | title = Protein folding activity of the ribosome (PFAR) – a target for antiprion compounds | journal = Viruses | volume = 6 | issue = 10 | pages = 3907–3924 | pmid = 25341659 | pmc = 4213570 | doi = 10.3390/v6103907 | doi-access = free }}</ref><ref>{{cite journal | vauthors = Fedorov AN, Baldwin TO | date = December 1997 | title = Cotranslational protein folding | journal = The Journal of Biological Chemistry | volume = 272 | issue = 52 | pages = 32715–32718 | pmid = 9407040 | doi = 10.1074/jbc.272.52.32715 | doi-access = free }}</ref>。このようにして得られた構造は、最終産物に至る経路は異なることがあるが、通常、タンパク質の化学的リフォールディングで得られた構造と同じである<ref>{{cite journal | vauthors = Baldwin RL | date = June 1975 | title = Intermediates in protein folding reactions and the mechanism of protein folding | journal = Annual Review of Biochemistry | volume = 44 | issue = 1 | pages = 453–475 | pmid = 1094916 | doi = 10.1146/annurev.bi.44.070175.002321 }}</ref><ref>{{cite journal | vauthors = Das D, Das A, Samanta D, Ghosh J, Dasgupta S, Bhattacharya A, Basu A, Sanyal S, Das Gupta C | date = August 2008 | title = Role of the ribosome in protein folding | journal = Biotechnology Journal | volume = 3 | issue = 8 | pages = 999–1009 | pmid = 18702035 | doi = 10.1002/biot.200800098 | url = http://repository.ias.ac.in/8881/1/313.pdf }}</ref>。場合によっては、機能的なタンパク質の形状を得るために、リボソームが重要な役割を果たすこともある。たとえば、{{Ill2|結び目タンパク質|en|Knotted protein|label=結び目の深いタンパク質}}の[[タンパク質フォールディング|フォールディング]]機構として、リボソームがタンパク質鎖を押し出してくっついたループを通過させることが考えられる<ref>{{cite journal | vauthors = Dabrowski-Tumanski P, Piejko M, Niewieczerzal S, Stasiak A, Sulkowska JI | date = December 2018 | title = Protein knotting by active threading of nascent polypeptide chain exiting from the ribosome exit channel | journal = The Journal of Physical Chemistry B | volume = 122 | issue = 49 | pages = 11616–11625 | pmid = 30198720 | doi = 10.1021/acs.jpcb.8b07634 | s2cid = 52176392 }}</ref>。 |
|||
=== 翻訳非依存的なアミノ酸付加 === |
|||
リボソーム品質管理タンパク質[https://www.uniprot.org/uniprotkb/Q12532/entry Rqc2]の存在は、mRNA非依存的なタンパク質の伸長と関連している<ref>{{cite journal | vauthors = Brandman O, Stewart-Ornstein J, Wong D, Larson A, Williams CC, Li GW, Zhou S, King D, Shen PS, Weibezahn J, Dunn JG, Rouskin S, Inada T, Frost A, Weissman JS | date = November 2012 | title = A ribosome-bound quality control complex triggers degradation of nascent peptides and signals translation stress | journal = Cell | volume = 151 | issue = 5 | pages = 1042–1054 | pmid = 23178123 | pmc = 3534965 | doi = 10.1016/j.cell.2012.10.044 }}</ref><ref>{{cite journal | vauthors = Defenouillère Q, Yao Y, Mouaikel J, Namane A, Galopier A, Decourty L, Doyen A, Malabat C, Saveanu C, Jacquier A, Fromont-Racine M | date = March 2013 | title = Cdc48-associated complex bound to 60S particles is required for the clearance of aberrant translation products | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 110 | issue = 13 | pages = 5046–5051 | pmid = 23479637 | pmc = 3612664 | doi = 10.1073/pnas.1221724110 | doi-access = free | bibcode = 2013PNAS..110.5046D }}</ref>。この伸長は、Rqc2が運ぶtRNAを介して、リボソームが'''''CAT'''''テールを付加するものである。リボソームは、停止したタンパク質の[[C末端]]を、[[アラニン]]と[[スレオニン]]の無作為で翻訳非依存的な配列で伸長する<ref>{{cite journal | vauthors = Shen PS, Park J, Qin Y, Li X, Parsawar K, Larson MH, Cox J, Cheng Y, Lambowitz AM, Weissman JS, Brandman O, Frost A | date = January 2015 | title = Protein synthesis. Rqc2p and 60S ribosomal subunits mediate mRNA-independent elongation of nascent chains | journal = Science | volume = 347 | issue = 6217 | pages = 75–78 | pmid = 25554787 | pmc = 4451101 | doi = 10.1126/science.1259724 | bibcode = 2015Sci...347...75S }}</ref><ref>{{cite press release |author1=Keeley, J. |author2=Gutnikoff, R. |date=2015-01-02 |df=dmy-all |title=Ribosome studies turn up new mechanism of protein synthesis |publisher=[[:en:Howard Hughes Medical Institute|Howard Hughes Medical Institute]] |url=http://www.hhmi.org/news/ribosome-studies-turn-new-mechanism-protein-synthesis |access-date=2015-01-16 |url-status=live |archive-url=https://web.archive.org/web/20150112034722/http://www.hhmi.org/news/ribosome-studies-turn-new-mechanism-protein-synthesis |archive-date=2015-01-12 }}</ref>(''CAT''は、C末端、アラニン、スレオニンのそれぞれの頭文字をとった造語)。 |
|||
== リボソームの位置 == |
|||
リボソームは「遊離(型)リボソーム (free ribosomes)」と「膜結合(型)リボソーム (Membrane-bound ribosomes)」に分類される。[[Image:Protein translation.gif|thumb|200px|'''図6''': 翻訳のアニメーション: 真核生物の翻訳における伸長段階と膜結合段階。リボソームは緑と黄色、tRNAは紺色、関与するタンパク質は水色で示されている。生成したタンパク質(ペプチド)は[[小胞体]]に分泌される。]] |
|||
遊離リボソームと膜結合リボソームは、空間的な分布が異なるだけで、構造は同じである。リボソームが遊離状態と膜結合状態のどちらで存在するかは、合成されるタンパク質上の[[シグナルペプチド|小胞体ターゲティング・シグナル配列]]<!-- ER-targeting signal sequence -->の有無に依存する。したがって個々のリボソームは、あるタンパク質を合成しているときは膜結合状態であり、別のタンパク質を作っているときは細胞質で遊離している可能性がある<ref>{{Cite journal|last=Kobayashi|author=小林 幹|first=Kan|year=2018|date=2018|title=シグナル認識粒子とシグナル認識粒子受容体に よる新生タンパク質の小胞体膜ターゲティング 機構の構造基盤 (Structural Basis for the Nascent Protein Targeting to Endoplasmic Reticulum by SRP and SR)|url=https://www.jstage.jst.go.jp/article/biophys/58/5/58_255/_article/-char/ja/|journal=生物物理|volume=58|issue=5|pages=255–257|language=en|doi=10.2142/biophys.58.255|issn=0582-4052}}</ref>。 |
|||
リボソームは[[オルガネラ]](''organelle、''細胞小器官)と呼ばれることもあるが、オルガネラという用語はもっぱら[[生体膜|リン脂質膜]]を持った細胞分画を指すことが多く、全体が粒子状であるリボソームはオルガネラではない。このため、リボソームは「非膜系オルガネラ(non-membranous organelles)」と呼ばれることもある。 |
|||
=== 遊離リボソーム === |
|||
遊離リボソームは、[[細胞質]]内を自由に移動することができるが、[[細胞核]]や他のオルガネラからは排除されている。遊離リボソームで作られたタンパク質は細胞質内に放出され、細胞内で利用される。細胞質は高濃度の[[グルタチオン]]が存在する[[酸化還元反応|還元性環境]]であるため、酸化されたシステイン残基から形成される[[ジスルフィド結合]]を含むタンパク質は細胞質内で生成されない。 |
|||
=== 膜結合リボソーム === |
|||
リボソームが、どれかのオルガネラで必要なタンパク質を合成し始めると、そのタンパク質を作っているリボソームが「膜結合型」になることがある。真核細胞では、小胞体(ER)の[[粗面小胞体]]と呼ばれる領域でこの現象が起こる。新たに生成されたポリペプチド鎖は、{{Ill2|ベクター生成|en|Vectorial synthesis}}中のリボソームから小胞体に直接挿入され、[[分泌|分泌経路]]を経て目的地に送り届けられる。膜結合リボソームは通常、[[細胞膜]](原形質膜)内で使用されるタンパク質や、[[エキソサイトーシス]]により細胞外に排出されるタンパク質を生成する<ref>{{cite book | vauthors = Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P | chapter = Membrane-bound Ribosomes Define the Rough ER | chapter-url = https://www.ncbi.nlm.nih.gov/books/NBK26841/#A2204 | title = Molecular Biology of the Cell | edition = 4th | location = New York | publisher = Garland Science | date = 2002 | isbn = 978-0-8153-4072-0 }}</ref>。 |
|||
== 生合成 == |
|||
{{Main|リボソーム生合成|en:Ribosome biogenesis<!-- 日本語版の強化を待つため追加した -->}} |
|||
原核細胞では、リボソームは細胞質内で合成され、複数のリボソーム遺伝子[[オペロン]]が転写されることで作られる。真核生物では、このプロセスは細胞質および[[細胞核]]内の[[核小体]]の両方で行われる。この組立過程では、4つのrRNAの合成と[[プロセシング]]、およびそれらのrRNAとリボソームタンパク質の組み立てに200以上のタンパク質が協調して機能することが必要である<ref name="Kressler">{{cite journal|last1=Kressler|first1=Dieter|last2=Hurt|first2=Ed|last3=Babler|first3=Jochen|date=2009|title=Driving ribosome assembly|url=http://doc.rero.ch/record/17283/files/kressler_dra.pdf|journal=Biochimica et Biophysica Acta (BBA) - Molecular Cell Research|volume=1803|issue=6|pages=673–683|doi=10.1016/j.bbamcr.2009.10.009|pmid=19879902}}</ref>。 |
|||
== 起源 == |
|||
リボソームは[[RNAワールド]]で誕生し、自己複製する複合体として出現し、その後、[[アミノ酸]]が出現したときにタンパク質を合成する能力を進化させたのではないかと考えられている<ref name=":0">{{cite journal | vauthors = Noller HF | title = Evolution of protein synthesis from an RNA world | journal = Cold Spring Harbor Perspectives in Biology | volume = 4 | issue = 4 | pages = a003681 | date = April 2012 | pmid = 20610545 | pmc = 3312679 | doi = 10.1101/cshperspect.a003681 }}</ref>。古代のリボソームはすべて[[リボソームRNA|rRNA]]で構成され、[[ペプチド結合]]を合成する能力を発達させたのではないかとする研究もある<ref>{{cite book | vauthors = Dabbs ER | date = 1986 | title = Mutant studies on the prokaryotic ribosome. | publisher = Springer-Verlag | location = New York }}</ref><ref>{{cite journal | vauthors = Noller HF, Hoffarth V, Zimniak L | title = Unusual resistance of peptidyl transferase to protein extraction procedures | journal = Science | volume = 256 | issue = 5062 | pages = 1416–9 | date = June 1992 | pmid = 1604315 | doi = 10.1126/science.1604315 | bibcode = 1992Sci...256.1416N }}</ref><ref>{{cite journal | vauthors = Nomura M, Mizushima S, Ozaki M, Traub P, Lowry CV | title = Structure and function of ribosomes and their molecular components | journal = Cold Spring Harbor Symposia on Quantitative Biology | volume = 34 | pages = 49–61 | year = 1969 | pmid = 4909519 | doi = 10.1101/sqb.1969.034.01.009 }}</ref><ref name="pmid21930590">{{cite journal | vauthors = Krupkin M, Matzov D, Tang H, Metz M, Kalaora R, Belousoff MJ, Zimmerman E, Bashan A, Yonath A | title = A vestige of a prebiotic bonding machine is functioning within the contemporary ribosome | journal = Phil. Trans. R. Soc. B | volume = 366 | issue = 1580 | pages = 2972–8 | date = Oct 2011 | pmid = 21930590 | doi = 10.1098/rstb.2011.0146 | pmc = 3158926 }}</ref><ref name="pmid35137169">{{cite journal | vauthors = Bose T, Fridkin G, Davidovich C, Krupkin M, Dinger N, Falkovich AH, Peleg Y, Agmon I, Bashan A, Yonat A | title = Origin of life: protoribosome forms peptide bonds and links RNA and protein dominated worlds | journal = Nucleic Acids Res | volume = 50 | issue = 4 | pages = 1815–1828 | date = Feb 2022 | pmid = 35137169 | doi = 10.1093/nar/gkac052 | pmc = 8886871 }}</ref>。さらに、古代のリボソームは自己複製複合体であったことを示唆する強い証拠があり、リボソーム内のrRNAは、リボソームの自己複製に必要な[[転移RNA|tRNA]]やタンパク質をコードし、情報伝達・構造・触媒の目的を持っていた可能性がある<ref name=":1">{{cite journal | vauthors = Root-Bernstein M, Root-Bernstein R | title = The ribosome as a missing link in the evolution of life | journal = Journal of Theoretical Biology | volume = 367 | pages = 130–158 | date = February 2015 | pmid = 25500179 | doi = 10.1016/j.jtbi.2014.11.025 | doi-access = free }}</ref>。自己複製RNAを持ち、DNAを持たない仮説上の細胞生物は、リボサイト(ribocyte)またはリボセル(ribocells)と呼ばれる<ref name="Yarus">{{cite journal | vauthors = Yarus M | title = Primordial genetics: phenotype of the ribocyte | journal = Annual Review of Genetics | volume = 36 | pages = 125–51 | year = 2002 | pmid = 12429689 | doi = 10.1146/annurev.genet.36.031902.105056 }}</ref><ref>{{cite book | doi=10.1007/978-94-007-4899-6_3|chapter = The Origin of Virions and Virocells: The Escape Hypothesis Revisited|title = Viruses: Essential Agents of Life| pages=43–60|year = 2012| vauthors = Forterre P, Krupovic M | isbn=978-94-007-4898-9}}</ref>。 |
|||
原始的な条件下でRNAワールドにアミノ酸が徐々に出現すると<ref>{{cite journal | vauthors = Caetano-Anollés G, Seufferheld MJ | title = The coevolutionary roots of biochemistry and cellular organization challenge the RNA world paradigm | journal = Journal of Molecular Microbiology and Biotechnology | volume = 23 | issue = 1–2 | pages = 152–77 | year = 2013 | pmid = 23615203 | doi = 10.1159/000346551 | s2cid = 41725226 }}</ref><ref>{{cite journal | vauthors = Saladino R, Botta G, Pino S, Costanzo G, Di Mauro E | title = Genetics first or metabolism first? The formamide clue | journal = Chemical Society Reviews | volume = 41 | issue = 16 | pages = 5526–65 | date = August 2012 | pmid = 22684046 | doi = 10.1039/c2cs35066a | hdl = 11573/494138 }}</ref>、アミノ酸との相互作用によって、触媒となるRNA分子の機能範囲と効率がそれぞれ向上する<ref name=":0" />。したがって、リボソームの形態が古代の自己複製機械から現在の翻訳機械へと進化した原動力は、リボソームの[[自己複製機械|自己複製機構]]にタンパク質を組み込んで自己複製能力を高めることを可能にする[[選択 (進化)|選択圧]]である可能性がある<ref name=":1" /><ref>{{cite journal | vauthors = Fox GE| title = Origin and Evolution of the Ribosome| journal = Cold Spring Harb Perspect Biol | volume = 2 | issue = 9 | date = September 2010 | pages = a003483| pmid = 20534711 | doi = 10.1101/cshperspect.a003483| pmc = 2926754| doi-access = free }}</ref><ref>{{cite book | vauthors = Fox GE | veditors = Hernández G, Jagus R| year = 2016| title = Evolution of the Protein Synthesis Machinery and Its Regulation| chapter= Origins and early evolution of the ribosome | url = https://doi.org/10.1007/978-3-319-39468-8 |pages=31–60|location = Switzerland | publisher = Springer, Cham| doi = 10.1007/978-3-319-39468-8| isbn=978-3-319-39468-8 | s2cid = 27493054}}</ref>。 |
|||
== リボソームの不均一性 == |
|||
同じ真核細胞内でも細胞質リボソームとミトコンドリアリボソームが存在することで明示されているように、リボソームは、生物種間だけでなく同一細胞内でも組成が不均質である。一部の研究者は、哺乳動物のリボソームタンパク質の組成の不均一性が遺伝子調節に重要であることを示唆している。これは特殊化リボソーム仮説(specialized ribosome hypothesis)である<ref name="Shi Fujii Kovary Genuth 2017 pp. 71–83.e7">{{cite journal | vauthors = Shi Z, Fujii K, Kovary KM, Genuth NR, Röst HL, Teruel MN, Barna M | title = Heterogeneous Ribosomes Preferentially Translate Distinct Subpools of mRNAs Genome-wide | journal = Molecular Cell | volume = 67 | issue = 1 | pages = 71–83.e7 | date = July 2017 | pmid = 28625553 | pmc = 5548184 | doi = 10.1016/j.molcel.2017.05.021 | publisher = Elsevier BV | doi-access = free }}</ref><ref name="Xue Barna pp. 355–369">{{cite journal | vauthors = Xue S, Barna M | title = Specialized ribosomes: a new frontier in gene regulation and organismal biology | journal = Nature Reviews. Molecular Cell Biology | volume = 13 | issue = 6 | pages = 355–369 | date = May 2012 | pmid = 22617470 | pmc = 4039366 | doi = 10.1038/nrm3359 | publisher = Springer Science and Business Media LLC }}</ref>。しかし、この仮説は論争の的であり、研究が進行中のトピックである<ref name="Ferretti Karbstein pp. 521–538">{{cite journal | vauthors = Ferretti MB, Karbstein K | title = Does functional specialization of ribosomes really exist? | journal = RNA | volume = 25 | issue = 5 | pages = 521–538 | date = May 2019 | pmid = 30733326 | pmc = 6467006 | doi = 10.1261/rna.069823.118 | publisher = Cold Spring Harbor Laboratory | doi-access = free }}</ref><ref name="Farley-Barnes Ogawa Baserga 2019 pp. 754–767">{{cite journal | vauthors = Farley-Barnes KI, Ogawa LM, Baserga SJ | title = Ribosomopathies: Old Concepts, New Controversies | journal = Trends in Genetics | volume = 35 | issue = 10 | pages = 754–767 | date = October 2019 | pmid = 31376929 | pmc = 6852887 | doi = 10.1016/j.tig.2019.07.004 | publisher = Elsevier BV }}</ref>。 |
|||
リボソーム組成の不均一性は、Vince Mauroと[[ジェラルド・モーリス・エデルマン|ジェラルド・エデルマン]]によって、タンパク質合成の翻訳制御に関与することが初めて提案された<ref>{{cite journal | vauthors = Mauro VP, Edelman GM | title = The ribosome filter hypothesis | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 99 | issue = 19 | pages = 12031–6 | date = September 2002 | pmid = 12221294 | pmc = 129393 | doi = 10.1073/pnas.192442499 | bibcode = 2002PNAS...9912031M | doi-access = free }}</ref>。彼らは、リボソームの制御機能を説明するために、リボソームフィルター仮説を提唱した。その結果、異なる細胞集団に特異的な特殊化リボソームが、遺伝子の翻訳方法に影響を与える可能性が示唆された<ref>{{cite journal | vauthors = Xue S, Barna M | title = Specialized ribosomes: a new frontier in gene regulation and organismal biology | journal = Nature Reviews. Molecular Cell Biology | volume = 13 | issue = 6 | pages = 355–69 | date = May 2012 | pmid = 22617470 | pmc = 4039366 | doi = 10.1038/nrm3359 }}</ref>。いくつかのリボソームタンパク質は、組み立てられた複合体から[[細胞質]]のコピーと差し替えられ<ref>{{cite journal | vauthors = Mathis AD, Naylor BC, Carson RH, Evans E, Harwell J, Knecht J, Hexem E, Peelor FF, Miller BF, Hamilton KL, Transtrum MK, Bikman BT, Price JC | title = Mechanisms of In Vivo Ribosome Maintenance Change in Response to Nutrient Signals | journal = Molecular & Cellular Proteomics | volume = 16 | issue = 2 | pages = 243–254 | date = February 2017 | pmid = 27932527 | pmc = 5294211 | doi = 10.1074/mcp.M116.063255 }}</ref>、まったく新しいリボソームを合成しなくても、生体内のリボソームの構造を変更できることが示唆された。 |
|||
リボソームタンパク質には、細胞の生存に限りなく重要なものもあれば、そうでないものもある。[[出芽酵母]]では、14/78のリボソームタンパク質が成長に必須ではないが、ヒトでは、これらは研究対象の細胞によって異なる<ref name="Steffen McCormick Pham MacKay pp. 107–118">{{cite journal | vauthors = Steffen KK, McCormick MA, Pham KM, MacKay VL, Delaney JR, Murakami CJ, Kaeberlein M, Kennedy BK | display-authors = 6 | title = Ribosome deficiency protects against ER stress in Saccharomyces cerevisiae | journal = Genetics | volume = 191 | issue = 1 | pages = 107–118 | date = May 2012 | pmid = 22377630 | pmc = 3338253 | doi = 10.1534/genetics.111.136549 | publisher = Genetics Society of America }}</ref>。その他の不均一性には、アセチル化、メチル化、およびリン酸化などのリボソームタンパク質の[[翻訳後修飾]]が含まれる<ref>{{cite journal | vauthors = Lee SW, Berger SJ, Martinović S, Pasa-Tolić L, Anderson GA, Shen Y, Zhao R, Smith RD | display-authors = 6 | title = Direct mass spectrometric analysis of intact proteins of the yeast large ribosomal subunit using capillary LC/FTICR | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 99 | issue = 9 | pages = 5942–5947 | date = April 2002 | pmid = 11983894 | pmc = 122881 | doi = 10.1073/pnas.082119899 | doi-access = free | bibcode = 2002PNAS...99.5942L }}</ref>。シロイヌナズナ(''Arabidopsis'')<ref>{{cite journal | vauthors = Carroll AJ, Heazlewood JL, Ito J, Millar AH | title = Analysis of the Arabidopsis cytosolic ribosome proteome provides detailed insights into its components and their post-translational modification | journal = Molecular & Cellular Proteomics | volume = 7 | issue = 2 | pages = 347–369 | date = February 2008 | pmid = 17934214 | doi = 10.1074/mcp.m700052-mcp200 | doi-access = free }}</ref><ref>{{cite journal | vauthors = Odintsova TI, Müller EC, Ivanov AV, Egorov TA, Bienert R, Vladimirov SN, Kostka S, Otto A, Wittmann-Liebold B, Karpova GG | display-authors = 6 | title = Characterization and analysis of posttranslational modifications of the human large cytoplasmic ribosomal subunit proteins by mass spectrometry and Edman sequencing | journal = Journal of Protein Chemistry | volume = 22 | issue = 3 | pages = 249–258 | date = April 2003 | pmid = 12962325 | doi = 10.1023/a:1025068419698 | s2cid = 10710245 }}</ref><ref>{{cite journal | vauthors = Yu Y, Ji H, Doudna JA, Leary JA | title = Mass spectrometric analysis of the human 40S ribosomal subunit: native and HCV IRES-bound complexes | journal = Protein Science | volume = 14 | issue = 6 | pages = 1438–1446 | date = June 2005 | pmid = 15883184 | pmc = 2253395 | doi = 10.1110/ps.041293005 }}</ref><ref>{{cite journal | vauthors = Zeidan Q, Wang Z, De Maio A, Hart GW | title = O-GlcNAc cycling enzymes associate with the translational machinery and modify core ribosomal proteins | journal = Molecular Biology of the Cell | volume = 21 | issue = 12 | pages = 1922–1936 | date = June 2010 | pmid = 20410138 | pmc = 2883937 | doi = 10.1091/mbc.e09-11-0941 }}</ref>やウイルスの[[配列内リボソーム進入部位]](IRES)は、組成的に異なるリボソームによって翻訳を媒介する可能性がある。たとえば、酵母や哺乳動物細胞のリボソームタンパク質{{Ill2|40Sリボソームタンパク質S25|en|40S ribosomal protein S25|label=eS25}}を持たない40Sリボソームサブユニットは、{{Ill2|クリパウイルスのリボソーム進入部位|en|Cripavirus internal ribosome entry site|label=CrPV IGR IRES}}を動員することができない<ref>{{cite journal | vauthors = Landry DM, Hertz MI, Thompson SR | title = RPS25 is essential for translation initiation by the Dicistroviridae and hepatitis C viral IRESs | journal = Genes & Development | volume = 23 | issue = 23 | pages = 2753–2764 | date = December 2009 | pmid = 19952110 | pmc = 2788332 | doi = 10.1101/gad.1832209 }}</ref>。 |
|||
リボソームRNAの修飾の不均一性は、構造維持や機能において重要な役割を果たしており、ほとんどのmRNA修飾は[[保存配列|高度に保存された領域]]に見られる<ref>{{cite journal | vauthors = Decatur WA, Fournier MJ | title = rRNA modifications and ribosome function | journal = Trends in Biochemical Sciences | volume = 27 | issue = 7 | pages = 344–51 | date = July 2002 | pmid = 12114023 | doi = 10.1016/s0968-0004(02)02109-6 }}</ref><ref>{{cite journal | vauthors = Natchiar SK, Myasnikov AG, Kratzat H, Hazemann I, Klaholz BP | title = Visualization of chemical modifications in the human 80S ribosome structure | journal = Nature | volume = 551 | issue = 7681 | pages = 472–477 | date = November 2017 | pmid = 29143818 | doi = 10.1038/nature24482 | bibcode = 2017Natur.551..472N | s2cid = 4465175 }}</ref>。最も一般的なrRNA修飾はリボースの[[シュードウリジル化]]と{{Ill2|2'-O-メチル化|en|2'-O-methylation}}である<ref name=":2">{{cite journal | vauthors = Guo H | title = Specialized ribosomes and the control of translation | journal = Biochemical Society Transactions | volume = 46 | issue = 4 | pages = 855–869 | date = August 2018 | pmid = 29986937 | doi = 10.1042/BST20160426 | s2cid = 51609077 }}</ref>。 |
|||
== 関連項目 == |
== 関連項目 == |
||
{{Div col|colwidth=18em}} |
|||
<!-- {{Commonscat|Ribosom}} --> |
|||
* [[アミノグリコシド系抗生物質]] - アミノ変性グリコシド(アミノ糖)を分子に含む抗生物質でタンパク質合成を阻害する |
|||
* [[リボソームRNA]] |
|||
* [[分子マシン#生体分子機械|生体分子機械]] - 生体内に存在する最も複雑な高分子機械 |
|||
* [[リボソーム生合成]] |
|||
* [[翻訳後修飾]] - タンパク質の生合成の後に起こる一般的には酵素による修飾 |
|||
* [[トマス・A・スタイツ]] |
|||
* {{ill2|タンパク質動力学|en|Protein dynamics}} - タンパク質の立体構造やその状態間の遷移を扱う学問領域 |
|||
* [[アダ・ヨナス]] |
|||
* [[核酸の三次構造]] - 核酸ポリマーの三次元的な形状 |
|||
* [[ヴェンカトラマン・ラマクリシュナン]] |
|||
* [[翻訳 (生物学)]] - 細胞質または小胞体でリボソームがタンパク質を合成する過程 |
|||
* [[ゆらぎ塩基対]] - RNA分子の2つのヌクレオチド間で、ワトソン=クリック塩基対合則に従わない塩基対 |
|||
{{Div col end}} |
|||
== 脚注 == |
|||
{{reflist|25em}} |
|||
== 外部リンク == |
== 外部リンク == |
||
{{commons category|Ribosomes}} |
|||
* [https://www.nig.ac.jp/museum/OLD-MS/dataroom-x/translation/01_introduction/index.html リボソームとは?] - [[国立遺伝学研究所]]マルチメディア資料館 |
* [https://www.nig.ac.jp/museum/OLD-MS/dataroom-x/translation/01_introduction/index.html リボソームとは?] - [[国立遺伝学研究所]]マルチメディア資料館 |
||
*{{PDB Molecule of the Month|10}} |
* {{PDB Molecule of the Month|10}} |
||
*{{PDB Molecule of the Month|121}} |
* {{PDB Molecule of the Month|121}} |
||
* {{PDB Molecule of the Month|81}} |
|||
* [https://web.archive.org/web/20061027104335/http://www.cytochemistry.net/Cell-biology/ribosome.htm リボソームの役割], Gwen V. Childs, copied [https://web.archive.org/web/20071024162617/http://cellbio.utmb.edu/cellbio/ribosome.htm here] {{En icon}} |
|||
* [http://www.proteopedia.org/wiki/index.php/Ribosome Ribosome] in [http://www.proteopedia.org ''Proteopedia'']—The free, collaborative 3D encyclopedia of proteins & other molecules{{En icon}} |
|||
* [http://www.expasy.org/cgi-bin/lists?ribosomp.txt Ribosomal proteins families in ExPASy] {{Webarchive|url=https://web.archive.org/web/20110430181946/http://expasy.org/cgi-bin/lists?ribosomp.txt |date=2011-04-30 }}{{En icon}} |
|||
* [http://www.pdbe.org/emsearch/ribosome リボソームの3次元電子顕微鏡構造 EM Data Bank (EMDB)] {{En icon}} |
|||
{{四次構造}} |
|||
{{細胞小器官}} |
{{細胞小器官}} |
||
{{ |
{{四次構造}} |
||
{{Normdaten}} |
{{Normdaten}} |
||
{{DEFAULTSORT:りほそむ}} |
|||
{{DEFAULTSORT:りほそおむ}} |
|||
[[Category:リボザイム]] |
[[Category:リボザイム]] |
||
[[Category:タンパク質生合成]] |
[[Category:タンパク質生合成]] |
||
2022年12月11日 (日) 08:06時点における版
| 細胞生物学 | |
|---|---|
| 動物細胞の模式図 | |
 |
リボソーム(英: ribosome 独: Ribosom、リボゾーム)は、すべての細胞に存在する生体タンパク質合成(mRNAの翻訳)を行う分子機械である。リボソームは、伝令RNA(mRNA)分子のコドンによって指定された順序でアミノ酸をつなぎ合わせ、ポリペプチド鎖を形成する。リボソームは、リボソーム小サブユニットとリボソーム大サブユニットという2つの主要な構成要素からなる。それぞれのサブユニットは、1つまたは複数のリボソームRNA(rRNA)分子と多数のリボソームタンパク質(RPまたはr-タンパク質)から構成されている[1][2][3]。リボソームとそれらが会合する分子を合わせて翻訳装置(translational apparatus)とも呼ぶ。
概要

タンパク質を構成するアミノ酸の配列をコードするデオキシリボ核酸(DNA)の配列は、mRNA鎖に転写される。リボソームはmRNAに結合し、その塩基配列から、特定のタンパク質を生成するための正しいアミノ酸の配列を決定する。アミノ酸は、転移RNA(tRNA)分子によって選択され、リボソームへと運ばれ、アンチコドンのステムループを介してmRNA鎖に結合する。mRNAの各コーディングトリプレット(三連符、コドン)に対して、アンチコドンが正確に一致した固有のtRNAがあり、成長するポリペプチド鎖に組み込むために正しいアミノ酸を運ぶ。タンパク質が生成されると、折り重なって機能的な三次元構造を形成することができる。
リボソームは、リボ核酸(RNA)とタンパク質の複合体からなるリボ核タンパク質複合体である。各リボソームは、サブユニットと呼ばれる小サブユニット(30S)と大サブユニット(50S)が互いに結合して構成される。
- 小サブユニット(30S)は主に解読機能を担っており、mRNAとも結合する。
- 大サブユニット(50S)は主に触媒機能を担っており、アミノアシルtRNAとも結合する。
タンパク質が構成要素から合成される過程は、開始、伸長、終結、リサイクルの4つの段階で行われる。すべてのmRNA分子の開始コドンはAUGという配列を持っている。終止コドンはUAA、UAG、UGAのいずれかであり、これらのコドンを認識するtRNA分子が存在しないため、リボソームは翻訳の完了を認識する[4]。リボソームがmRNA分子の読み取りを終えると、2つのサブユニットが分離し、通常は分解されるが、再利用されることもある。リボソームはリボザイムの一種であり、リボソームRNAは触媒的アミノ酸をに結合するペプチジルトランスフェラーゼ活性を担っているからである[5]。
リボソームは、多くの場合、粗面小胞体を構成する細胞内膜と関連している。
細菌、古細菌、真核生物の3ドメイン系のリボソームは、互いに驚くほど似ていることから、共通の起源を示す証拠と考えられている。しかし、これらの大きさ、配列、構造、およびタンパク質とRNAの比率は異なっている。この構造の違いにより、ある種の抗生物質は細菌のリボソームを阻害して細菌を殺滅させるが、ヒトのリボソームは影響を受けない。すべての生物種において、複数のリボソームが1本のmRNA鎖に沿って同時に移動し(ポリソームと呼ばれる)、それぞれが特定の配列を「読み取り」、対応するタンパク質分子を生成することがある。
真核細胞のミトコンドリアリボソームは、機能的には細菌のリボソームと多くの特徴が類似しており、ミトコンドリアの進化的起源を反映しているものと考えられる[6][7]。
発見
リボソームは、1950年代半ばにルーマニア系アメリカ人の細胞生物学者ジョージ・エミール・パラーデによって、電子顕微鏡を使って高密度の粒子または顆粒として初めて観察された[8]。その粒状の構造から、当初はパラーデ顆粒(Palade granules)と呼ばれていた。「リボソーム」という用語は、1958年末に科学者アグノーによって提案された。
シンポジウムの途中で、意味上の問題が明らかになった。ある参加者は、「ミクロソーム」とは、他のタンパク質や脂質の物質によって汚染されたミクロソーム画分のリボ核タンパク質粒子を意味すると考え、別の参加者は、ミクロソームは粒子によって汚染されたタンパク質と脂質から構成されていると考えた。「ミクロソーム粒子」という表現は適切とは思えず、「ミクロソーム画分のリボ核タンパク質粒子」という表現はあまりにも不自然であった。会議中に「リボソーム」という用語が提案され、これはとても納得のゆく名前であり、響きもよい。もし「リボソーム」が35-100Sサイズのリボ核タンパク質粒子を表すのに採用されれば、現在の混乱は解決するだろう。 — Albert、Microsomal Particles and Protein Synthesis[9]
1974年、アルベルト・クラウデ、クリスチャン・ド・デューブ、ジョージ・エミール・パラーデが、リボソームの発見によりノーベル生理学・医学賞を共同受賞した[10]。2009年のノーベル化学賞は、リボソームの詳細な構造と機構の解明により、ヴェンカトラマン・ラマクリシュナン、トマス・A・スタイツ、アダ・ヨナスに贈られた[11]。
構造

リボソームは、複雑な細胞内機械(cellular machine)である。リボソームの大部分は、リボソームRNA(rRNA)と呼ばれる特殊なRNAと、数十種類の異なるタンパク質(正確な数は生物種によって多少異なる)で構成されている。リボソームタンパク質とrRNAは一般に、大サブユニットと小サブユニットと呼ばれる大きさが異なる2つのリボソーム断片の中にある。リボソームは2つのサブユニットが組み合わさって構成され(図2)、タンパク質合成の際に一体となって、協働してmRNAをポリペプチド鎖に翻訳する(図1)。大きさの異なる2つのサブユニットから形成されるため、直径よりも軸長の方がわずかに長くなっている。分子量としては、大腸菌では2.7 MDa、哺乳類では4.6 MDaに達する。
原核生物のリボソーム
原核生物のリボソームは直径が約20 nm(200 Å)、65%のrRNAと35%のリボソームタンパク質からなる[12]。真核生物のリボソームは直径が25-30 nm(250-300 Å)、rRNAとタンパク質の比率は1に近い[13]。結晶学的研究により[14]、ポリペプチド合成の反応部位近傍にリボソームタンパク質が存在しないことが明らかになった。このことから、リボソームのタンパク質成分は、ペプチド結合形成の触媒作用には直接関与せず、むしろrRNAのタンパク質合成能力を高める足場として機能している可能性が考えられる(リボザイムを参照)。
原核生物のリボソームサブユニットと真核生物のリボソームサブユニットは非常によく似ている[16]。
リボソームサブユニットやrRNA断片の表記に用いられる単位はスヴェドベリ(S)で、サイズではなく遠心分離における沈降速度を表している。たとえば細菌の70Sリボソームは50Sと30Sのサブユニットで構成されるなど、断片の名称が一致しないのはこのためである。
原核生物は70Sリボソームを持っており、小サブユニット(30S)と大サブユニット(50S)のそれぞれから構成されている。 たとえば、大腸菌(E. coli)の小サブユニットは、16S RNAサブユニット(1,540ヌクレオチドからなる)を持ち、21個のタンパク質と結合している。大サブユニットは、5S RNAサブユニット(120ヌクレオチド)、23S RNAサブユニット(2,900ヌクレオチド)、および31個のタンパク質から構成されている[16]。
大腸菌 (細菌) のリボソーム[17]:962 リボソーム サブユニット rRNA r-タンパク質 70S 50S 23S (2904 nt) 31 5S (120 nt) 30S 16S (1542 nt) 21
大腸菌リボソームのtRNA結合部位のアフィニティーラベリングにより、ペプチジルトランスフェラーゼ活性に関連する可能性が高いA部位およびP部位のタンパク質が同定された[5]。標識されたタンパク質は、L27、L14、L15、L16、L2であり、少なくともL27はドナー部位に位置していることが、E. CollatzとA. P. Czernilofskyによって示された[18][19]。さらなる研究によって、S1およびS21タンパク質が16SリボソームRNAの3′末端と関連して、翻訳開始に関与していることが確認された[20]。
古細菌のリボソーム
古細菌のリボソームは、50S大サブユニットと30S小サブユニットからなる70Sリボソームであり、3本のrRNA鎖を含むという点で細菌と共通の側面を呈している。しかし、配列の観点では、細菌のものよりも真核生物のものにはるかに近い。古細菌が細菌に比べて持っている余分なリボソームタンパク質はすべて真核生物のものに対応するが、古細菌と細菌の間にはそのような関係はない[21][22][23]。
真核生物のリボソーム
真核生物は、細胞質内に80Sリボソームを持ち、小サブユニット (40S)と大サブユニット (60S)のそれぞれから構成されている。小サブユニットは、18S RNA(1,900ヌクレオチド)と33個のタンパク質から構成されている[24][25]。大サブユニットは、5S RNA(120ヌクレオチド)、28S RNA(4,700ヌクレオチド)、5.8S RNAサブユニット(160ヌクレオチド)および49個のタンパク質で構成されている[16][24][26]。
真核生物の細胞質リボソーム (ドブネズミ、R. norvegicus)[17]:65 リボソーム サブユニット rRNA r-タンパク質 80S 60S 28S (4718 nt) 49 5.8S (160 nt) 5S (120 nt) 40S 18S (1874 nt) 33
1977年、Czernilofskyは、アフィニティーラベリングを用いて、ラット肝リボソームのtRNA結合部位を特定する研究を発表した。L32/33、L36、L21、L23、L28/29、L13などのいくつかのタンパク質がペプチジルトランスフェラーゼ中心またはその近傍にあることが示唆された[27]。
プラストリボソームとミトコンドリアリボソーム
真核生物では、リボソームはミトコンドリア(ミトリボソームともいう)や葉緑体などのプラスチド(プラストリボソームともいう)に存在する。また、それらは大小のサブユニットが複数のタンパク質によって結合された1つの70S粒子を構成している[16]。これらのリボソームは細菌のリボソームと類似しており、これらのオルガネラは共生細菌に由来すると考えられている[16]。中でも葉緑体リボソームは、ミトコンドリアのものよりも細菌のものに近い。ミトコンドリア内では、リボソームRNAの多くの断片が短縮されており、動物や菌類では、5S rRNAが別の構造に置き換わっている[28]。特に原生生物であるリーシュマニア・タレントラ(Leishmania tarentolae)は、ミトコンドリアrRNAの集合体を最小限に抑えている[29]。一方、植物のミトコンドリアは、細菌と比較して、拡張したrRNAと追加のタンパク質、特に多くのペンタトリコペプチドリピートタンパク質の両方を持っている[30]。
クリプト藻やクロララクニオン藻は、真核生物の痕跡核に似たヌクレオモルフ(退化した共生体の核)を持っている可能性がある[31]。真核生物の80Sリボソームは、ヌクレオモルフが含まれる細胞内区画に存在する可能性がある[32]。
相違点の利用
細菌のリボソームと真核生物のリボソームの違いを利用して、感染者の細胞に害を与えずに感染細菌だけを破壊できる抗生物質が、創薬化学者によって開発されている。その構造の違いから、細菌の70Sリボソームはこれらの抗生物質に対して脆弱であるが、真核生物の80Sリボソームは脆弱ではない[33]。真核生物のミトコンドリアは、細菌と同様のリボソームを持っているが、二重膜に囲まれているため、これらの抗生物質がオルガネラに容易に侵入できないからである[34]。注目すべき反例として、抗悪性腫瘍剤の抗生物質クロラムフェニコールは、細菌だけでなく、真核生物でもミトコンドリアの50Sリボソームを阻害することが知られている[35]。しかし、葉緑体のリボソームは異なり、葉緑体リボソームタンパク質に抗生物質耐性という形質を持たせるためには、遺伝子工学を利用してマーカーとして導入する必要がある[36]。
共通の特性
さまざまなリボソームでは、サイズこそ大きく異なるものの、コア構造は非常によく似ている。多くのRNAは、たとえば同軸スタッキングを示すシュードノットのように、さまざまな三次構造モチーフに高度に組織化されている。大型のリボソームの余分なRNAは、いくつかの長く連続した挿入部分に含まれ[37]、コア構造を破壊したり変化させることなくループを形成している[16]。リボソームの触媒活性はすべてリボザイムであるRNAによって担われており、リボソームタンパク質は表面に存在して構造を安定化させているようである[16]。また、タンパク質は翻訳の開始・終結点の決定、翻訳の制御と維持も行っている[38]。
高分解能での構造

リボソームの一般的な分子構造は、1970年代初頭から知られていたが、2000年代初頭、その構造が数オングストロームの高分解能で明らかにされた。
リボソームの構造を原子レベルの分解能で示した最初の論文は、2000年後半にほぼ同時に発表された。原核生物の50S大サブユニットの構造は、古細菌のハロアーキュラ・マリスモルツイ(Haloarcula marismortui)(図4)[39]と細菌のデイノコッカス・ラディオデュランス(Deinococcus radiodurans)[40]から、30Sサブユニットの構造は高度好熱菌のサーマス・サーモフィルス(Thermus thermophilus)から決定された[15]。これらの構造研究は2009年のノーベル化学賞を受賞した。2001年5月には、この座標を用いて、サーマス・サーモフィルスの70S粒子全体を5.5 Åの分解能で再現した(図3)[41]。
2005年11月、大腸菌(Escherichia coli)の70Sリボソームの構造について、2つの論文が発表された。空のリボソームの構造は、X線結晶構造解析により3.5 Åの分解能で決定された[42]。その2週間後、新たに合成されたタンパク質鎖がタンパク質伝導路を通過する際のリボソームの構造を、低温電子顕微鏡(クライオ電子顕微鏡)により11-15 Åの分解能で描写した論文が発表された[43]。
また、リボソームがtRNAやmRNAと形成する複合体の原子構造が、独立した2つのX線結晶構造解析のグループによって、2.8 Å[44]および3.7 Å[45]の分解能で初めて明らかにされた。これらの構造から、サーマス・サーモフィルスのリボソームとmRNA、および古典的なリボソーム部位に結合したtRNAとの相互作用の詳細が確認された。その後まもなく、シャイン・ダルガノ配列を含む長いmRNAとリボソームとが相互作用が、4.5-5.5 Åの分解能で可視化された[46]。
2011年、出芽酵母(Saccharomyces cerevisiae)の真核生物80Sリボソームの完全な原子構造が、結晶構造解析によって初めて明らかにされた[24]。このモデルにより、真核生物に特有な要素の構造と、普遍的に保存されたコアとの相互作用が明らかになった。同時に、テトラヒメナ・サーモフィラ(Tetrahymena thermophila)の真核生物40Sリボソームの完全な構造モデルが示され、40Sサブユニットの構造と、翻訳開始時におけるeIF1と40Sサブユニットの相互作用の多くが説明された[25]。同様に、真核生物の60Sサブユニットについても、テトラヒメナ・サーモフィラからeIF6との複合体としてその構造が決定された[26]。
機能
リボソームは、RNAと関連タンパク質からなる微粒子で、タンパク質を合成する機能を持っている。これらのタンパク質は、損傷の修復や化学プロセスの制御など、多くの細胞機能に必要である。リボソームは、細胞質内を浮遊しているか、小胞体に付着しているかのどちらかである。リボソームの主な機能は、遺伝暗号をアミノ酸配列に変換し、アミノ酸モノマーからタンパク質ポリマーを構築することである。
リボソームは、ペプチジル転移とペプチジル加水分解という2つの極めて重要な生物学的プロセスにおいて触媒として機能している[5][47]。ペプチジルトランスフェラーゼ(ペプチド転移酵素)中心は、タンパク質の伸長時にタンパク質結合を生成する役割を担っている[47]。
まとめると、リボソームにはメッセージの解読とペプチド結合の生成という2つの大きな機能がある。この2つの機能は、リボソームのサブユニットに存在する。各サブユニットは、1つまたは複数のrRNAと、いくつかのr-タンパク質から構成されている。小サブユニット(細菌と古細菌は30S、真核生物は40S)は解読機能を担っているのに対し、大サブユニット(細菌と古細菌は50S、真核生物は60S)はペプチド結合の形成を触媒し、これをペプチジルトランスフェラーゼ活性(ペプチド転移酵素活性)という。細菌と古細菌の小サブユニットは16S rRNAと21個のr-タンパク質(大腸菌)を含み、真核生物の小サブユニットは18S rRNAと32個のr-タンパク質(出芽酵母、数は種によって異なる)を含む。細菌の大サブユニットには5Sおよび23S rRNAと34個のr-タンパク質(大腸菌)を含み、真核生物の大サブユニットは5S、5.8Sおよび25S/28S rRNAと46個のr-タンパク質(出芽酵母の場合、正確な数は種によって異なる)を含む[48]。
翻訳
リボソームは、mRNAをタンパク質に翻訳する過程「タンパク質生合成」の場である。mRNAは一連のコドンからなり、リボソームによって解読され、タンパク質が作られる。リボソームは、mRNAを鋳型として、mRNAの各コドン(3連のヌクレオチド)を走査して、アミノアシルtRNAが運ぶ適切なアミノ酸と対になるようにする。アミノアシルtRNAは、片方の端にコドンと相補的なアンチコドン、もう片方の端に適切なアミノ酸を持っている。コドンは、コドンとアンチコドンの対合と、小サブユニットの復号中心(decoding center)との相互作用によって認識される[49]。大規模な立体構造変化(立体配座選択という)を利用して、適切なtRNAを迅速かつ正確に同定する[50]。リボソーム小サブユニットは、通常、最初のアミノ酸であるメチオニンを運ぶアミノアシルtRNAに結合し、mRNA上のAUGコドンに結合してリボソーム大サブユニットを動員する。リボソームにはA、P、Eと呼ばれる3つのRNA結合部位があり、A部位はアミノアシルtRNAまたは終結因子[51][52]、P部位はペプチジルtRNA(ポリペプチド鎖に結合したtRNA)、E部位(出口)は遊離tRNAをそれぞれ結合させる。タンパク質合成は、mRNAの5'末端付近にある開始コドンAUGから始まる。mRNAは、最初にリボソームのP部位に結合する。リボソームは、原核生物ではシャイン・ダルガノ配列を、真核生物ではコザック配列を用いて、mRNAの開始コドンを認識する。
ペプチド結合の触媒作用には、RNAのP部位アデノシンのC2ヒドロキシルがプロトンシャトル機構で関与し、タンパク質合成の他の段階(転移など)は、タンパク質の立体構造の変化によって引き起こされる。リボソームは、触媒コアがRNAでできていることから「リボザイム」に分類され[53]、RNAワールドの名残の可能性があると考えられている[54]。

図5では、リボソームの小サブユニットと大サブユニットの両方が開始コドン(mRNAの5'末端側)に集まっている。リボソームは、mRNA上の現在のコドン(トリプレット、三連符)に一致するtRNAを用いて、ポリペプチド鎖にアミノ酸を付加する。この作業は、リボソームがmRNAの3'末端に向かって移動する間に、mRNA上の各トリプレットに対して行われる。通常、細菌の細胞では、1つのmRNAに対して複数のリボソームが並行して働き、ポリリボソーム(polyribosome)またはポリソームと呼ばれるものを形成している。
細菌や古細菌では、翻訳速度は平均して1秒間に20アミノ酸、つまりmRNA上の60ヌクレオチドであり、これはRNAポリメラーゼによる1秒間に50-100ヌクレオチドに近い合成速度である。真核生物では、翻訳速度は1秒間に2-4アミノ酸と遅い速度で進行する[38]。
共翻訳フォールディング
リボソームはタンパク質のフォールディングに積極的に関与していることが知られている[55][56]。このようにして得られた構造は、最終産物に至る経路は異なることがあるが、通常、タンパク質の化学的リフォールディングで得られた構造と同じである[57][58]。場合によっては、機能的なタンパク質の形状を得るために、リボソームが重要な役割を果たすこともある。たとえば、結び目の深いタンパク質のフォールディング機構として、リボソームがタンパク質鎖を押し出してくっついたループを通過させることが考えられる[59]。
翻訳非依存的なアミノ酸付加
リボソーム品質管理タンパク質Rqc2の存在は、mRNA非依存的なタンパク質の伸長と関連している[60][61]。この伸長は、Rqc2が運ぶtRNAを介して、リボソームがCATテールを付加するものである。リボソームは、停止したタンパク質のC末端を、アラニンとスレオニンの無作為で翻訳非依存的な配列で伸長する[62][63](CATは、C末端、アラニン、スレオニンのそれぞれの頭文字をとった造語)。
リボソームの位置
リボソームは「遊離(型)リボソーム (free ribosomes)」と「膜結合(型)リボソーム (Membrane-bound ribosomes)」に分類される。

遊離リボソームと膜結合リボソームは、空間的な分布が異なるだけで、構造は同じである。リボソームが遊離状態と膜結合状態のどちらで存在するかは、合成されるタンパク質上の小胞体ターゲティング・シグナル配列の有無に依存する。したがって個々のリボソームは、あるタンパク質を合成しているときは膜結合状態であり、別のタンパク質を作っているときは細胞質で遊離している可能性がある[64]。
リボソームはオルガネラ(organelle、細胞小器官)と呼ばれることもあるが、オルガネラという用語はもっぱらリン脂質膜を持った細胞分画を指すことが多く、全体が粒子状であるリボソームはオルガネラではない。このため、リボソームは「非膜系オルガネラ(non-membranous organelles)」と呼ばれることもある。
遊離リボソーム
遊離リボソームは、細胞質内を自由に移動することができるが、細胞核や他のオルガネラからは排除されている。遊離リボソームで作られたタンパク質は細胞質内に放出され、細胞内で利用される。細胞質は高濃度のグルタチオンが存在する還元性環境であるため、酸化されたシステイン残基から形成されるジスルフィド結合を含むタンパク質は細胞質内で生成されない。
膜結合リボソーム
リボソームが、どれかのオルガネラで必要なタンパク質を合成し始めると、そのタンパク質を作っているリボソームが「膜結合型」になることがある。真核細胞では、小胞体(ER)の粗面小胞体と呼ばれる領域でこの現象が起こる。新たに生成されたポリペプチド鎖は、ベクター生成中のリボソームから小胞体に直接挿入され、分泌経路を経て目的地に送り届けられる。膜結合リボソームは通常、細胞膜(原形質膜)内で使用されるタンパク質や、エキソサイトーシスにより細胞外に排出されるタンパク質を生成する[65]。
生合成
原核細胞では、リボソームは細胞質内で合成され、複数のリボソーム遺伝子オペロンが転写されることで作られる。真核生物では、このプロセスは細胞質および細胞核内の核小体の両方で行われる。この組立過程では、4つのrRNAの合成とプロセシング、およびそれらのrRNAとリボソームタンパク質の組み立てに200以上のタンパク質が協調して機能することが必要である[66]。
起源
リボソームはRNAワールドで誕生し、自己複製する複合体として出現し、その後、アミノ酸が出現したときにタンパク質を合成する能力を進化させたのではないかと考えられている[67]。古代のリボソームはすべてrRNAで構成され、ペプチド結合を合成する能力を発達させたのではないかとする研究もある[68][69][70][71][72]。さらに、古代のリボソームは自己複製複合体であったことを示唆する強い証拠があり、リボソーム内のrRNAは、リボソームの自己複製に必要なtRNAやタンパク質をコードし、情報伝達・構造・触媒の目的を持っていた可能性がある[73]。自己複製RNAを持ち、DNAを持たない仮説上の細胞生物は、リボサイト(ribocyte)またはリボセル(ribocells)と呼ばれる[74][75]。
原始的な条件下でRNAワールドにアミノ酸が徐々に出現すると[76][77]、アミノ酸との相互作用によって、触媒となるRNA分子の機能範囲と効率がそれぞれ向上する[67]。したがって、リボソームの形態が古代の自己複製機械から現在の翻訳機械へと進化した原動力は、リボソームの自己複製機構にタンパク質を組み込んで自己複製能力を高めることを可能にする選択圧である可能性がある[73][78][79]。
リボソームの不均一性
同じ真核細胞内でも細胞質リボソームとミトコンドリアリボソームが存在することで明示されているように、リボソームは、生物種間だけでなく同一細胞内でも組成が不均質である。一部の研究者は、哺乳動物のリボソームタンパク質の組成の不均一性が遺伝子調節に重要であることを示唆している。これは特殊化リボソーム仮説(specialized ribosome hypothesis)である[80][81]。しかし、この仮説は論争の的であり、研究が進行中のトピックである[82][83]。
リボソーム組成の不均一性は、Vince Mauroとジェラルド・エデルマンによって、タンパク質合成の翻訳制御に関与することが初めて提案された[84]。彼らは、リボソームの制御機能を説明するために、リボソームフィルター仮説を提唱した。その結果、異なる細胞集団に特異的な特殊化リボソームが、遺伝子の翻訳方法に影響を与える可能性が示唆された[85]。いくつかのリボソームタンパク質は、組み立てられた複合体から細胞質のコピーと差し替えられ[86]、まったく新しいリボソームを合成しなくても、生体内のリボソームの構造を変更できることが示唆された。
リボソームタンパク質には、細胞の生存に限りなく重要なものもあれば、そうでないものもある。出芽酵母では、14/78のリボソームタンパク質が成長に必須ではないが、ヒトでは、これらは研究対象の細胞によって異なる[87]。その他の不均一性には、アセチル化、メチル化、およびリン酸化などのリボソームタンパク質の翻訳後修飾が含まれる[88]。シロイヌナズナ(Arabidopsis)[89][90][91][92]やウイルスの配列内リボソーム進入部位(IRES)は、組成的に異なるリボソームによって翻訳を媒介する可能性がある。たとえば、酵母や哺乳動物細胞のリボソームタンパク質eS25を持たない40Sリボソームサブユニットは、CrPV IGR IRESを動員することができない[93]。
リボソームRNAの修飾の不均一性は、構造維持や機能において重要な役割を果たしており、ほとんどのmRNA修飾は高度に保存された領域に見られる[94][95]。最も一般的なrRNA修飾はリボースのシュードウリジル化と2'-O-メチル化である[96]。
関連項目
脚注
- ^ Konikkat S (February 2016). Dynamic Remodeling Events Drive the Removal of the ITS2 Spacer Sequence During Assembly of 60S Ribosomal Subunits in S. cerevisiae (Ph.D. thesis). Carnegie Mellon University. 2017年8月3日時点のオリジナルよりアーカイブ。
- ^ (ドイツ語) Allgemeine und Molekulare Botanik. Stuttgart: Georg Thieme Verlag. (2008). pp. 532. ISBN 9783131527912
- ^ “Functions of ribosomal proteins in assembly of eukaryotic ribosomes in vivo”. Annual Review of Biochemistry 84: 93–129. (2015). doi:10.1146/annurev-biochem-060614-033917. PMC 4772166. PMID 25706898.
- ^ “Scitable by nature translation / RNA translation”. 2022年11月28日閲覧。
- ^ a b c “The Peptidyl Transferase Center: a Window to the Past”. Microbiol Mol Biol Rev 85 (4): e0010421. (November 2021). doi:10.1128/MMBR.00104-21. PMC 8579967. PMID 34756086.
- ^ “Evolution of the mitochondrial protein synthetic machinery”. Bio Systems 21 (1): 51–68. (1987). doi:10.1016/0303-2647(87)90006-2. PMID 2446672.
- ^ “Ribosomes”. 2009年3月20日時点のオリジナルよりアーカイブ。2011年4月28日閲覧。
- ^ “A small particulate component of the cytoplasm”. The Journal of Biophysical and Biochemical Cytology 1 (1): 59–68. (January 1955). doi:10.1083/jcb.1.1.59. PMC 2223592. PMID 14381428.
- ^ “Introduction”. Microsomal Particles and Protein Synthesis. New York: Pergamon Press, Inc.. (1958)
- ^ “The Nobel Prize in Physiology or Medicine 1974”. Nobelprize.org. The Nobel Foundation. 26 January 2013時点のオリジナルよりアーカイブ。10 December 2012閲覧。
- ^ “2009 Nobel Prize in Chemistry”. The Nobel Foundation. 28 April 2012時点のオリジナルよりアーカイブ。10 December 2012閲覧。
- ^ “Molecular characterization of ribonucleic acid from Escherichia coli ribosomes”. Journal of Molecular Biology 2 (2): 83–91. (1960). doi:10.1016/s0022-2836(60)80029-0.
- ^ “The structure and function of the eukaryotic ribosome”. Cold Spring Harbor Perspectives in Biology 4 (5): a011536. (May 2012). doi:10.1101/cshperspect.a011536. PMC 3331703. PMID 22550233.
- ^ “The structural basis of ribosome activity in peptide bond synthesis”. Science 289 (5481): 920–30. (August 2000). Bibcode: 2000Sci...289..920N. doi:10.1126/science.289.5481.920. PMID 10937990. オリジナルの2020-11-30時点におけるアーカイブ。.
- ^ a b “Structure of the 30S ribosomal subunit”. Nature 407 (6802): 327–39. (September 2000). Bibcode: 2000Natur.407..327W. doi:10.1038/35030006. PMID 11014182.
- ^ a b c d e f g “Membrane-bound Ribosomes Define the Rough ER”. Molecular Biology of the Cell (4th ed.). New York: Garland Science. (2002). pp. 342. ISBN 978-0-8153-4072-0
- ^ a b Biochemistry (4th ed.). Cengage Learning Services. (2009). ISBN 978-0-495-11464-2
- ^ “The site of reaction on ribosomal protein L27 with an affinity label derivative of tRNA Met f”. FEBS Letters 63 (2): 283–6. (April 1976). doi:10.1016/0014-5793(76)80112-3. PMID 770196.
- ^ “Proteins at the tRNA binding sites of Escherichia coli ribosomes”. Proceedings of the National Academy of Sciences of the United States of America 71 (1): 230–4. (January 1974). Bibcode: 1974PNAS...71..230C. doi:10.1073/pnas.71.1.230. PMC 387971. PMID 4589893.
- ^ “30S ribosomal proteins associated with the 3'-terminus of 16S RNA”. FEBS Letters 58 (1): 281–4. (October 1975). doi:10.1016/0014-5793(75)80279-1. PMID 1225593.
- ^ “Archaeal Ribosomes”. Encyclopedia of Life Science. New York: Facts On File. (2009). pp. 1–5. doi:10.1002/9780470015902.a0000293.pub3. ISBN 9780470015902
- ^ “Net Charges of the Ribosomal Proteins of the S10 and spc Clusters of Halophiles Are Inversely Related to the Degree of Halotolerance”. Microbiol. Spectrum 9 (3): e0178221. (November 2021). doi:10.1128/spectrum.01782-21. PMC 8672879. PMID 34908470.
- ^ “Many nonuniversal archaeal ribosomal proteins are found in conserved gene clusters”. Archaea 2 (4): 241–51. (28 April 2009). doi:10.1155/2009/971494. PMC 2686390. PMID 19478915.
- ^ a b c “The structure of the eukaryotic ribosome at 3.0 Å resolution”. Science 334 (6062): 1524–9. (December 2011). Bibcode: 2011Sci...334.1524B. doi:10.1126/science.1212642. PMID 22096102.
- ^ a b “Crystal structure of the eukaryotic 40S ribosomal subunit in complex with initiation factor 1”. Science 331 (6018): 730–6. (February 2011). Bibcode: 2011Sci...331..730R. doi:10.1126/science.1198308. hdl:20.500.11850/153130. PMID 21205638.
- ^ a b “Crystal structure of the eukaryotic 60S ribosomal subunit in complex with initiation factor 6”. Science 334 (6058): 941–8. (November 2011). Bibcode: 2011Sci...334..941K. doi:10.1126/science.1211204. PMID 22052974.
- ^ “Identification of proteins at the peptidyl-tRNA binding site of rat liver ribosomes”. Molecular & General Genetics 184 (3): 551–6. (1977). doi:10.1007/BF00431588. PMID 6950200.
- ^ “Structural aspects of mitochondrial translational apparatus”. Current Opinion in Structural Biology 22 (6): 797–803. (December 2012). doi:10.1016/j.sbi.2012.08.003. PMC 3513651. PMID 22959417.
- ^ “Structure of a mitochondrial ribosome with minimal RNA”. Proceedings of the National Academy of Sciences of the United States of America 106 (24): 9637–42. (June 2009). Bibcode: 2009PNAS..106.9637S. doi:10.1073/pnas.0901631106. PMC 2700991. PMID 19497863.
- ^ “Small is big in Arabidopsis mitochondrial ribosome”. Nature Plants 5 (1): 106–117. (January 2019). doi:10.1038/s41477-018-0339-y. PMID 30626926.
- ^ “Going, going, not quite gone: nucleomorphs as a case study in nuclear genome reduction”. The Journal of Heredity 100 (5): 582–90. (2009). doi:10.1093/jhered/esp055. PMID 19617523.
- ^ “Specialized Internal Structures of Prokaryotes | Boundless Microbiology”. courses.lumenlearning.com. 2021年9月24日閲覧。
- ^ “Basis for prokaryotic specificity of action of aminoglycoside antibiotics”. The EMBO Journal 18 (11): 3133–8. (June 1999). doi:10.1093/emboj/18.11.3133. PMC 1171394. PMID 10357824.
- ^ “The general occurrence of 55 S ribosomes in mammalian liver mitochondria”. The Journal of Biological Chemistry 246 (10): 3409–17. (May 1971). doi:10.1016/S0021-9258(18)62239-2. PMID 4930061.
- ^ “Chloramphenicol-lnduced Bone Marrow Suppression” (英語). JAMA 213 (7): 1183–1184. (1970-08-17). doi:10.1001/jama.1970.03170330063011. ISSN 0098-7484. PMID 5468266.
- ^ “Transformation of chloroplast ribosomal RNA genes in Chlamydomonas: molecular and genetic characterization of integration events”. Genetics 126 (4): 875–88. (December 1990). doi:10.1093/genetics/126.4.875. PMC 1204285. PMID 1981764.
- ^ “Supersized ribosomal RNA expansion segments in Asgard archaea”. Genome Biology and Evolution 12 (10): 1694–1710. (August 2020). doi:10.1093/gbe/evaa170. PMC 7594248. PMID 32785681.
- ^ a b Bruce Alberts, Dennis Bray, Karen Hopkin他 『Essential 細胞生物学(原書第2版)』 中村桂子・松原謙一 監訳、南江堂、2005年9月、p.251-252
- ^ a b “The complete atomic structure of the large ribosomal subunit at 2.4 A resolution”. Science 289 (5481): 905–20. (August 2000). Bibcode: 2000Sci...289..905B. doi:10.1126/science.289.5481.905. PMID 10937989.
- ^ “Structure of functionally activated small ribosomal subunit at 3.3 angstroms resolution”. Cell 102 (5): 615–23. (September 2000). doi:10.1016/S0092-8674(00)00084-2. PMID 11007480.
- ^ “Crystal structure of the ribosome at 5.5 A resolution”. Science 292 (5518): 883–96. (May 2001). Bibcode: 2001Sci...292..883Y. doi:10.1126/science.1060089. PMID 11283358.
- ^ “Structures of the bacterial ribosome at 3.5 A resolution”. Science 310 (5749): 827–34. (November 2005). Bibcode: 2005Sci...310..827S. doi:10.1126/science.1117230. PMID 16272117.
- ^ “Structure of the E. coli protein-conducting channel bound to a translating ribosome”. Nature 438 (7066): 318–24. (November 2005). Bibcode: 2005Natur.438..318M. doi:10.1038/nature04133. PMC 1351281. PMID 16292303.
- ^ “Structure of the 70S ribosome complexed with mRNA and tRNA”. Science 313 (5795): 1935–42. (September 2006). Bibcode: 2006Sci...313.1935S. doi:10.1126/science.1131127. PMID 16959973.
- ^ “Crystal structure of a 70S ribosome-tRNA complex reveals functional interactions and rearrangements”. Cell 126 (6): 1065–77. (September 2006). doi:10.1016/j.cell.2006.08.032. PMID 16962654.
- ^ “Structural basis for messenger RNA movement on the ribosome”. Nature 444 (7117): 391–4. (November 2006). Bibcode: 2006Natur.444..391Y. doi:10.1038/nature05281. PMID 17051149.
- ^ a b “Specialized Internal Structures of Prokaryotes”. courses.lumenlearning.com. 2018年9月27日閲覧。
- ^ Lafontaine, D.; Tollervey, D. (2001). “The function and synthesis of ribosomes”. Nat Rev Mol Cell Biol 2 (7): 514–520. doi:10.1038/35080045. hdl:1842/729. PMID 11433365.
- ^ James D. Watson, T. A. Baker, S. P. Bell他 『ワトソン 遺伝子の分子生物学【第5版】』 中村桂子 監訳、東京電機大学出版局、2006年3月、p.423-430
- ^ “The ribosome as an optimal decoder: A lesson in molecular recognition”. Cell 153 (2): 471–479. (April 2013). Bibcode: 2013APS..MARY46006T. doi:10.1016/j.cell.2013.03.032. PMID 23582332.
- ^ “Escherichia coli”. The Journal of Biological Chemistry 292 (36): 15134–15142. (September 2017). doi:10.1074/jbc.M117.785238. PMC 5592688. PMID 28743745.
- ^ “Purine bases at position 37 of tRNA stabilize codon-anticodon interaction in the ribosomal A site by stacking and Mg2+-dependent interactions”. RNA 10 (1): 90–101. (January 2004). doi:10.1261/rna.5142404. PMC 1370521. PMID 14681588.
- ^ “How ribosomes make peptide bonds”. Trends in Biochemical Sciences 32 (1): 20–26. (January 2007). doi:10.1016/j.tibs.2006.11.007. PMID 17157507.
- ^ Cech, T.R. (August 2000). “Structural biology. The ribosome is a ribozyme”. Science 289 (5481): 878–879. doi:10.1126/science.289.5481.878. PMID 10960319.
- ^ “Protein folding activity of the ribosome (PFAR) – a target for antiprion compounds”. Viruses 6 (10): 3907–3924. (October 2014). doi:10.3390/v6103907. PMC 4213570. PMID 25341659.
- ^ “Cotranslational protein folding”. The Journal of Biological Chemistry 272 (52): 32715–32718. (December 1997). doi:10.1074/jbc.272.52.32715. PMID 9407040.
- ^ “Intermediates in protein folding reactions and the mechanism of protein folding”. Annual Review of Biochemistry 44 (1): 453–475. (June 1975). doi:10.1146/annurev.bi.44.070175.002321. PMID 1094916.
- ^ “Role of the ribosome in protein folding”. Biotechnology Journal 3 (8): 999–1009. (August 2008). doi:10.1002/biot.200800098. PMID 18702035.
- ^ “Protein knotting by active threading of nascent polypeptide chain exiting from the ribosome exit channel”. The Journal of Physical Chemistry B 122 (49): 11616–11625. (December 2018). doi:10.1021/acs.jpcb.8b07634. PMID 30198720.
- ^ “A ribosome-bound quality control complex triggers degradation of nascent peptides and signals translation stress”. Cell 151 (5): 1042–1054. (November 2012). doi:10.1016/j.cell.2012.10.044. PMC 3534965. PMID 23178123.
- ^ “Cdc48-associated complex bound to 60S particles is required for the clearance of aberrant translation products”. Proceedings of the National Academy of Sciences of the United States of America 110 (13): 5046–5051. (March 2013). Bibcode: 2013PNAS..110.5046D. doi:10.1073/pnas.1221724110. PMC 3612664. PMID 23479637.
- ^ “Protein synthesis. Rqc2p and 60S ribosomal subunits mediate mRNA-independent elongation of nascent chains”. Science 347 (6217): 75–78. (January 2015). Bibcode: 2015Sci...347...75S. doi:10.1126/science.1259724. PMC 4451101. PMID 25554787.
- ^ Keeley, J.; Gutnikoff, R. (2 January 2015). "Ribosome studies turn up new mechanism of protein synthesis" (Press release). Howard Hughes Medical Institute. 2015年1月12日時点のオリジナルよりアーカイブ。2015年1月16日閲覧。
- ^ Kobayashi, Kan (2018). “シグナル認識粒子とシグナル認識粒子受容体に よる新生タンパク質の小胞体膜ターゲティング 機構の構造基盤 (Structural Basis for the Nascent Protein Targeting to Endoplasmic Reticulum by SRP and SR)” (英語). 生物物理 58 (5): 255–257. doi:10.2142/biophys.58.255. ISSN 0582-4052.
- ^ “Membrane-bound Ribosomes Define the Rough ER”. Molecular Biology of the Cell (4th ed.). New York: Garland Science. (2002). ISBN 978-0-8153-4072-0
- ^ Kressler, Dieter; Hurt, Ed; Babler, Jochen (2009). “Driving ribosome assembly”. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1803 (6): 673–683. doi:10.1016/j.bbamcr.2009.10.009. PMID 19879902.
- ^ a b “Evolution of protein synthesis from an RNA world”. Cold Spring Harbor Perspectives in Biology 4 (4): a003681. (April 2012). doi:10.1101/cshperspect.a003681. PMC 3312679. PMID 20610545.
- ^ Mutant studies on the prokaryotic ribosome.. New York: Springer-Verlag. (1986)
- ^ “Unusual resistance of peptidyl transferase to protein extraction procedures”. Science 256 (5062): 1416–9. (June 1992). Bibcode: 1992Sci...256.1416N. doi:10.1126/science.1604315. PMID 1604315.
- ^ “Structure and function of ribosomes and their molecular components”. Cold Spring Harbor Symposia on Quantitative Biology 34: 49–61. (1969). doi:10.1101/sqb.1969.034.01.009. PMID 4909519.
- ^ “A vestige of a prebiotic bonding machine is functioning within the contemporary ribosome”. Phil. Trans. R. Soc. B 366 (1580): 2972–8. (Oct 2011). doi:10.1098/rstb.2011.0146. PMC 3158926. PMID 21930590.
- ^ “Origin of life: protoribosome forms peptide bonds and links RNA and protein dominated worlds”. Nucleic Acids Res 50 (4): 1815–1828. (Feb 2022). doi:10.1093/nar/gkac052. PMC 8886871. PMID 35137169.
- ^ a b “The ribosome as a missing link in the evolution of life”. Journal of Theoretical Biology 367: 130–158. (February 2015). doi:10.1016/j.jtbi.2014.11.025. PMID 25500179.
- ^ “Primordial genetics: phenotype of the ribocyte”. Annual Review of Genetics 36: 125–51. (2002). doi:10.1146/annurev.genet.36.031902.105056. PMID 12429689.
- ^ “The Origin of Virions and Virocells: The Escape Hypothesis Revisited”. Viruses: Essential Agents of Life. (2012). pp. 43–60. doi:10.1007/978-94-007-4899-6_3. ISBN 978-94-007-4898-9
- ^ “The coevolutionary roots of biochemistry and cellular organization challenge the RNA world paradigm”. Journal of Molecular Microbiology and Biotechnology 23 (1–2): 152–77. (2013). doi:10.1159/000346551. PMID 23615203.
- ^ “Genetics first or metabolism first? The formamide clue”. Chemical Society Reviews 41 (16): 5526–65. (August 2012). doi:10.1039/c2cs35066a. hdl:11573/494138. PMID 22684046.
- ^ “Origin and Evolution of the Ribosome”. Cold Spring Harb Perspect Biol 2 (9): a003483. (September 2010). doi:10.1101/cshperspect.a003483. PMC 2926754. PMID 20534711.
- ^ “Origins and early evolution of the ribosome”. Evolution of the Protein Synthesis Machinery and Its Regulation. Switzerland: Springer, Cham. (2016). pp. 31–60. doi:10.1007/978-3-319-39468-8. ISBN 978-3-319-39468-8
- ^ “Heterogeneous Ribosomes Preferentially Translate Distinct Subpools of mRNAs Genome-wide”. Molecular Cell (Elsevier BV) 67 (1): 71–83.e7. (July 2017). doi:10.1016/j.molcel.2017.05.021. PMC 5548184. PMID 28625553.
- ^ “Specialized ribosomes: a new frontier in gene regulation and organismal biology”. Nature Reviews. Molecular Cell Biology (Springer Science and Business Media LLC) 13 (6): 355–369. (May 2012). doi:10.1038/nrm3359. PMC 4039366. PMID 22617470.
- ^ “Does functional specialization of ribosomes really exist?”. RNA (Cold Spring Harbor Laboratory) 25 (5): 521–538. (May 2019). doi:10.1261/rna.069823.118. PMC 6467006. PMID 30733326.
- ^ “Ribosomopathies: Old Concepts, New Controversies”. Trends in Genetics (Elsevier BV) 35 (10): 754–767. (October 2019). doi:10.1016/j.tig.2019.07.004. PMC 6852887. PMID 31376929.
- ^ “The ribosome filter hypothesis”. Proceedings of the National Academy of Sciences of the United States of America 99 (19): 12031–6. (September 2002). Bibcode: 2002PNAS...9912031M. doi:10.1073/pnas.192442499. PMC 129393. PMID 12221294.
- ^ “Specialized ribosomes: a new frontier in gene regulation and organismal biology”. Nature Reviews. Molecular Cell Biology 13 (6): 355–69. (May 2012). doi:10.1038/nrm3359. PMC 4039366. PMID 22617470.
- ^ “Mechanisms of In Vivo Ribosome Maintenance Change in Response to Nutrient Signals”. Molecular & Cellular Proteomics 16 (2): 243–254. (February 2017). doi:10.1074/mcp.M116.063255. PMC 5294211. PMID 27932527.
- ^ “Ribosome deficiency protects against ER stress in Saccharomyces cerevisiae”. Genetics (Genetics Society of America) 191 (1): 107–118. (May 2012). doi:10.1534/genetics.111.136549. PMC 3338253. PMID 22377630.
- ^ “Direct mass spectrometric analysis of intact proteins of the yeast large ribosomal subunit using capillary LC/FTICR”. Proceedings of the National Academy of Sciences of the United States of America 99 (9): 5942–5947. (April 2002). Bibcode: 2002PNAS...99.5942L. doi:10.1073/pnas.082119899. PMC 122881. PMID 11983894.
- ^ “Analysis of the Arabidopsis cytosolic ribosome proteome provides detailed insights into its components and their post-translational modification”. Molecular & Cellular Proteomics 7 (2): 347–369. (February 2008). doi:10.1074/mcp.m700052-mcp200. PMID 17934214.
- ^ “Characterization and analysis of posttranslational modifications of the human large cytoplasmic ribosomal subunit proteins by mass spectrometry and Edman sequencing”. Journal of Protein Chemistry 22 (3): 249–258. (April 2003). doi:10.1023/a:1025068419698. PMID 12962325.
- ^ “Mass spectrometric analysis of the human 40S ribosomal subunit: native and HCV IRES-bound complexes”. Protein Science 14 (6): 1438–1446. (June 2005). doi:10.1110/ps.041293005. PMC 2253395. PMID 15883184.
- ^ “O-GlcNAc cycling enzymes associate with the translational machinery and modify core ribosomal proteins”. Molecular Biology of the Cell 21 (12): 1922–1936. (June 2010). doi:10.1091/mbc.e09-11-0941. PMC 2883937. PMID 20410138.
- ^ “RPS25 is essential for translation initiation by the Dicistroviridae and hepatitis C viral IRESs”. Genes & Development 23 (23): 2753–2764. (December 2009). doi:10.1101/gad.1832209. PMC 2788332. PMID 19952110.
- ^ “rRNA modifications and ribosome function”. Trends in Biochemical Sciences 27 (7): 344–51. (July 2002). doi:10.1016/s0968-0004(02)02109-6. PMID 12114023.
- ^ “Visualization of chemical modifications in the human 80S ribosome structure”. Nature 551 (7681): 472–477. (November 2017). Bibcode: 2017Natur.551..472N. doi:10.1038/nature24482. PMID 29143818.
- ^ “Specialized ribosomes and the control of translation”. Biochemical Society Transactions 46 (4): 855–869. (August 2018). doi:10.1042/BST20160426. PMID 29986937.
外部リンク
- リボソームとは? - 国立遺伝学研究所マルチメディア資料館
- 蛋白質構造データバンク 今月の分子10:リボソーム(Ribosome)
- 蛋白質構造データバンク 今月の分子121:70Sリボソーム(70S Ribosomes)
- 蛋白質構造データバンク 今月の分子81:伸長因子(Elongation Factors)
- リボソームの役割, Gwen V. Childs, copied here
- Ribosome in Proteopedia—The free, collaborative 3D encyclopedia of proteins & other molecules
- Ribosomal proteins families in ExPASy Archived 2011-04-30 at the Wayback Machine.
- リボソームの3次元電子顕微鏡構造 EM Data Bank (EMDB)
