立体配座選択
立体配座選択(りったいはいざせんたく、英: conformational selection)または立体構造校正(英: conformational proofreading)は、分子認識系の一般的な機構であり、分子認識系とその標的との間に構造的不一致またはエネルギー的障壁を導入することによって、認識の特異性と質を向上させる[1][2][3][4][5][6]。立体配座選択はエネルギーを消費しないので、どのような分子認識系でも利用することができる。立体配座選択は、認識系が多くの類似した競合相手から適切な標的を選択しなければならない場合に特に有効である。
正しい結合と誤った結合のバランス
[編集]分子認識は、ノイズが多く混雑した生物環境下で行われるので、認識系はしばしば、さまざまな類似した競合相手の中から標的を選択するという課題に対処しなければならない。たとえば、リボソームは、構造的に類似した多くのtRNAの中から、mRNAのコドンと一致する正しいtRNAを選択しなくてならない。もし、認識系と標的が鍵と鍵穴のように完全に一致すれば、結合時に変形する必要がないため、結合確率は高くなる。同時に、認識系は類似した構造を持つ競合物質に高い確率で結合してしまう可能性もある。認識系(錠前)と鍵の間に「構造的な不一致」を導入すると、正しい標的に結合する確率は下がるが、類似した誤った標的への結合確率はさらに下がるため、特異性が向上する。しかし、変形を大きくしすぎると、正しい標的への結合確率が著しく低下する。したがって、正しい結合確率の最大化と誤った結合確率の最小化との最適なバランスは、認識系がわずかに「オフターゲット(標的から外れた)」したときに達成される。このことから、分子認識過程における構造的変化が(たとえば誘導適合機構[7])、認識の特異性を高めるために有利であることが示唆される。

上図: 相同な(正しい)DNA配列と非相同な(誤った)DNA配列への結合確率は、変形エネルギー障壁とともに減少する。誤った結合確率は、正しい結合確率よりも先に減少する。
下図: 結果として、その差である Fitness = Prob(Correct) - Prob(Wrong) は、変形エネルギーが0でないときに最大となる。この障壁は、正しい結合確率をほぼ同じに保ちながら、結合確率を著しく低下させるという意味で最適である。RecA誘導組換えを生化学的に測定した結果、観察された変形はほぼ最適であることが示唆された[3][4]。
相同組換えにおける相同性探索の利用
[編集]相同組換えの機構は、「立体配座選択」の体系を用いて、類似したDNA配列を識別している[3][4]。相同組換えは、相同なDNA分子間で遺伝物質の交換を促進する。この重要なプロセスでは、膨大な数の異なる配列の中から、特定の相同なDNA配列を検出する必要がある。大腸菌ではRecAが、他の生物ではそのスーパーファミリーのメンバーがこの検出を担っている。RecAは、初めに一本鎖DNAに沿って結合し、次にこのタンパク質-DNAフィラメントが二本鎖DNAに沿って相同性を探索する。RecA-DNAフィラメントでは、塩基間の距離が露出した二本鎖の3.4 Åに比べて大幅に広がっている(平均50%[8])。相同性を調べるためには、二本鎖DNAを同じ大きさだけ引き伸ばさないとならないので、探索には大きなエネルギー的障壁が存在する。DNA認識過程を信号検出問題として定式化することで、実験的に観測されたRecA誘導DNA変形と結合エネルギーが、最適な配列検出を確実にするように微調整されていることが明らかになった。その変化量は、相同なDNA配列への結合ではわずかに減少し、誤った配列への結合では大きく減少するようなものである。これはまさに立体配座選択の機構である。
相同組換えによる立体配座選択の実験的証拠
[編集]シース・デッカー(デルフト大学)のグループは、磁気ピンセットと光ピンセットを組み合わせて、相同性探索に関わる相互作用を直接的に探索した[9]。その結果、相同性探索や認識にはヘリックス(らせん)を開く必要があり、そのためにはDNAを巻き戻すことで加速できることを発見した。これはまさに、立体配座選択モデルで予測されるエネルギー障壁である。そのデータは、DNA結合部位間の距離によって探索プロセスの忠実度(正確さ)が支配されるという、相同性認識の物理的な図式を示している。著者らは、この測定結果の解釈について、『RecAフィラメントではなくdsDNAが、活発な認識探索の主体であり、「立体配座選択」のスキームに似ている。dsDNAと標的の結合状態と非結合状態の間には、大きな構造的差異が存在する。標的の結合状態には、上述のようにエネルギー的に不利な中間状態を経て到達する。この構造的な不一致が、認識反応の選択性を向上させる。』と結論付けた。言い換えれば、彼らはエネルギー的障壁を特定し、二本鎖DNAはこの障壁を通過しなければならず、本当に能動的な関与者であることを明らかにした。
tRNA解読のためのリボソームによる利用
[編集]リボソームは複雑な分子機械であり、翻訳過程でタンパク質を合成するために、mRNAのコドンをそれに対応するtRNAの対を解読する必要がある。この解読には、多くの類似した競合相手の中から正しいtRNAを正確かつ迅速に選択する必要があり、適合度を決定する主要な要因となっている。ほとんどの結合イベントは不一致のtRNA(非同族tRNA)によるもので、リボソームはできるだけ早くそれらを拒否して結合部位を解放する必要があることを留意しておく必要がある。同時にリボソームはタンパク質合成プロセスを進めるために、適合するtRNAの結合を十分な時間維持する必要がある。tRNA解読は重要であるにもかかわらず、現代のリボソームが特に解読時に行う「大きな構造変化」が、解読器としての課題に適応した結果なのか、それとも他の制約による結果なのかは、最近まで不明であった。最近の研究では[5]、競合するtRNA基質を最適に識別し、tRNAの解読を最適化するエネルギーランドスケープ(エネルギー地形)を導き出した。最適なランドスケープは「対称的」である(図の説明を参照)。この研究で、原核生物のリボソームで測定されたランドスケープが、実際に対称的であることが示された。このモデルは、解読中のリボソームとtRNAの構造変化が、このような最適なtRNA解読器を得るための手段であることを示唆している。このように、相同組換えとtRNA解読の両方が「立体配座選択」を利用していることから、この機構は分子認識系で広く利われている汎用的な機構であることが示唆される。
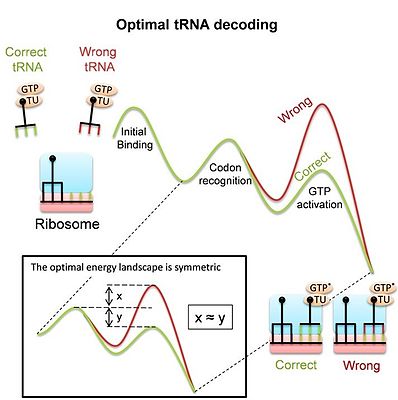
全体図: 曲線は、実験によって示唆されたコドン認識の自由エネルギー・ランドスケープ(地形)を示す。コドンの同一性に敏感な段階では、正しいtRNA(緑)と誤ったtRNA(赤)で経路が分かれる。多段階の動力学は次のようになる。
* 最初の結合とコドン認識:伸長因子(EF-Tu)とアミノアシルtRNAの複合体がリボソームに結合する。コドンはアンチコドンとの対合、さらにリボソームの「解読センター(decoding center)」との付加的な相互作用によって認識される。その結果、正しい(同族)tRNAは、誤った(同族)tRNAよりも安定である。
* GTPの活性化と加水分解:コドン認識は、リボソームとtRNAは全体的な構造変化を引き起こす。この構造変化は、同族tRNAと非同族tRNAで異なり、EF-TuによるGTPの活性化と加水分解に影響を及ぼす。立体配座選択モデルではこれらの構造変化をtRNAの認識力を向上させるための手段として説明している。
挿入図: 対称的な適応ランドスケープとは、正しいエネルギーランドスケープと誤ったエネルギーランドスケープの間で順方向速度と逆方向速度の比率が逆転していることを意味する[5]。
その他の生物系
[編集]ヒトのDNA紫外線損傷修復
[編集]最近の研究から、ヒトのDNA修復機構において立体配座選択が起こることが明らかになった[10]。この研究では、ヌクレオチド除去修復(NER)の初期段階において、DNA修復タンパク質が、紫外線によってヒトゲノムに生じた損傷をどのように走査しているかという疑問に焦点を当てた。詳細な単一分子計測により、ヒトの紫外線損傷DNA結合タンパク質(UV-DDB)がどのように3次元的な探索を行うのかが明らかになった。著者の発見は次の通りである。『UV-DDBは、損傷部位に長寿命で非運動性のUV-DDB二量体 (DDB1-DDB2)2 を形成する前に、DNA上の部位を個別的な段階で調査する。損傷していないDNAと損傷したDNAの両方における一時的な結合分子の解離速度を分析したところ、3桁を超える複数の滞留時間があることがわかった。(中略)これらの中間状態は、安定した損傷検出への軌道上にある個別のUV-DDB立体配座異性体を表していると考えられる』。詳細な動力学的測定から、著者らは『UV-DDBは複数の中間体を介した立体配座選択機構を利用して損傷を認識している。』と結論付けた。
その他の認識法
[編集]動力学的校正との関係
[編集]動力学的校正(kinetic proofreading)の方法では[11][12]、正しいあるいは誤った複合体の形成中に、「時間的遅延」(不可逆的な中間段階と等価)が導入される。この時間的な遅れにより、両方の複合体の生成速度が低下するが、平衡限界を超えて忠実度が向上する。この方法の不可逆性には、エネルギー源を必要とする。動力学的校正における時間遅れは、立体配座選択における場所的相違と類似している。しかし、立体配座選択はエネルギーを消費しない平衡法となりうる。
脚注
[編集]- ^ Savir Y & Tlusty T (2007). Scalas, Enrico. ed. “Conformational Proofreading: The Impact of Conformational Changes on the Specificity of Molecular Recognition”. PLOS ONE 2 (5): e468. Bibcode: 2007PLoSO...2..468S. doi:10.1371/journal.pone.0000468. PMC 1868595. PMID 17520027.
- ^ Savir Y, Tlusty T (2008). “Optimal Design of a Molecular Recognizer : Molecular Recognition as a Bayesian Signal Detection Problem”. IEEE J Sel Topics Signal Process 2 (3): 390–399. arXiv:1007.4527. Bibcode: 2008ISTSP...2..390S. doi:10.1109/JSTSP.2008.923859.
- ^ a b c Savir Y, Tlusty T (2010). “RecA-mediated homology search as a nearly optimal signal detection system”. Molecular Cell 40 (3): 388–96. arXiv:1011.4382. doi:10.1016/j.molcel.2010.10.020. PMID 21070965.
- ^ a b c Rambo RP, Williams GJ, Tainer JA (2010). “Achieving Fidelity in Homologous Recombination Despite Extreme Complexity: Informed Decisions by Molecular Profiling”. Molecular Cell 40 (3): 347–48. doi:10.1016/j.molcel.2010.10.032. PMC 3003302. PMID 21070960.
- ^ a b c Savir, Yonatan; Tlusty, Tsvi (Apr 11, 2013). “The ribosome as an optimal decoder: a lesson in molecular recognition.”. Cell 153 (2): 471–9. Bibcode: 2013APS..MARY46006T. doi:10.1016/j.cell.2013.03.032. PMID 23582332.
- ^ Alon U (2008). “Journal Club”. Nature 453 (7196): 701. Bibcode: 2008Natur.453..701A. doi:10.1038/453701e.
- ^ Koshland, D. E. (1958). “Application of a Theory of Enzyme Specificity to Protein Synthesis”. Proc Natl Acad Sci U S A 44 (2): 98–104. Bibcode: 1958PNAS...44...98K. doi:10.1073/pnas.44.2.98. PMC 335371. PMID 16590179.
- ^ Chen Z, Yang H, Pavletich NP (2008). “Mechanism of homologous recombination from the RecA-ssDNA/dsDNA structures”. Nature 453 (7194): 489–4. Bibcode: 2008Natur.453..489C. doi:10.1038/nature06971. PMID 18497818.
- ^ De Vlaminck I, van Loenhout MT, Zweifel L, den Blanken J, Hooning K, Hage S, Kerssemakers J, Dekker C (2012). “Mechanism of Homology Recognition in DNA Recombination from Dual-Molecule Experiments”. Molecular Cell 46 (5): 616–624. doi:10.1016/j.molcel.2012.03.029. PMID 22560720.
- ^ Ghodke H, Wang H, Hsieh CL, Woldemeskel S, Watkins SC, Rapić-Otrin V, Van Houten B (May 6, 2014). “Single-molecule analysis reveals human UV-damaged DNA-binding protein (UV-DDB) dimerizes on DNA via multiple kinetic intermediates.”. Proc Natl Acad Sci U S A 111 (18): 1862–71. Bibcode: 2014PNAS..111E1862G. doi:10.1073/pnas.1323856111. PMC 4020048. PMID 24760829.
- ^ Hopfield JJ (1974). “Kinetic Proofreading: A New Mechanism for Reducing Errors in Biosynthetic Processes Requiring High Specificity”. Proc Natl Acad Sci U S A 71 (10): 4135–4139. Bibcode: 1974PNAS...71.4135H. doi:10.1073/pnas.71.10.4135. PMC 434344. PMID 4530290.
- ^ Ninio J (1975). “Kinetic amplification of enzyme discrimination Biochimie”. Biochimie 57 (5): 587–595. doi:10.1016/S0300-9084(75)80139-8. PMID 1182215.
