古倍数性

古倍数性[1][2](こばいすうせい、英語: paleopolyploidy)は、少なくとも数百万年前に起こったゲノム重複の結果、倍数性を示す現象である。倍数化により遺伝子の機能的重複が起こり、重複したゲノム中の遺伝子は急速に不活性化または消失する。古倍数体 (paleopolyploid) はほとんどの場合、進化の過程で複相化と呼ばれる過程によって倍数体としての性質を失い、現在では二倍体として扱われている。例えば、パン酵母 Saccharomyces cerevisiae[3] やシロイヌナズナ Arabidopsis thaliana[4]、ダイズ Glycine max[1][2]、そしておそらくヒト Homo sapiens[5][6][7]も古倍数体であることが知られている。
古倍数性は植物の系統で広く研究されており、ほぼすべての被子植物が進化の過程で少なくとも1回のゲノム重複を経験していることが分かっている。また、脊椎動物においては、四足動物を含む硬骨魚類の共通祖先でも、真骨類のステムの系統でも、非常に古い時代にゲノム重複が起こっている。小さなゲノムを持つパン酵母でさえ、進化の過程で倍数体を経験したとする証拠がある。
また、過去1700万年以内のような、より新しい時代における全ゲノム重複、全ゲノム3倍体化などの全ゲノム重複イベントを経た種に対して、mesopolyploid(中倍数体)という用語が用いられることもある[8]。
真核生物
[編集]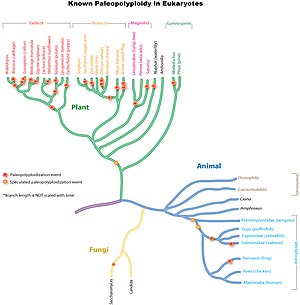
植物
[編集]古代のゲノム重複は、真核生物の系統に広く見られるが、特に植物でよく知られる。イネ科の共通祖先では、約7000万年前に全ゲノム重複を共有していたことが示唆されている[9]。より古い時代の単子葉植物の系統では、真正双子葉植物と別箇に1回以上の全ゲノム重複が起きていた[10]。
すべての現生被子植物の共通祖先で、1億6000万年前に倍数化イベントが起こったとする説もある[11]。その古倍数性イベントは、基部被子植物であるアンボレラ Amborella trichopoda のゲノムを配列決定することによって研究されている[12]。真正双子葉類は共通の全ゲノム3倍体(paleo-hexaploidy[注釈 1])を共有しており、これは単子葉植物と真正双子葉類が分岐後、バラ類とキク類の分岐前に起こったと推定されている[13][14][15]。多くの真正双子葉類では、さらに全ゲノム重複や3倍体化を経験している。例えば、初めて全ゲノムが解読された植物であるモデル植物のシロイヌナズナは、コア真正双子葉類が共有するゲノム重複以降にも、加えて少なくとも2回の全ゲノム重複を経験している[4]。
真菌
[編集]13 Mbp未満と小さなゲノムであるにもかかわらず古倍数性が実証されている例の1つはパン酵母で、Kluyveromyces lactis や K. marxianus などのクリベロミセス属酵母[16]から分岐した後に全ゲノム重複が起こったことが強く示唆されている[17]。
脊椎動物
[編集]植物に比べ、動物界では古倍数性は非常に稀で、主に両生類や硬骨魚類で確認されている。ヒトを含む脊椎動物には1つ以上の共通のゲノム重複があるとする研究もあるが[5][6][7]、重複があったとしてもかなり古い時代で[注釈 2]、まだ議論がある。
脊椎動物のゲノム中には2万個から3万個の遺伝子が含まれているが、その大部分は遺伝子重複によるパラログを含んでいる[18]。脊椎動物の系統では、全ゲノム重複が何度か起きたと考えられている[18]。そのうち、脊椎動物の進化の初期段階に起こった全ゲノム重複は「2R (two-round)」と呼ばれ、頭索動物や尾索動物と分岐した後、軟骨魚類の分岐以前に起こったことが確実だと考えられている[18]。1度目は円口類の分岐以前であることが確実だと分かっているが、2度目の全ゲノム重複は円口類の分岐以前なのか分岐後なのかは議論があった[18]。また、条鰭類のうち、真骨類の系統では、「3R」と呼ばれる3度目の全ゲノム重複が起きた[19]。
1970年、大野乾によって「脊椎動物では進化の初期段階で全ゲノムの重複が2回起こり、すべての脊椎動物は古倍数性起源である」とする2R仮説が提唱された[20]。2R仮説では、脊椎動物のゲノムは無脊椎動物のゲノムの4倍となるはずだと予測されていた。脊椎動物では4つの遺伝子ファミリーに((AB)(CD))木構造が存在しないという2R説への反論があったが、もし2つのゲノム重複が近接して起これば、このトポロジーにはならない[21]。ナメクジウオ Branchiostoma lanceolatum のゲノム配列は、全ゲノムの重複が2回行われ、その後、ほとんどの遺伝子で重複コピーが失われたという仮説を支持するものであった[22]。最近の研究ではウミヤツメ Petromyzon marinus の遺伝地図が作成され、脊椎動物の基部系統で1つの全ゲノム重複が起こり、その前後に脊索動物の進化上独立したいくつかのセグメント重複が起こったという仮説を強く支持する結果となった[23]。
この2回の全ゲノム重複は、Hox遺伝子のクラスターが4つに増えたことや[18]、脊椎動物の系統で複雑化した補体系の進化にも影響していると考えられている[24]。
進化における影響
[編集]- 表現型の進化
- 全ゲノム重複は、生物が新しい形質を獲得する速度と効率を高める可能性がある。しかし、この仮説を検証するために、硬骨魚類についてゲノムの重複がある初期に分岐した真骨類と重複がない初期に分岐した全骨類間で革新的進化に関わる進化速度を比較したところ、ほとんど差がないことが分かった[25]。
- 種分化
- 多くの倍数化現象は、適応形質の獲得や、2倍体との不和合性によって新しい種を生み出したと考えられている。スパルティナ属 Spartina[注釈 3] では、雑種化と倍数化の両方により高度に侵略的な種スパルティナ・アングリカ S. anglica を進化させたことでよく知られている[26]。19世紀初頭に6倍体のヒガタアシ S. alterniflora がアメリカ東海岸からイギリス南部とフランス西部に導入され、在来の6倍体であるスパルティナ・マリティマ[27] S. maritima と交雑し、6倍体の雑種 S. × townsendii を形成したが、その S. × townsendii でゲノム重複が起こった結果、繁殖力と生命力の強い同質倍数体(12倍体、複二倍体)の新しい種 S. anglica が形成され、イギリスとフランスの塩性湿地や河口を急速に帰化した[26][28]。両親種は限られた分布を保っているため、倍数化によってより適応的な形質を獲得したといえる[26]。
異質倍数性と同質倍数性
[編集]倍数性は大きく分けて、1つの種のゲノムが2倍になる同質倍数性と、2つの種のゲノムが結合する異質倍数性がある。異質倍数体は近縁種同士の交配によって生じるのに対し、同質倍数体は同種の親同士の交配[29]もしくは親の生殖組織における体細胞倍加によって、1つの種のゲノムが重複することによって生じる。自然界では異質倍数体の方が多く存在すると考えられているが[29]、これは異なるゲノムを受け継いでヘテロ接合性が高くなり、より高い適応度が得られるためと考えられる。このような異なるゲノムは、有利に働くこともあるが有害になることもある大規模なゲノム再編成の可能性を高める[29][30]。それに対し、一般に同質倍数性は中立的な過程だと考えられているが[30]、異質倍数性の種では必要な時間とコストのかかるゲノム再編成を受けずに新しい生息地に素早く適応することができ、種分化をもたらす有用な機構として機能するという仮説が立てられている。自家受粉が可能な完全花では、減数分裂のエラーによる異数性とともに、同質倍数性の可能性が非常に高い環境を作り出すことができる。
倍数化イベントの後、重複した遺伝子には、両方の重複遺伝子が機能的な遺伝子として保持される可能性や、どちらかか両方の重複遺伝子で機能の変化が起こる可能性、遺伝子サイレンシングにより片方または両方の重複遺伝子に起こる可能性、そして完全な遺伝子欠失が起こる可能性が考え得る[29][31]。ゲノム重複の後、時間が経つにつれて、重複遺伝子の機能が変化するか[注釈 4]、同質倍数性においてゲノム重複によって引き起こされるゲノム再配置によって遺伝子発現に変化が生じるかによって多くの遺伝子の機能が変化する。ある遺伝子の重複遺伝子が両方とも保持され、コピー数が2倍になると、その遺伝子の発現が比例して増加し、2倍のmRNA転写物が生産される可能性がある。また、重複した遺伝子の転写が抑制され、その遺伝子の転写の増加が2倍以下になる可能性もあるし、重複によって転写が2倍以上に増加する可能性もある[32]。例えば、ダイズ属のナガミツルマメ Glycine dolichocarpa では、約50万年前に起こったゲノム重複の後、転写が1.4倍増加したことが観察されており、遺伝子コピー数が倍になったのに対して転写が相対的に減少したことが示唆されている[32]。
脚注
[編集]注釈
[編集]出典
[編集]- ^ a b 農林水産省 2007, p. 8.
- ^ a b 金刺 2018, p. 1.
- ^ Kellis et al. 2004, pp. 617–624.
- ^ a b Bowers et al. 2003, pp. 433–438.
- ^ a b Wolfe 2001, pp. 333–341.
- ^ a b Blanc & Wolfe 2004a, pp. 1667–1678.
- ^ a b Blanc & Wolfe 2004b, pp. 1679–1691.
- ^ Wang et al. 2011, pp. 1035–1039.
- ^ Paterson, Bowers & Chapman 2004, pp. 9903–9908.
- ^ Tang et al. 2010, pp. 472–477.
- ^ Callaway 2013.
- ^ Adams 2013, pp. 1456–1457.
- ^ Tang et al. 2008a, pp. 486–488.
- ^ Tang et al. 2008b, pp. 1944–1954.
- ^ Jaillon et al. 2009, pp. 463–467.
- ^ 山岡千鶴、栗田修、山﨑栄次「清酒酵母とクリベロミセス属酵母との異種酵母混合培養法による清酒小仕込試験」『日本醸造協会誌』第109巻第9号、2014年、679-686頁、doi:10.6013/jbrewsocjapan.109.679。
- ^ Wong et al. 2002, pp. 9272–9277.
- ^ a b c d e 工樂 2018, p. 156.
- ^ 工樂 2018, p. 157.
- ^ Ohno 1970.
- ^ Furlong & Holland 2002, p. 531-544.
- ^ Putnam et al. 2008, pp. 1064–1071.
- ^ Smith & Keinath 2015, pp. 1081–1090.
- ^ 中尾 2018, p. 509.
- ^ Clarke, Lloyd & Friedman 2016, pp. 11531–11536.
- ^ a b c te Beest et al. 2012, pp. 19–45.
- ^ 環境省 2005.
- ^ 環境省 2011, p. 5.
- ^ a b c d Soltis & Soltis 2000, pp. 7051–7057.
- ^ a b Parisod, Holderegger & Brochmann 2010, pp. 5–17.
- ^ Wendel 2000, pp. 225–249.
- ^ a b Coate & Doyle 2010, pp. 534–546.
参考文献
[編集]- Adams, K (2013-12). “Genomics. Genomic clues to the ancestral flowering plant”. Science 342 (6165): 1456-1457. Bibcode: 2013Sci...342.1456A. doi:10.1126/science.1248709. PMID 24357306.
- te Beest, Mariska; Le Roux, Johannes J.; Richardson, David M.; Brysting, Anne K.; Suda, Jan; Kubešová, Magdalena; Pyšek, Petr (2012-01). “The more the better? The role of polyploidy in facilitating plant invasions”. Annals of Botany 109 (1): 19-45. doi:10.1093/aob/mcr277.
- Blanc, G; Wolfe, KH (2004-07). “Widespread paleopolyploidy in model plant species inferred from age distributions of duplicate genes”. The Plant Cell 16 (7): 1667-1678. doi:10.1105/tpc.021345. PMC 514152. PMID 15208399.
- Blanc, G; Wolfe, KH (July 2004-07). “Functional divergence of duplicated genes formed by polyploidy during Arabidopsis evolution”. The Plant Cell 16 (7): 1679-1691. doi:10.1105/tpc.021410. PMC 514153. PMID 15208398.
- Bowers, JE; Chapman, BA; Rong, J; Paterson, AH (2003-03). “Unravelling angiosperm genome evolution by phylogenetic analysis of chromosomal duplication events”. Nature 422 (6930): 433-438. Bibcode: 2003Natur.422..433B. doi:10.1038/nature01521. PMID 12660784.
- Callaway, E (2013-12). “Shrub genome reveals secrets of flower power”. Nature. doi:10.1038/nature.2013.14426.
- Clarke, JT; Lloyd, GT; Friedman, M (2016-10). “Little evidence for enhanced phenotypic evolution in early teleosts relative to their living fossil sister group”. Proceedings of the National Academy of Sciences of the United States of America 113 (41): 11531-11536. doi:10.1073/pnas.1607237113. PMC 5068283. PMID 27671652.
- Coate, JE; Doyle, JJ (2010). “Quantifying whole transcriptome size, a prerequisite for understanding transcriptome evolution across species: an example from a plant allopolyploid”. Genome Biology and Evolution 2: 534-546. doi:10.1093/gbe/evq038. PMC 2997557. PMID 20671102.
- Furlong, RF; Holland (2002-04). “Were vertebrates octoploid?”. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 357 (1420): 531-544. doi:10.1098/rstb.2001.1035. PMC 1692965. PMID 12028790.
- Jaillon, O; Aury, JM; Noel, B; Policriti, A; Clepet, C; Casagrande, A et al. (2007-09). “The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla”. Nature 449 (7161): 463-467. Bibcode: 2007Natur.449..463J. doi:10.1038/nature06148. PMID 17721507.
- Kellis, M; Birren, BW; Lander, ES (2004-04). “Proof and evolutionary analysis of ancient genome duplication in the yeast Saccharomyces cerevisiae”. Nature 428 (6983): 617-624. Bibcode: 2004Natur.428..617K. doi:10.1038/nature02424. PMID 15004568.
- Ohno, Susumu (1970). Evolution by Gene Duplication. Springer-Verlag. ISBN 978-3-642-86659-3
- Parisod, C; Holderegger, R; Brochmann, C (2010-04). “Evolutionary consequences of autopolyploidy”. The New Phytologist 186 (1): 5-17. doi:10.1111/j.1469-8137.2009.03142.x. PMID 20070540.
- Paterson, AH; Bowers, JE; Chapman, BA (2004-06). “Ancient polyploidization predating divergence of the cereals, and its consequences for comparative genomics”. Proceedings of the National Academy of Sciences of the United States of America 101 (26): 9903-9908. Bibcode: 2004PNAS..101.9903P. doi:10.1073/pnas.0307901101. PMC 470771. PMID 15161969.
- Putnam, NH; Butts, T; Ferrier, DE; Furlong, RF; Hellsten, U; Kawashima, T; Robinson-Rechavi, M (2008-06). “The amphioxus genome and the evolution of the chordate karyotype”. Nature 453 (7198): 1064-1071. Bibcode: 2008Natur.453.1064P. doi:10.1038/nature06967. PMID 18563158.
- Smith, JJ; Keinath, MC (2015-08). “The sea lamprey meiotic map improves resolution of ancient vertebrate genome duplications”. Genome Research 25 (8): 1081-1090. doi:10.1101/gr.184135.114. PMC 4509993. PMID 26048246.
- Soltis, PS; Soltis, DE (2000-06). “The role of genetic and genomic attributes in the success of polyploids”. Proceedings of the National Academy of Sciences of the United States of America 97 (13): 7051-7057. Bibcode: 2000PNAS...97.7051S. doi:10.1073/pnas.97.13.7051. PMC 34383. PMID 10860970.
- Tang, H; Bowers, JE; Wang, X; Ming, R; Alam, M; Paterson, AH (2008-04). “Synteny and collinearity in plant genomes”. Science 320 (5875): 486-488. Bibcode: 2008Sci...320..486T. doi:10.1126/science.1153917. PMID 18436778.
- Tang, H; Wang, X; Bowers, JE; Ming, R; Alam, M; Paterson, AH (2008-12). “Unraveling ancient hexaploidy through multiply-aligned angiosperm gene maps”. Genome Research 18 (12): 1944-1954. doi:10.1101/gr.080978.108. PMC 2593578. PMID 18832442.
- Tang, H; Bowers, JE; Wang, X; Paterson, AH (2010-01). “Angiosperm genome comparisons reveal early polyploidy in the monocot lineage”. Proceedings of the National Academy of Sciences of the United States of America 107 (1): 472-477. Bibcode: 2010PNAS..107..472T. doi:10.1073/pnas.0908007107. PMC 2806719. PMID 19966307.
- Wang, X; Wang, H; Wang, J; Sun, R; Wu, J; Liu, S; Bai, Y; Mun, JH et al. (2011-08). “The genome of the mesopolyploid crop species Brassica rapa”. Nature Genetics 43 (10): 1035-1039. doi:10.1038/ng.919. PMID 21873998.
- Wendel, JF (2000). Genome evolution in polyploids.. 42. 225-249. doi:10.1007/978-94-011-4221-2_12. ISBN 978-94-010-5833-9. PMID 10688139
- Wolfe, KH (2001-05). “Yesterday's polyploids and the mystery of diploidization”. Nature Reviews. Genetics 2 (5): 333-341. doi:10.1038/35072009. PMID 11331899.
- Wong, S; Butler, G; Wolfe, KH (2002-07). “Gene order evolution and paleopolyploidy in hemiascomycete yeasts”. Proceedings of the National Academy of Sciences of the United States of America 99 (14): 9272-9277. Bibcode: 2002PNAS...99.9272W. doi:10.1073/pnas.142101099. PMC 123130. PMID 12093907.
- 金刺佑平 (8 February 2018). CRISPR/Cas9 を介したダイズゲノム編集における遺伝変異誘発の特徴(修士論文発表会 要旨) (PDF) (Report). 北海道大学大学院農学院. 2022年3月6日閲覧。
- 環境省 (2005年). “第3回 特定外来生物等分類群専門家グループ会合(植物)議事録に関する情報(案)”. 環境省. 2022年3月11日閲覧。
- 環境省『外来生物問題調査検討業務報告書 平成23年度 参考資料』(レポート)環境省、2011年。2022年3月11日閲覧。
- 工樂樹洋 著「脊椎動物のゲノム重複ヒトの基本遺伝子セットは約5億年前に成立」、公益社団法人 日本動物学会 編『動物学の百科事典』丸善出版、2018年9月28日、156-157頁。ISBN 978-4621303092。
- 中尾実樹 著「補体系の進化:病原菌感染と戦う太古の仕組み」、公益社団法人 日本動物学会 編『動物学の百科事典』丸善出版、2018年9月28日、508-509頁。ISBN 978-4621303092。
- 農林水産省農林水産技術会議事務局 技術政策課『米国におけるダイズゲノム研究の現状と動向』(レポート)農林水産省農林水産技術会議事務局、2007年1月、1-23頁。2022年3月6日閲覧。
