サイレンサー (遺伝学)
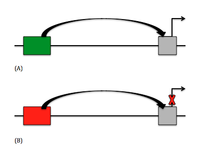
遺伝学においてサイレンサー(英: silencer)は、リプレッサーと呼ばれる転写調節因子が結合するDNA配列である。DNAには遺伝子が含まれ、mRNA産生の鋳型となる。その後、mRNAはタンパク質へと翻訳される。リプレッサーがDNAのサイレンサー領域に結合すると、RNAポリメラーゼによるDNAからRNAへの転写が妨げられる。転写が遮断されるとRNAからタンパク質への翻訳は不可能となる。サイレンサーは、このようにタンパク質としての発現を防いでいる[1]。
サイレンサーについては未解明の部分が多いが、サイレンサーの分類やゲノム上の位置、サイレンサーに関連する疾患などの研究が続けられている[1][2]。
機能
[編集]ゲノム上の位置
[編集]
サイレンサーは、特定の遺伝子の転写に負の影響を与える配列特異的エレメントである。サイレンサーエレメントのDNA上の位置には多くの種類が存在する。最も一般的な位置は転写抑制を行う標的遺伝子の上流である[3]。標的遺伝子からの距離は-20 bpから-2000 bpまで大きな差異が存在する。サイレンサーはプロモーターよりも下流に位置し、遺伝子のイントロンやエクソンの内部に位置している場合もある。また、サイレンサーはmRNAの3' UTRの部分にも存在する[4]。
タイプ
[編集]現在、DNA上のサイレンサーには、典型的なサイレンサーエレメントと非典型的な負の調節エレメント(negative regulatory element、NRE)が知られている。典型的なサイレンサーでは、サイレンサーが基本転写因子の組み立てを防ぐことで遺伝子は抑制される[4]。一方NREは、通常遺伝子の上流に位置する他のエレメントを阻害することで受動的な抑制を行う。NREの中でも、一部のサイレンサーには方向依存性が存在し、すなわちサイレンサーが機能するためには特異的結合因子が他の調節配列に対して特定の向きでに結合していることが必要である。プロモーター依存性のサイレンサーは、位置と方向に依存し、プロモーター特異的な因子も利用するサイレンサーエレメントとして理解されている[4]。近年発見されたポリコーム群応答エレメント(Polycomb-group response element、PRE)は、結合したタンパク質やノンコーディング転写の存在に依存して、抑制を行ったり阻害したりする[3]。
機構
[編集]典型的なサイレンサーでは、そのシグナル伝達経路は比較的単純であり、抑制の際にはサイレンサーエレメントは遺伝子の転写に必要な基本転写因子の組み立てを標的とする。こうしたサイレンサーエレメントは大部分が遺伝子の上流に位置するが、遺伝子との距離は長い場合も短い場合もある。長距離サイレンサーの場合、サイレンサーをプロモーターの近傍にもたらすためDNAがループを形成することが観察されている[3]。サイレンサーはヘリカーゼ結合部位を標的とする場合もある。こうした配列はアデニンとチミンに富むためDNAは巻き戻りやすく、転写を開始するための空間の形成が可能となる。ヘリカーゼ活性の阻害は転写の阻害をもたらす。こうした現象はヒト甲状腺刺激ホルモンβサブユニット遺伝子のプロモーターで一般的に観察される。NREはプロモーター領域の屈曲を誘導して相互作用を遮断する。こうした現象はYY1が結合したNREで観察される[4]。サイレンサー領域がイントロン内にある場合、2種類の抑制が行われる。1つはスプライシング部位の物理的遮断であり、もう1つはRNAのプロセシングを阻害するDNAの屈曲である[4]。
サイレンサーがエクソンまたは非翻訳領域に位置する場合、主に典型的サイレンサーまたは位置依存的サイレンサーとして機能する。これらのサイレンサーは転写の前にその活性を発揮する。ほとんどのサイレンサーは生体内で構成的に活性化されており、サイレンサーを阻害するか、エンハンサー領域を活性化することによってのみ、遺伝子の活性化が可能となる。その最たる例が、REST遺伝子によって産生されるNRSF(neuronal-restrictive silencer factor)である。REST遺伝子はNRSFを産生し、神経組織の局在化に不可欠な神経遺伝子の転写を抑制する[4]。
エンハンサーとの類似性
[編集]遺伝子の上流に位置する他の調節エレメントとしてはエンハンサーがある。エンハンサーは遺伝子発現を「オン」にするスイッチとして機能し、特定の遺伝子のプロモーター領域を活性化する一方、サイレンサーは「オフ」にするスイッチとして機能する。この2つの制御要素は互いに相反する機能を果たすが、どちらの配列タイプも非常によく似た方法でプロモーター領域に影響を与える[3]。サイレンサーはまだ十分な同定と分析がなされていないため、エンハンサーに関する広範な研究がサイレンサーの仕組みを理解する上で生物学者の助けとなっている。サイレンサーが位置する領域の多くにはエンハンサーも存在し、プロモーターの何千塩基対も上流や、下流の遺伝子のイントロン内にも位置している[3]。また、エンハンサーがDNAのループ形成を利用してプロモーターとの距離を縮める。サイレンサーがリプレッサーとともに機能するのと同様に、エンハンサーは転写因子とともに機能して発現を開始させる[3]。
原核生物と真核生物におけるサイレンサー
[編集]原核生物
[編集]
真核生物と原核生物では、代謝制御にいくつかの差異が存在する。原核生物は、細胞内で作られる特定の酵素の数を変化させる、遺伝子発現の調節によるゆっくりとした代謝制御を行うとともに、フィードバック阻害やアロステリック調節などの機構によって、酵素経路の調節による迅速な代謝制御も行っている[5]。原核生物の遺伝子は機能の類似性に基づいてまとまって存在し、プロモーターとオペレーターを含むオペロンと呼ばれる単位にまとめられている。オペレーターはリプレッサーの結合部位であり、真核生物のDNAにおけるサイレンサー領域と同等の機能を持つ。リプレッサータンパク質がオペレーターに結合していると、RNAポリメラーゼはプロモーターに結合できず、オペロンの転写は開始されない。
lacオペロンの抑制
[編集]原核生物の大腸菌Escherichia coliのlacオペロンは、ラクトースを分解する酵素を産生する遺伝子から構成される。このオペロンは原核生物におけるサイレンサーの一例である。このオペロンの3つの機能的遺伝子は、lacZ、lacY、lacAである[5]。リプレッサーの遺伝子lacIはリプレッサータンパク質LacIを産生し、LacIはアロステリック調節が行われる。lacオペロンの遺伝子はラクトースの存在下で活性化され、ラクトースはLacIに結合するエフェクター分子として機能する。ラクトースが結合したリプレッサーはオペレーターへ結合せず、RNAポリメラーゼがプロモーターに結合してオペロンの転写を開始できる状態となる。リプレッサーのアロステリック部位にラクトースが結合していないときには、リプレッサーの活性部位はオペレーターに結合し、RNAポリメラーゼによるlacオペロンの転写を阻害する。
真核生物
[編集]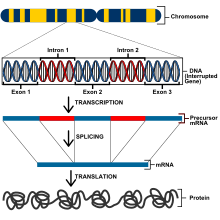
真核生物は原核生物よりもずっと大きなゲノムを持ち、異なる遺伝子調節の手法が存在する。真核生物の個体の全ての細胞は同じDNAを持つが、遺伝子発現の差異によって各細胞に特有の機能が生じる。この現象は遺伝的な全能性として知られる[6]。細胞が適切に機能するためには、遺伝子発現の厳密な制御が必要である。真核生物の遺伝子は、転写、転写後、翻訳、翻訳後の段階で制御されている[7]。転写レベルでは、遺伝子発現は転写率を変化させることで調節される。タンパク質をコードする遺伝子には、ポリペプチドをコードするエクソン、タンパク質への翻訳する前にmRNAから除去されるイントロン、RNAポリメラーゼが結合する転写開始点、プロモーターなどが存在している[8]。
TATAボックスの抑制
[編集]
真核生物の遺伝子には、上流のプロモーターとコアプロモーター(基本プロモーター)が存在する。一般的なコアプロモーターはTATAAAAAA配列であり、TATAボックスとして知られている。TATAボックスはいくつかのタンパク質と複合体を形成する。TFIIDには、TATAボックスに結合するTATA結合タンパク質(TBP)とTBPに結合する13個のタンパク質が含まれている。TATAボックスに結合するタンパク質には、DNAとRNAポリメラーゼの双方に結合するTFIIBも含まれる[8]。
真核生物のサイレンサーは、mRNAへの転写が起こる前の転写段階で遺伝子発現を制御する。これらのDNA配列は、結合する転写因子に基づいて、サイレンサーまたはエンハンサーのいずれかとして作用し、サイレンサーへの結合はTATAボックスなどのプロモーターへのRNAポリメラーゼの結合を妨げる[6]。リプレッサータンパク質にはDNA配列に結合する領域と遺伝子のプロモーターで組み立てられた転写因子に結合する領域が存在する場合があり、これらによって染色体のループ化機構が形成される[8]。ループの形成によってサイレンサーがプロモーターの近傍にもたらされ、最適な遺伝子発現に必要なタンパク質群の協働が保証される。
サイレンサーが関係する変異の影響
[編集]遺伝子変異は生物のヌクレオチド配列が変化した際に生じる。こうした変異は個体の観察可能な表現型に影響を与えるだけでなく、表現型では検出することができない変化も生じる。こうした変異は複製時のエラー、偶発的な変異、化学的・物理的な変異原(紫外線や電離放射線、熱)などによって生じる[9]。ゲノムにコードされているサイレンサーはこうした変化に対する感受性があり、サイレンサーの変異は多くの場合、深刻な表現型的・機能的異常を引き起こす。一般的に、サイレンサーエレメントの変異はサイレンサー作用の阻害か必要な遺伝子の強固な抑制のいずれかを引き起こす。その結果、望ましくない表現型の発現や抑制が引き起こされ、個体の特定の系の正常な機能に影響が生じる。サイレンサーエレメントやそこに結合する多くのタンパク質の中でも、REST/NSRFは重要なサイレンサー結合因子であり、神経発生に限らずさまざまな影響を与える。多くの場合、REST/NSRFはRE-1/NRSE(neuron-restrictive silencer element)とともに抑制機能を果たし、神経細胞以外にも影響を与える[10]。その影響はカエル(アフリカツメガエルXenopus laevis)からヒトにまで及び、表現型と発生に多くの影響を与える。アフリカツメガエルでは、REST/NRSFの機能不全や損傷は発生過程での外胚葉のパターン形成の異常と関係しており、神経管、脳神経節、眼の発生に重大な影響を与える[11]。ヒトでは、REST/NRSFはBDNFの転写を減少させ、ハンチントン病と関係している。
さらに現在進行中の研究では、NRSEは心房性ナトリウム利尿ペプチド(ANP)をコードする遺伝子の調節に関与していることが示されている。この遺伝子の過剰発現は心室肥大を引き起こす[12]。ポリコーム群(PcG)複合体の変異も生物の生理系に大きな変化をもたらす。このように、サイレンサー結合因子やサイレンサー配列の変化は壊滅的な変化をもたらす場合も、目立たない変化をもたらす場合もある。
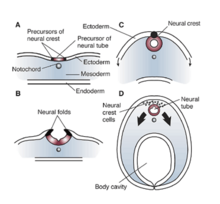
アフリカツメガエルのREST/NRSF
[編集]RE1/NRSEとREST/NRSFの効果と影響は、神経関連遺伝子の抑制(サイレンシング)を必要とする非神経細胞において重要である。これらのサイレンサーエレメントは神経特異的タンパク質以外の発現も調節し、これらの因子が広範囲の細胞過程に及ぼす影響の研究が行われている。アフリカツメガエルでは、RE1/NRSEとREST/NRSFの調節異常または変異は、神経管、脳神経節、眼の発生に大きな影響を与えることが示されている[11]。こうした変化はすべて、発生時の外胚葉の不適切なパターン形成を原因とするものである。RE1/NRSEまたはREST/NRSF変異や変化はいずれも分化や神経上皮ドメインの規定に異常をもたらし、皮膚や外胚葉の形成も妨げる[11]。これらの因子の欠損は骨形成タンパク質(BMP)の産生の低下をもたらし、その結果として神経堤の発生に欠陥が生じる[11]。
REST/NRSFとハンチントン病
[編集]ハンチントン病(HD)は遺伝性の神経変性疾患であり、症状は中年期に生じる。この進行性疾患の最も顕著な症状は、認知機能と運動機能の低下、そして行動の変化である[13]。こうした機能低下は、認知症、舞踏運動、そして最終的には死を引き起こす。分子レベルでは、HDはハンチンチンタンパク質(Htt)の変異が原因である。より具体的には、遺伝子の5'末端にCAG配列の異常な反復が存在し、タンパク質に有毒なポリグルタミン配列(poly-Q)が生じる。Httタンパク質は、REST/NRSFの作用を阻害することで適切な神経機能に影響を与える。REST/NRSFは神経機能に関与する特定のタンパク質の遺伝子の調節領域に結合して発現を制御する重要な因子である。Httの作用機序は十分には理解されていないが、HDの発症にはHttとREST/NRSFが関係している。HttはREST/NRSFに結合して細胞質に保持し、サイレンサーエレメントへの作用を阻害する。すなわち、REST/NRSFは核へ移行して21塩基対のRE-1/NRSE調節エレメントへ結合することができなくなる。変異型ハンチンチンタンパク質はREST/NRSFを細胞質に保持することができず、REST/NRSFは核に移行して脳由来神経栄養因子(BDNF)遺伝子の転写を低下させる。BDNFは中枢神経系と末梢神経系の神経細胞の生存と発達に影響を与える。この異常な抑制は、BDNFのプロモーター領域内のRE1/NRSE領域がREST/NRSFの結合によって活性化され、BDNF遺伝子の転写が行われなくなるためである。また、REST/NRSFは神経の受容体、神経伝達物質、シナプス小胞タンパク質、チャネルタンパク質の適切な発現にも関与しており、これらのタンパク質の発現の欠乏によってハンチントン病でみられるような神経機能障害が引き起こされる[14]。
REST/NRSFと哺乳類の心室肥大
[編集]REST/NRSFは、RE1/NRSEとともに、神経系外でも調節因子やリプレッサーとして作用する。現在の研究では、RE1/NRSEの活性は心房性ナトリウム利尿ペプチド(ANP)遺伝子の発現の調節と関連づけられている。ANP遺伝子の3' UTRにはNRSE調節領域が存在し、その適切な発現の媒介因子として作用している。ANP遺伝子にコードされるタンパク質は、胚発生の過程における心筋細胞の成熟と発生に重要な役割を果たしている。しかし、幼少期から成人期にかけて、心室ではANPの発現は抑制されているか、最小限に抑えられている。そのため、ANP遺伝子の異常な誘導は心室肥大や心臓に深刻な影響を引き起こす可能性がある。この遺伝子の抑制を維持するために、ANP遺伝子の3'非翻訳領域のNRSE領域にREST/NRSFが結合する。さらに、NRSF-NRSE複合体はmSin3と呼ばれる転写コリプレッサーをリクルートする[12]。その結果、この領域にヒストンデアセチラーゼ活性化がもたらされ、遺伝子が抑制される。このように、REST/NRSFとRE1/NRSEとの関係が心室の筋細胞でのANP遺伝子の発現を調節していることが明らかにされている。NRSFまたはNRSEのいずれかに変異があると、抑制が起こらないために心室の筋細胞で望ましくない発生が行われ、心室肥大が引き起こされる可能性がある。一例として、左心室肥大は心室心筋重量の増加を原因とする心室性不整脈による突然死の可能性を高める[15]。ANP遺伝子への影響に加えて、NRSE配列は脳性ナトリウム利尿ペプチド(BNP)、細胞骨格のα-アクチン、Na+/K+-ATPアーゼのα3サブユニットなど、心臓に関する他の胚性遺伝子も調節している[12]。このように、哺乳類におけるNRSEとNRSFによる調節活性は神経機能の異常だけでなく、体の非神経領域の生理や表現型の異常も防いでいる。

ポリコーム群タンパク質の変異
[編集]ポリコーム群(PcG)調節複合体は、幹細胞、特に造血幹細胞のエピジェネティックな調節に影響を与えることが知られている。PRC1は造血過程に直接関与しており、例えばPcGタンパク質BMI1などと共に機能する。マウスでの研究では、Bmi1が変異した個体ではミトコンドリアの機能に欠陥が生じ、造血細胞の自己複製能も妨げられることが示されている。同様にPRC2の遺伝子も白血病の一種である急性リンパ性白血病(ALL)などの血液疾患と関係している。このように、ポリコーム群の遺伝子やタンパク質は体内での適切な造血の維持に関与している[16]。
出典
[編集]- ^ a b Doni Jayavelu, Naresh; Jajodia, Ajay; Mishra, Arpit; Hawkins, R. David (2020-02-26). “Candidate silencer elements for the human and mouse genomes”. Nature Communications 11 (1): 1061. doi:10.1038/s41467-020-14853-5. ISSN 2041-1723. PMC 7044160. PMID 32103011.
- ^ Pang, Baoxu; Snyder, Michael P. (2020-03). “Systematic identification of silencers in human cells”. Nature Genetics 52 (3): 254–263. doi:10.1038/s41588-020-0578-5. ISSN 1546-1718. PMC 7148122. PMID 32094911.
- ^ a b c d e f Maston, Glenn; Sarah Evans; Michael Green (23 May 2006). “Transcriptional regulatory elements in the Human Genome”. Annual Review of Genomics and Human Genetics 7: 29–59. doi:10.1146/annurev.genom.7.080505.115623. PMID 16719718 2 April 2013閲覧。.
- ^ a b c d e f Ogbourne, Steven; Toni Antalis (1998). “Transcriptional control and the role of silencers in transcriptional regulation in eukaryotes”. Biochem. J. 331 (1): 1–14. doi:10.1042/bj3310001. PMC 1219314. PMID 9512455.
- ^ a b “Control of Genetic Systems in Prokaryotes and Eukaryotes”. University of Illinois at Chicago. 2 April 2013閲覧。
- ^ a b “Eukaryotic Gene Control”. Kenyon College. 1 April 2013閲覧。
- ^ “Gene Regulation in Eukaryotes”. Eastern Michigan University. 7 April 2013閲覧。
- ^ a b c “Gene Regulation in Eukaryotes”. Kimball's Biology Pages. 2021年6月12日閲覧。
- ^ Brown, TA (2002). Genomes. Oxford: Wiley-Liss
- ^ Schoenherr, CJ; Anderson DJ (3 March 1995). “The neuron-restrictive silencer factor (NRSF): a coordinate repressor of multiple neuron-specific genes.”. Science 267 (5202): 1360–3. doi:10.1126/science.7871435. PMID 7871435.
- ^ a b c d Olguín, Patricio; Oteíza, Pablo; Gamboa, Eduardo; Gómez-Skármeta, José Luis; Kukuljan, Manuel (2006-03-08). “RE-1 silencer of transcription/neural restrictive silencer factor modulates ectodermal patterning during Xenopus development”. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience 26 (10): 2820–2829. doi:10.1523/JNEUROSCI.5037-05.2006. ISSN 1529-2401. PMC 6675167. PMID 16525062.
- ^ a b c Kuwahara, Koichiro; Yoshihiko Saito; Emiko Ogawa; Nobuki Takahashi; Yasuaki Nakagawa; Yoshihisa Naruse; Masaki Harada; Ichiro Hamanaka et al. (21 March 2001). “The Neuron-Restrictive Silencer Element–Neuron-Restrictive Silencer Factor System Regulates Basal and Endothelin 1-Inducible Atrial Natriuretic Peptide Gene Expression in Ventricular Myocytes”. Molecular and Cellular Biology 21 (6): 2085–97. doi:10.1128/MCB.21.6.2085-2097.2001. PMC 86819. PMID 11238943.
- ^ Walker, FO (20 January 2007). “Huntington's disease.”. Lancet 369 (9557): 218–28. doi:10.1016/S0140-6736(07)60111-1. PMID 17240289.
- ^ Zuccato, Chiara; Belyaev, Nikolai; Conforti, Paola; Ooi, Lezanne; Tartari, Marzia; Papadimou, Evangelia; MacDonald, Marcy; Fossale, Elisa et al. (2007-06-27). “Widespread disruption of repressor element-1 silencing transcription factor/neuron-restrictive silencer factor occupancy at its target genes in Huntington's disease”. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience 27 (26): 6972–6983. doi:10.1523/JNEUROSCI.4278-06.2007. ISSN 1529-2401. PMC 6672230. PMID 17596446.
- ^ Rials, S. J.; Wu, Y.; Ford, N.; Pauletto, F. J.; Abramson, S. V.; Rubin, A. M.; Marinchak, R. A.; Kowey, P. R. (1995-01-15). “Effect of left ventricular hypertrophy and its regression on ventricular electrophysiology and vulnerability to inducible arrhythmia in the feline heart”. Circulation 91 (2): 426–430. doi:10.1161/01.cir.91.2.426. ISSN 0009-7322. PMID 7805247.
- ^ Sashida, Goro; Iwama, Atsushi (2012-10). “Epigenetic regulation of hematopoiesis”. International Journal of Hematology 96 (4): 405–412. doi:10.1007/s12185-012-1183-x. ISSN 1865-3774. PMID 23054647.
外部リンク
[編集]- Silencer Elements - MeSH・アメリカ国立医学図書館・生命科学用語シソーラス
