DPANN群
表示
| DPANN | |||||||||
|---|---|---|---|---|---|---|---|---|---|
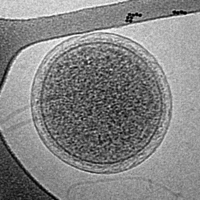 Ca. P. acidiphilumの電子顕微鏡写真
| |||||||||
| 分類 | |||||||||
| |||||||||
| 学名 | |||||||||
| なし | |||||||||
| 門 | |||||||||
DPANN群(DPANNぐん、DPANN group)とは、2013年にリンケらによって提唱された古細菌の系統及び分類に関する仮説である。ナノ古細菌を中核としたグループで、上門相当とされる。DPANNというのはDiapherotrites、Parvarchaeota、Aenigmarchaeota、Nanohaloarchaeota、Nanoarchaeotaの頭文字をとったもので、ラテン語による命名は行われていない。これを古細菌を分けて考える仮説では、系統樹的には、生命誕生後、細菌となるグループと別れたグループの中から、後の古細菌や真核生物に繋がるグループよりも先に分化した形で表現されることが多い。
これら5系統は何れも未培養系統であり、名称も暫定的なものである。しかし、パルウ古細菌、ナノ好塩古細菌、ナノ古細菌門については集積培養系が存在しており、電子顕微鏡により形状が観察されていたり、全ゲノムが解析された株が存在する。非常に小さな細胞サイズと、コンパクトにまとまったゲノムが特徴である。パルウ古細菌とナノ古細菌門については、他の古細菌に付着して増殖する寄生性生物である。
一方で、これらの生物は進化速度が速く、DPANN群は長枝誘引によるアーティファクトとする主張もある。これを考慮した解析では、DPANNは存在せず、実際にはユーリ古細菌のバラバラな位置に現れるという。[1]
分類
[編集]- "ディアペロトリテス"/"Diapherotrites"/pMC2A384
- 1999年、明神海丘(伊豆・小笠原弧)の水深1330mにあるブラックスモーカーより最初に報告されたもの[2]。これまでにいくつかのサンプルが報告されており、何れも純粋培養には成功していないが、"Candidatus Iainarchaeum andersonii"と呼ばれる株についてゲノムの大部分が解析されている[3]。
- "パルウ古細菌"/"Parvarchaeota"
- 2006年に強酸性の排水中から発見されたもの[4]。アルカリ温泉からも発見されている[5]。"Ca. Parvarchaeum acidiphilum"、"Ca. P. acidophilus"のゲノムサイズは、それぞれ801 kbp、921 kbpである[6]。これらはテルモプラズマ目古細菌に付着して生育すると考えられている。
- "ミクル古細菌"/"Micrarchaeota"
- 以前はパルウ古細菌と併せてARMANと呼ばれていた。"Ca. Micrarchaeum acidiphilum"は長さ200 nm×幅60 nm、体積も0.009 μm3から0.04 μm3しかなく[7]、ゲノムサイズも999 kbpしかない。パルウ古細菌と同様テルモプラズマ目古細菌に関係していると考えられている。
- "アエニグム古細菌"/"Aenigmarchaeota"/DSEG[8]
- 深海の熱水噴出孔に存在する系統。"Ca. Aenigmarchaeum subterraneum"と呼ばれる株などはゲノムの一部が読まれている。DHVE3、DUSEL2、104A5などと呼ばれる系統もここに入る。
- "ナノ好塩古細菌"/"Nanohaloarchaeota"
- 高塩環境に分布する。分布は非常に広範囲である。高度好塩菌よりも細胞やゲノムサイズが小さい。"Ca. Nanosalina sp." J07AB43(1,227 kbp)、"Ca. Nanosalinarum sp." J07AB56(1,216 kbp)[9]の2系統について全ゲノムが解読されている。平均サイズは0.6 μm程度である[9]。
- "ナノ古細菌"/"Nanoarchaeota"
- 2002年に発見されたIgnicoccus hospitalisに寄生する古細菌、"Ca. Nanoarchaeum equitans"に対して提唱されたもの。この他スルフォロブス目古細菌に寄生する"Ca. Nanobsidianus stetteri" Nst1などが知られる。細胞サイズは"N. equitans"が400 nm、ゲノムサイズはそれぞれ499 kbp("N. equitans")[10]、593 kbp("N. stetteri")[11]とごく小型で、"N. equitans"のゲノムサイズは古細菌最小である。
参考文献
[編集]- ^ Petitjean, C., Deschamps, P., López-García, P., and Moreira, D. (2014). “Rooting the Domain archaea by phylogenomic analysis supports the foundation of the new kingdom proteoarchaeota.”. Genome Biol. Evol. 7: 191-204.
- ^ Takai, K. & Horikoshi, K. Genetic diversity of Archaea in deep-sea hydrothermal vent environments. Genetics 152, 1285–1297 (1999).
- ^ http://genomesonline.org/cgi-bin/GOLD/bin/GOLDCards.cgi?goldstamp=Gi15106
- ^ Baker, B. J., Tyson, G. W., Webb, R. I., et al.(2006)“Lineages of acidophilic archaea revealed by community genomic analysis,” Science, 314(5807):1933–1935.
- ^ Murakami, S., Fujishima, K., Tomita, M., Kanai, A.(2012) Metatranscriptomic analysis of microbes in an ocean-front deep subsurface hot spring reveals novel small RNAs and type-specific tRNA degradation. Applied and Environmental Microbiology 78(4):1015-22.
- ^ 1.B. J. Baker, L. R. Comolli, G. J. Dick, L. J. Hauser, D. Hyatt, B. D. Dill, M. L. Land, N. C. VerBerkmoes, R. L. Hettich, J. F. Banfield. Enigmatic, ultrasmall, uncultivated Archaea. Proceedings of the National Academy of Sciences (2010).
- ^ Comolli LR, Baker BJ, Downing KH, Siegerist CE, Banfield JF (2009) Three-dimensional analysis of the structure and ecology of a novel, ultra-small archaeon. ISME J 3:159–167.
- ^ Takai, K., Moser, D. P., DeFlaun, M., Onstott, T. C. & Fredrickson, J. K. Archaeal diversity in waters fromdeep south african gold mines. Appl. Environ. Microbiol. 67, 5750–5760 (2001).
- ^ a b Narasingarao, P. et al. (2012). "De novo metagenomic assembly reveals abundant novel major lineage of Archaea in hypersaline microbial communities". ISME J. 6 (1): 81–93.
- ^ Waters, E., Hohn, M. J., Ahel, I., Graham, D. E., Adams, M. D., Barnstead, M., Beeson, K. Y., Bibbs, L., Bolanos, R., Keller, M., Kretz, K., Lin, X., Mathur, E., Ni, J., Podar, M., Richardson, T., Sutton, G. G., Simon, M., Soll, D., Stetter, K. O., Short, J. M., Noordewier, M. (2003). “The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism”. Proc Natl Acad Sci U S A 100 (22): 12984–8.
- ^ Podar, M., Makarova, K. S., Graham, D. E., Wolf, Y. I., Koonin, E. V., Reysenbach, A. L. (2013). Insights into archaeal evolution and symbiosis from the genomes of a nanoarchaeon and its inferred crenarchaeal host from Obsidian Pool, Yellowstone National Park. Biol. Direct 8:9
