DMSOレダクターゼ
| Dimethylsulfoxide reductase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 識別子 | |||||||||
| EC番号 | 1.8.5.3 | ||||||||
| データベース | |||||||||
| IntEnz | IntEnz view | ||||||||
| BRENDA | BRENDA entry | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | KEGG entry | ||||||||
| MetaCyc | metabolic pathway | ||||||||
| PRIAM | profile | ||||||||
| PDB構造 | RCSB PDB PDBj PDBe PDBsum | ||||||||
| |||||||||
DMSOレダクターゼ (DMSO reductase, DMSOR) はジメチルスルホキシド (DMSO) からジメチルスルフィド (DMS) への還元反応を触媒するモリブデン含有酵素である。特定の細菌では嫌気的条件下において、この酵素はDMSOを最終電子受容体とする末端還元酵素として機能する。一連の反応でDMSOの酸素原子はモリブデンに移され、さらに水へと還元される。
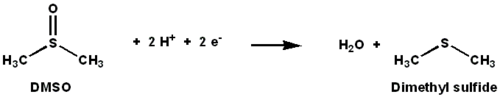
DMSOレダクターゼや他のDMSOレダクターゼファミリーは細菌と古細菌に特有の酵素である。これらの酵素はオキソアニオンを分解するよう設計されており、嫌気的な酸化的リン酸化や無機物を電子供与体とした無機栄養呼吸に用いられる[1][2][3]。DMSORは2つの電子を1つの酸素原子に移動する反応を触媒し、その活性部位には生物において稀な元素であるモリブデンが含まれている[2]。
三次構造と活性部位
[編集]
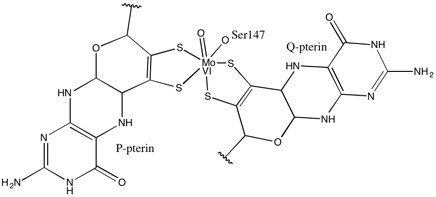
他のDMSOレダクターゼファミリーの酵素と同様に、DMSORの三次構造はモリブデンを取り巻くI-IVのドメインから構成されている。ドメインIVは活性部位のピラノプテリンジチオレンモリブデン補因子(それぞれP-、Q-プテリンと呼ばれる)と強く相互作用している[2][3]。
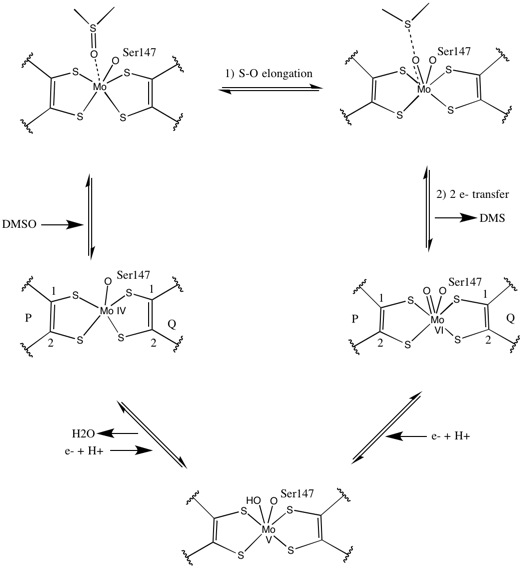
DMSOレダクターゼファミリーはその活性部位の構造によって分類される[3]。DMSORの場合、モリブデンは2つのピラノプテリンから供与される2つのジチオレンと結合している。モリブドプテリンと呼ばれるこれらの有機補因子はGMPと結合し、ジヌクレオチド型となっている。5番目の配位子はアミノ酸に由来し、DMSOレダクターゼファミリーのタイプIではシステイン、タイプIIではアスパラギン酸、タイプIII(DMSORを含む)ではセリン残基の側鎖である。研究では、酵素の触媒機構の重要な決定要因となるモリブデンの酸化還元電位と酸素原子のプロトン化状態は、モリブデン中心に配位するアミノ酸の種類に大きく影響されることが示された。モリブデンにさらに配位している酸素原子は、モリブデンの酸化状態によって4価の時はアクア配位子、5価の時はヒドロキソ配位子、6価の時はオキソ配位子となる[2]。
反応機構
[編集]DMSO18同位体を用いた初期の研究によって、Rhodobacter sphaeroides のDMSORにおける連続した酸素原子の移動機構が解明された。この研究では、標識されたO18原子が基質からモリブデンに移された後に1,3,5-トリアザ-7-ホスファアダマンタン (PTA) に移され、PTAO18が生成した[6]。生体内でも同様の機構によって、DMSOの酸素原子がモリブデンに移された後にMo(VI)O中心が還元されて水が生成する[7]。
モリブデンの合成bis-ジチオレン錯体に対する硫黄K吸収端X線吸収分光法とDFT計算を用いたモデル研究から、S-O結合の開裂と電子移動が同時に進むことが示された。反応速度は基質のX-O結合強度低下とプロトン親和性上昇に比例する[8]。
X線結晶構造解析により、酵素の三次構造は反応前後で全体として一定のままであることが確認された。しかしRhodobacter sphaeroidesのDMSORを用いて行われたいくつかの研究では、4つのジチオレン配位子の配位活性について異なる結果が報告されている。あるX線結晶構造解析研究と多数のX線吸収分光法 (XAS) 研究では酸化された状態においてMo-S間の距離は全て等しいと結論されたが、別の研究ではMo-S間の距離は非対称的であると結論された。電子スピン共鳴 (EPR) などの他の研究を含めいずれの研究でも、活性部位は配位子の位置と結合強度の観点から見て非常に柔軟であることが予測された[7][9]。
2つのピラノプテリン補因子が著しく非対称であるデータを基に提唱された反応機構は以下のようなものである。活性中心が完全に酸化され6価モリブデンとなっている場合、モリブデンに対するオキソ配位子とセリンの結合距離は1.7 Å、P-プテリンS1原子、S2原子、Q-プテリンS1原子の結合距離は2.4 Å、Q-プテリンS2原子は3.1 Åだった。P-プテリンとQ-プテリンの非対称性は、Q-プテリンS2原子の反対側に位置するオキソ配位子のトランス効果により結合が弱まっていることによる[7]。
活性中心が完全に還元され4価モリブデンとなっている場合、P-プテリンS1原子、S2原子、Q-プテリンS1原子は完全な配位結合を維持するが、Q-プテリンS2原子は配位結合性を弱めて金属中心から離れる。この結合長変化はDMSO基質からモリブデンへ酸素原子が直接移動する機構として提唱されているものと一致しており、還元型酵素ではジチオレン配位が弱いことによってスルフィニル基 (S=O) の結合が促進される。モリブデンの還元とオキソ配位子のプロトン化の際にはシトクロムから電子が受け渡されるが、その結合部位について、活性部位近くの凹みに結合してモリブデン中心を直接還元する、Q-プテリン近傍のよく溶媒和したポリペプチド鎖に結合してQ-プテリン経由で電子を受け渡す、の2通りの仮説が提唱されている[7]。
細胞での所在と調節
[編集]R. sphaeroides のDMSORは1サブユニットから構成される水溶性タンパク質で、プテリン以外の補因子は必要としない。大腸菌のDMSORは膜タンパク質で、プテリン補因子の結合したサブユニット、4Fe:4Sクラスターを含むサブユニット、還元型メナキノンの結合した膜貫通サブユニットの3サブユニットから構成されている。還元型メナキノンが受け取った電子は4Fe:4Sクラスターを介してプテリン-モリブデン活性部位に受け渡され、これによりATP合成に用いられるプロトン勾配を生み出す[7]。
DMSORは主に転写レベルでの調節を受けている。DMSORをコードするdor遺伝子は、DorS、DorR、DorCタンパクの制御下にある伝達カスケードにより活性化されて発現する。dorS、dorR、dorCプロモーターへのlacZ(レポーター遺伝子)融合遺伝子を用いた研究では、DorRとDorCの発現は酸素濃度の低い環境下で増加したが、DorSの発現は酸素濃度に影響されなかった。また、DorCの発現はDMSO濃度の増加に伴って上昇した[10]。
環境への影響
[編集]DMSORが生成するジメチルスルフィドは硫黄循環の一部を構成する。DMSは酸化されてメタンスルホン酸となり、これは塵などの乏しい大洋上において凝結核として機能する。雲は地球の反射能を増加させ大気の温度に影響を与えるため、この酵素とそれが触媒する反応は気候の制御に利用できる可能性がある[11]。
参考文献
[編集]- ^ Kappler, Ulrike; Schäfer, Hendrik (2014). “Chapter 11. Transformations of Dimethylsulfide”. The Metal-Driven Biogeochemistry of Gaseous Compounds in the Environment. Metal Ions in Life Sciences. 14. Springer. pp. 279–313. doi:10.1007/978-94-017-9269-1_11. ISBN 978-94-017-9268-4. PMID 25416398
- ^ a b c d McEwan, Alistair G.; Kappler, Ulrike (2004). “The DMSO Reductase Family of Microbial Molybdenum Enzymes”. Australian Biochemist 35 (3): 17–20. オリジナルの2014-03-07時点におけるアーカイブ。 2014年2月27日閲覧。.
- ^ a b c d e McEwan, Alistair G.; Ridge, Justin P.; McDevitt, Christopher A.; Hugenholtz, Phillip (2002). “The DMSO Reductase Family of Microbial Molybdenum Enzymes; Molecular Properties and Role in the Dissimilatory Reduction of Toxic Elements”. Geomicrobiology Journal 19 (1): 3–21. doi:10.1080/014904502317246138.
- ^ PDB: 1DMS; “Crystal structure of dimethyl sulfoxide reductase from Rhodobacter capsulatus at 1.88 A resolution”. Journal of Molecular Biology 263 (1): 53–69. (October 1996). doi:10.1006/jmbi.1996.0555. PMID 8890912.
- ^ PDB: 4DMR; “The high resolution crystal structure of DMSO reductase in complex with DMSO”. Journal of Molecular Biology 275 (4): 613–23. (January 1998). doi:10.1006/jmbi.1997.1513. PMID 9466935.
- ^ Schultz, Brian E.; Hille, Russ; Holm, R. H. (1995), “Direct oxygen atom transfer in the mechanism of action of Rhodobacter sphaeroides dimethyl sulfoxide reductase”, Journal of the American Chemical Society 117 (2): 827–828, doi:10.1021/ja00107a031, ISSN 0002-7863
- ^ a b c d e “Molybdenum-cofactor-containing enzymes: structure and mechanism”. Annual Review of Biochemistry 66: 233–67. (1997). doi:10.1146/annurev.biochem.66.1.233. PMID 9242907.
- ^ “Sulfur K-edge X-ray absorption spectroscopy and density functional calculations on Mo(IV) and Mo(VI)=O bis-dithiolenes: insights into the mechanism of oxo transfer in DMSO reductase and related functional analogues”. Journal of the American Chemical Society 132 (24): 8359–71. (June 2010). doi:10.1021/ja910369c. PMC 2907113. PMID 20499905.
- ^ “Molybdenum active centre of DMSO reductase from Rhodobacter capsulatus: crystal structure of the oxidised enzyme at 1.82-A resolution and the dithionite-reduced enzyme at 2.8-A resolution”. JBIC 2 (6): 690–701. (1997). doi:10.1007/s007750050185.
- ^ “Control of electron flow in Escherichia coli: coordinated transcription of respiratory pathway genes”. Journal of Bacteriology 174 (22): 7069–74. (November 1992). PMC 207394. PMID 1331024.
- ^ Sarkar, Bibudhendra (21 March 2002). Heavy Metals In The Environment. CRC Press. p. 456. ISBN 978-0-8247-4475-5
