レサズリン
| レサズリン | |
|---|---|

| |
7-hydroxy-10-oxidophenoxazin-10-ium-3-one | |
別称 Alamar Blue, Vybrant, UptiBlue, diazo-resorcinol, azoresorcin, resazoin, resazurine | |
| 識別情報 | |
| CAS登録番号 | 550-82-3 |
| PubChem | 11077 |
| ChemSpider | 10606 |
| 特性 | |
| 化学式 | C12H7NO4 |
| モル質量 | 229.19 g mol−1 |
| 水への溶解度 | 可溶 |
| 危険性 | |
| GHSピクトグラム | 
|
| GHSシグナルワード | Warning |
| Hフレーズ | H315, H319, H335 |
| Pフレーズ | P261, P305+351+338 |
| NFPA 704 | |
| 特記なき場合、データは常温 (25 °C)・常圧 (100 kPa) におけるものである。 | |
レサズリン (Resazurin) は、フェノキサジン色素の一種である。弱い蛍光を発し、無毒で細胞浸透性があり、酸化還元感受性を持つ[2][3]。
レサズリンは、pH 6.5以上で青色から紫色を呈する。還元されて不可逆的にピンク色と強い蛍光を示すレゾルフィン(7-ヒドロキシ-3H -フェノキサジン-3-オン)になるので、微生物学、細胞生物学、酵素アッセイに用いられる。中性付近のpHでは、レゾルフィンはピンク色の目視観察または蛍光分光分析法によって検出できる。最大励起波長は530 - 570 nm、最大発光波長は580 - 590 nmである[4]。
レゾルフィンを含む溶液を還元条件 (Eh < −110 mV)にかけると、ほとんどすべてのレゾルフィンが半透明非蛍光のジヒドロレゾルフィン(ハイドロレゾルフィン)に可逆的に還元される。溶液は半透明になる(レゾルフィン/ジヒドロレゾルフィンペアの酸化還元電位は、pH 7.0で標準水素電極に対して−51 mVである)。この同じ溶液のEhが増加すると、ジヒドロレゾルフィンは酸化されてレゾルフィンに戻り、この可逆反応を使用して、増殖培地の酸化還元電位が残っているかどうかを嫌気性生物に対して十分に低いレベルで監視できる。
| レサズリン (pH指示薬) | ||
| pH 6.5以下 | pH 6.5以上 | |
| 6.5 | ↔ | 6.5 |
レサズリン溶液は、クレフトの二色性指数で知られている最も高い値の1つを持っている[5]。これは、観察されたサンプルの厚さまたは濃度が増加または減少すると、知覚される色(色相)に大きな変化があることを意味する。
通常、レサズリンは、ナトリウム塩として市販されている。
細胞生存率への応用
[編集]レサズリンは、代謝的に活性な細胞の好気性呼吸によってレゾルフィンに還元され、細胞生存率の指標として使用できる。これは、1929年にPeschとSimmertによって牛乳中の細菌含有量を定量するために最初に使用された[6]。これは哺乳類細胞培養における生細胞の存在を検出するために使用できる[7]。当初はAlamar Blueの商標(Trek Diagnostic Systems, Inc.)で商業的に導入され、現在はABアッセイ、Vybrant(Molecular Probes)、UptiBlue(Interchim)などの他の名前でも入手できる。
レサズリンに基づくアッセイは、ホルマザンを使用したアッセイ(MTT/XTT)やトリチウム標識チミジンを使用した手法などの参照生存率アッセイとの優れた相関関係を示している[8]。毒性が低いため、より長い研究に適しており、動物細胞、細菌、真菌[8]に適用されている。また、細胞計数、細胞の生存、細胞増殖のような細胞培養アッセイに適用されている[9]。
標準的な生/死アッセイの代わりに、レサズリンは、アポトーシスを測定するためのサイトカインアッセイ、カスパーゼアッセイのような化学発光アッセイ、または遺伝子またはタンパク質の発現を測定するためのレポーターアッセイなど多角化されている[8]。
レゾルフィンに対するレサズリンの不可逆反応は有酸素呼吸に比例する[10]。
その他の応用
[編集]
レサズリンはミトコンドリア中で効果的に還元されるため、ミトコンドリア代謝活性の評価にも役立つ。
通常、酵素としてNADPHデヒドロゲナーゼまたはNADHデヒドロゲナーゼが存在する場合、NADPHまたはNADHはレサズリンをレゾルフィンに変換する還元剤である[11][12][13][14][15]。
レサズリンはグルタミン酸のアッセイに使用でき、96ウェルプレートでウェルあたり2.0 pmolの感度を達成する[16]。
レサズリンは、排水中に見られる有機物の好気性生分解を測定するためにも使用できまる[17]。
レサズリンは、小川の有酸素呼吸の量を測定するために使用される[18]。ほとんどの有酸素呼吸は河床で発生するため、レサズリンからレゾルフィンへの変換は、水流と河床の間の交換量の尺度でもある。
合成
[編集]レサズリンは、レゾルシノールと4-ニトロソレゾルシノールの間の酸触媒縮合と、それに続く酸化マンガン(IV)による中間体の酸化によって調製される。
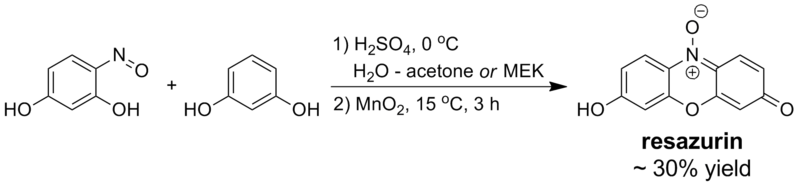
粗反応生成物を過剰の炭酸ナトリウムで処理すると、レサズリンのナトリウム塩が得られる。これは通常、染料の市販形態である。アルコール中で縮合工程を実行することは可能だが、生成物の収率が低下する。 純水または酢酸中では、反応は十分に進行しない[19]。
関連色素
[編集]- 10-アセチル-3,7-ジヒドロキシフェノキサジン
- Amlex Red として知られている) レサズリンに構造的に関連し、H2O2と1:1の化学量論的に反応して、同じ副産物のレゾルフィンを生成する(たとえば、ホースラディッシュペルオキシダーゼ[HRP]、または酵素を使用したNADH、NADPHを組み合わせた多くのアッセイで使用される)[20][21]。
- 7-エトキシレゾルフィン
- 細胞培養におけるエトキシレゾルフィン-O-デエチラーゼ(EROD)アッセイシステムを使用したシトクロムP450(CYP1A1)誘導の測定、および、アリール炭化水素への暴露で生成される環境試料の基質として使用される化合物。この化合物は酵素の触媒作用で同じ蛍光生成物であるレゾルフィンを生成する[22][23]。
- 1,3-ジクロロ-7-ヒドロキシ-9,9-ジメチルアクリジン-2(9H )-オン(DDAO色素)
- オリゴヌクレオチド標識に使用される蛍光色素[24]。
脚注
[編集]- ^ "Resazurin | C12H7NO4 | ChemSpider"
- ^ Bueno, C.; Villegas, M. L.; Bertolotti, S. G.; Previtali, C. M.; Neumann, M. G.; Encinas, M. V. (2002). “The Excited-State Interaction of Resazurin and Resorufin with Aminesin Aqueous Solutions. Photophysics and Photochemical Reaction”. Photochemistry and Photobiology 76 (4): 385–90. doi:10.1562/0031-8655(2002)0760385TESIOR2.0.CO2. PMID 12405144.
- ^ Haggerty, Roy; Martí, Eugènia; Argerich, Alba; Schiller, Daniel von; Grimm, Nancy B. (2009). “Resazurin as a "smart" tracer for quantifying metabolically active transient storage in stream ecosystems” (英語). Journal of Geophysical Research: Biogeosciences 114 (G3). doi:10.1029/2008JG000942. hdl:10261/38263. ISSN 2156-2202.
- ^ Chen, J. L., Steele, T. W., & Stuckey, D. C. (2015). “Modeling and application of a rapid fluorescence-based assay for biotoxicity in anaerobic digestion”. Environ. Sci. Technol. 49 (22): 13463-13471. doi:10.1021/acs.est.5b03050.
- ^ Kreft, Samo; Kreft, Marko (2009). “Quantification of dichromatism: A characteristic of color in transparent materials”. Journal of the Optical Society of America A 26 (7): 1576–81. Bibcode: 2009JOSAA..26.1576K. doi:10.1364/JOSAA.26.001576. PMID 19568292.
- ^ Pesch, K. L.; Simmert, U. (1929). “Combined assays for lactose and galactose by enzymatic reactions”. Milchw. Forsch 8: 551.
- ^ Anoopkumar-Dukie, S; Carey, JB; Conere, T; O'Sullivan, E; Van Pelt, FN; Allshire, A (2005). “Resazurin assay of radiation response in cultured cells”. British Journal of Radiology 78 (934): 945–7. doi:10.1259/bjr/54004230. PMID 16177019.
- ^ a b c UptiBlue viable cell assay technical manual
- ^ Kurin, Elena; Atanasov, Atanas; Donath, Oliver; Heiss, Elke; Dirsch, Verena; Nagy, Milan (2012). “Synergy Study of the Inhibitory Potential of Red Wine Polyphenols on Vascular Smooth Muscle Cell Proliferation”. Planta Medica 78 (8): 772–8. doi:10.1055/s-0031-1298440. PMID 22499559.
- ^ González-Pinzón, Ricardo; Haggerty, Roy; Myrold, David D. (2012). “Measuring aerobic respiration in stream ecosystems using the resazurin-resorufin system”. Journal of Geophysical Research 117 (G3): G00N06. Bibcode: 2012JGRG..117.0N06G. doi:10.1029/2012JG001965.
- ^ Shahangian, S.; Ash, K. O.; Rollins, D. E. (1984). “An Enzymatic Method for the Analysis of Formate in Human Plasma”. Journal of Analytical Toxicology 8 (6): 273–6. doi:10.1093/jat/8.6.273. PMID 6549198.
- ^ Hanson, NQ; Freier, EF (1983). “Effect of protein on the determination of total bile acids in serum”. Clinical Chemistry 29 (1): 171–5. doi:10.1093/clinchem/29.1.171. PMID 6571720.
- ^ De Jong, Donald W.; Woodlief, William G. (1977). “Fluorimetric assay of tobacco leaf dehydrogenases with resazurin”. Biochimica et Biophysica Acta (BBA) - Enzymology 484 (2): 249–59. doi:10.1016/0005-2744(77)90081-X. PMID 20957.
- ^ Barnes, Stephen; Spenney, Jerry G. (1980). “Stoichiometry of the nadh-oxidoreductase reaction for dehydrogenase determinations”. Clinica Chimica Acta 107 (3): 149–54. doi:10.1016/0009-8981(80)90442-8. PMID 6893684.
- ^ Winartasaputra, H; Mallet, VN; Kuan, SS; Guilbault, GG (1980). “Fluorometric and colorimetric enzymic determination of triglycerides (triacylglycerols) in serum”. Clinical Chemistry 26 (5): 613–7. doi:10.1093/clinchem/26.5.613. PMID 6894889.
- ^ Chapman, Justin; Zhou, Mingjie (1999). “Microplate-based fluorometric methods for the enzymatic determination of l-glutamate: application in measuring l-glutamate in food samples”. Analytica Chimica Acta 402 (1–2): 47–52. doi:10.1016/S0003-2670(99)00533-4.
- ^ Jouanneau, S.; Recoules, L.; Durand, M.J; Boukabache, A.; Picot, V.; Primault, Y.; Lakel, A.; Sengelin, M. et al. (2014). “Methods for assessing biochemical oxygen demand (BOD): A review”. Water Research 49: 62–82. doi:10.1016/j.watres.2013.10.066. PMID 24316182.
- ^ Haggerty, Roy; Martí, Eugènia; Argerich, Alba; Von Schiller, Daniel; Grimm, Nancy B. (2009). “Resazurin as a 'smart' tracer for quantifying metabolically active transient storage in stream ecosystems”. Journal of Geophysical Research 114 (G3): G03014. Bibcode: 2009JGRG..114.3014H. doi:10.1029/2008JG000942. hdl:10261/38263.
- ^ A US 2376283 A, Frank Short Wallace & Peter Oxley, "Dyestuffs suitable for use as indicators", published 1945-05-15, assigned to Boots Pure Drug Co Ltd
- ^ Zhou, M., Diwu, Z., Panchuk-Voloshina, N., et al. A stable nonfluorescent derivative of resorufin for the fluorometric determination of trace hydrogen peroxide: Applications in detecting the activity of phagocyte NADPH oxidase and other oxidases. Anal Biochem 253 162-168 (1997)
- ^ “10-Acetyl-3,7-dihydroxyphenoxazine (CAS 119171-73-2) | Cayman Chemical”
- ^ Mohammadi-Bardbori, Afshin (2014). “Assay for quantitative determination of CYP1A1 enzyme activity using 7-Ethoxyresorufin as standard substrate (EROD assay)”. Protocol Exchange (Research Square) 10 (5). doi:10.1038/protex.2014.043.
- ^ Chang, Thomas K.H.; Waxman, David J. (2005). “Enzymatic Analysis of cDNA-Expressed Human CYP1A1, CYP1A2, and CYP1B1 With 7-Ethoxyresorufin as Substrate”. Cytochrome P450 Protocols. Methods in Molecular Biology. 320 (2nd ed.). pp. 85–90. doi:10.1385/1-59259-998-2:85. ISBN 9781588294418. PMID 16719376
- ^ Dominiak, D.M.; Nielsen, J.L.; Nielsen, P.H. (2010). “Extracellular DNA is abundant and important for microcolony strength in mixed microbial biofilms”. Environmental Microbiology 13 (3): 710–721. doi:10.1111/j.1462-2920.2010.02375.x. PMID 21118344.

