ペラジバクター・ユビーク
| ペラジバクター・ユビーク | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||||||||||||||||
| 分類 | ||||||||||||||||||||||||
| ||||||||||||||||||||||||
| 学名 | ||||||||||||||||||||||||
| "Ca. Pelagibacter ubique" Rappé et al. 2002 |
ペラジバクター・ユビーク(Candidatus Pelagibacter ubique)は、2002年に分離されたペラジバクター目に属する細菌の未記載種である[1]。ペラギバクテル・ウビークウェと表記されることもある。
海洋に極めて大量に存在する、プロテオバクテリア門に含まれるSAR11系統 (ペラジバクター目) の中で比較的多く存在しているタイプ種であり、SAR11の中で初めて培養・ゲノム解析が行われた系統でもある[2]。また、P. ubiqueはPelagibacter属のタイプ種である[3][4]。
性質
[編集]SAR11系統は地球上の海水と淡水の両方の環境で普遍的に見られる系統群であり、おそらく地球上で最も個体数の多い生物と考えられている。もともとはオレゴン州立大学微生物学教室のStephen Giovannoniらによって、サルガッソー海のサンプルから得られた16S rRNA遺伝子の配列解析より、その存在が予想され[5]、それ以降の研究から世界中の海水からも発見されていた。存在量が莫大であることから(細胞数ベースで海水中の全微生物プランクトンの25%を占める)、海水中の溶存有機物を同化する主要な細菌として、地球の炭素循環に大きな役割を持つと考えられている。P. ubiqueとその近縁系統の総存在量は、約2×1028細胞と推定されている[6]。
最も研究されているHTCC1062株は、2002年にオレゴン州ニューポートの沖合27.6km地点より採取された[2]。プロテオロドプシンを恒常的に発現しているが、光照射の有無で増殖速度、最終収量ともに差が見られず、プロテオロドプシンは飢餓状態における生存性に関与すると考えられている[7]。
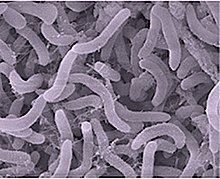
細胞サイズは極めて小さく、桿状または三日月形状の形態を持ち、対数期で長さ0.4〜1.3μm、直径0.19〜0.28μm程度の大きさである[8]。体積は0.019~0.037 µm3程度で、これは大腸菌の1/30程度に過ぎない[8]。暗条件下で飢餓状態に置くと、細胞サイズは0.014 µm3程度まで縮小する[9]。知られているものの中で最小の自己複製細胞の一つである。
Pelagibacterのゲノムは、細胞体積の約30%を占めている[10]。外膜と大きなペリプラズムを持ち、グラム陰性である[11]。溶存有機炭素をリサイクルするように取り込んで栄養とする。季節サイクルの影響を受け、夏の温帯海洋表層水においては海洋中の全細胞の約50%にも達するほど増殖することがある。
2013年2月にP. ubiqueに感染するバクテリオファージとしてHTVC010Pが発見されている[12][13]。
培養方法
[編集]分離技術の向上により、P. ubiqueは複数の株の培養が成功している[14]。最も研究されている株は、HTCC1062株(HTCCはHigh-Throughput Cultivation Collection;ハイスループット培養コレクション)である[3]。この株はP. ubiqueのタイプ株である[3][4]。メチオニン(硫黄源)、グリシン、ピルビン酸、ビタミンを添加した、完全人工培地で生育できる[15]。
SAR11の細胞数を制御している要因の大部分は不明である。この系統は窒素、リン酸塩、および鉄の制限に感受性があり、硫黄化合物を減らす必要があるという非常に珍しい特徴がある[16]。これらは、最初に発見されたサルガッソ海などの低栄養生態系において進化したことによって形成された性質であると考えられている[17]。
P. ubiqueの細胞集団の倍加速度は29時間ほどであり、かなり遅いが、低栄養条件下で培養することが可能である[18]。
ゲノム
[編集]P. ubique HTCC1062株の完全ゲノムは2005年に配列決定された[19]。知られている自由生活生物の中で最小のゲノム長(1,308,759 bp)を持ち、また自由生活生物群の中で最も少ない数となる1,354のオープンリーディングフレーム(ORF)をコードする(計1,389遺伝子)[19][20]。これより小さなゲノムを持つ系統は、Mycoplasma genitaliumやNanoarchaeum equitansなどの細胞内共生生物や寄生性生物のみが知られている[19]。20種の全アミノ酸の代謝経路や大方の補因子はコードしている[19]。一方で、イントロンやトランスポゾン、偽遺伝子などをほとんど含まず、遺伝子間スペーサー領域が短いなどコンパクトな構成をしている[19]。すなわち、細胞複製に必要なエネルギー量を削減するように、ゲノムが全体的に極めて合理化されているとみなすことができる[21]。 海洋において生物が環境中から十分に取得できずに不足しがちな窒素をできるだけ使用しないように、P. ubiqueはゲノム中においてシトシンやグアニン(CG)よりもアデニンとチミン(AT)の塩基の割合を増やしており(全塩基対の約70.3%を占める)、ゲノム複製に係るエネルギーを節約していると考えられている[21]。
公開ゲノムデータおよびメタゲノムデータのバイオインフォマティクス解析により、P. ubiqueはゲノム中に非コードRNA(ncRNA)をコードしていることが分かっている。この生物に見られるncRNAの例には、 SAM-Vリボスイッチ やrpsBモチーフのような他のシス調節エレメントが含まれている[22][23]。他に、P. ubiqueおよび他のSAR11メンバーにおける重要なncRNAとして、リンゴ酸シンターゼ上に保存されているグリシン活性化リボスイッチがある[24]。
また、P. ubiqueはプロテオロドプシン遺伝子を持っていることが知られている。プロテオロドプシンは光媒介プロトンポンプとして機能するタンパク質である。明条件処理や暗条件処理を行うと、遺伝子発現に微妙な違いが生じ、例えば暗闇にさらされると、 酸化的リン酸化に関連する遺伝子がより多く発現する[25]。
命名
[編集]属名のPelagibacterは、ラテン語の男性名詞pelagus (海)に接尾辞 -bacter(桿菌、細菌)を組み合わせたもので、「海の細菌」を意味する。最初の用語はラテン語の "pelagus"であり、語源となるギリシャ語のπέλαγος(pelagos)ではないため、接続母音は"i"であり、"o"ではない。単語pelagusはラテン語の詩で使用されるギリシャ語であり、ギリシャ語のように不規則な複数形の主格変化を伴う名詞のため、pelagiではなくpelagē となる[26]。
種名のubiqueは「どこでも」を意味するラテン語の副詞である。
Candidatusは、未記載系統で有効な学名が正式に決定されていない場合においてIBCNの 規則12c に従ってつけられる[27]。
脚注
[編集]- ^ EUZéBY, J. P. (1997-04-01). “List of Bacterial Names with Standing in Nomenclature: a Folder Available on the Internet” (英語). International Journal of Systematic and Evolutionary Microbiology 47 (2): 590–592. doi:10.1099/00207713-47-2-590. ISSN 1466-5026.
- ^ a b Rappé, M. S., et al. (2002). “Cultivation of the ubiquitous SAR11 marine bacterioplankton clade”. Nature 418 (6898): 630-3. doi:10.1038/nature00917. PMID 12167859.
- ^ a b c Michael S. Rappé; Stephanie A. Connon; Kevin L. Vergin; Stephen J. Giovannoni (2002). “Cultivation of the ubiquitous SAR11 marine bacterioplankton clade”. Nature 418 (6898): 630–633. Bibcode: 2002Natur.418..630R. doi:10.1038/nature00917. PMID 12167859.
- ^ a b Thrash, J. C.; Boyd, A.; Huggett, M. J.; Grote, J.; Carini, P.; Yoder, R. J.; Robbertse, B.; Spatafora, J. W. et al. (2011). “Phylogenomic evidence for a common ancestor of mitochondria and the SAR11 clade”. Scientific Reports 1: 13. Bibcode: 2011NatSR...1E..13T. doi:10.1038/srep00013. PMC 3216501. PMID 22355532.
- ^ Morris, Robert M.; Rappé, Michael S.; Connon, Stephanie A.; Vergin, Kevin L.; Siebold, William A.; Carlson, Craig A.; Giovannoni, Stephen J. (2002-12). “SAR11 clade dominates ocean surface bacterioplankton communities” (英語). Nature 420 (6917): 806–810. doi:10.1038/nature01240. ISSN 0028-0836.
- ^ "Candidatus Pelagibacter Ubique." European Bioinformatics Institute. European Bioinformatics Institute, 2011. Web. 08 Jan. 2012. http://www.ebi.ac.uk/2can/genomes/bacteria/Candidatus_Pelagibacter_ubique.html Archived December 1, 2008, at the Wayback Machine.
- ^ Steindler, L., et al. (2011). “Energy starved Candidatus Pelagibacter ubique substitutes light-mediated ATP production for endogenous carbon respiration”. PLoS One. 6 (5). doi:10.1371/journal.pone.0019725. PMID 21573025.
- ^ a b Zhao, X., et al. (2017). “Three-Dimensional Structure of the Ultraoligotrophic Marine Bacterium "Candidatus Pelagibacter ubique"”. Appl. Environ. Microbiol. 83 (3). doi:10.1128/AEM.02807-16. PMID 27836840.
- ^ Steindler, Laura; Schwalbach, Michael S.; Smith, Daniel P.; Chan, Francis; Giovannoni, Stephen J. (2011-05-09). “Energy Starved Candidatus Pelagibacter Ubique Substitutes Light-Mediated ATP Production for Endogenous Carbon Respiration”. PLoS ONE 6 (5). doi:10.1371/journal.pone.0019725. ISSN 1932-6203. PMC 3090418. PMID 21573025.
- ^ Stephen J. Giovannoni; H. James Tripp; Givan, S; Podar, M; Vergin, KL; Baptista, D; Bibbs, L; Eads, J et al. (2005). “Genome Streamlining in a Cosmopolitan Oceanic Bacterium”. Science 309 (5738): 1242–1245. Bibcode: 2005Sci...309.1242G. doi:10.1126/science.1114057. PMID 16109880.
- ^ “Archived copy”. 2006年3月5日時点のオリジナルよりアーカイブ。2012年2月2日閲覧。, Gauthier, Nicholas; Zinman, Guy; D’Antonio, Matteo; Abraham, Michael. Comparative Microbial Genomics DTU course. 2005.
- ^ “Flea market: A newly discovered virus may be the most abundant organism on the planet”. The Economist. (16 February 2013) 16 February 2013閲覧。
- ^ Zhao, Y.; Temperton, B.; Thrash, J. C.; Schwalbach, M. S.; Vergin, K. L.; Landry, Z. C.; Ellisman, M.; Deerinck, T. et al. (2013). “Abundant SAR11 viruses in the ocean”. Nature 494 (7437): 357–360. Bibcode: 2013Natur.494..357Z. doi:10.1038/nature11921. PMID 23407494.
- ^ Stingl, U.; Tripp, H. J.; Giovannoni, S. J. (2007). “Improvements of high-throughput culturing yielded novel SAR11 strains and other abundant marine bacteria from the Oregon coast and the Bermuda Atlantic Time Series study site”. The ISME Journal 1 (4): 361–71. doi:10.1038/ismej.2007.49. PMID 18043647.
- ^ Carini, Paul; Steindler, Laura; Beszteri, Sara; Giovannoni, Stephen J (2013-03). “Nutrient requirements for growth of the extreme oligotroph ‘Candidatus Pelagibacter ubique’ HTCC1062 on a defined medium” (英語). The ISME Journal 7 (3): 592–602. doi:10.1038/ismej.2012.122. ISSN 1751-7362. PMC 3578571. PMID 23096402.
- ^ H. James Tripp; Joshua B. Kitner; Michael S. Schwalbach; John W. H. Dacey; Larry J. Wilhelm; Stephen J. Giovannoni (April 2008). “SAR11 marine bacteria require exogenous reduced sulfur for growth”. Nature 452 (7188): 741–4. Bibcode: 2008Natur.452..741T. doi:10.1038/nature06776. PMID 18337719.
- ^ Giovannoni Lab http://giovannonilab.science.oregonstate.edu/
- ^ Giovannoni Stephen J.; Stingl Ulrich (2005). “Molecular diversity and ecology of microbial plankton”. Nature 437 (7057): 343–348. Bibcode: 2005Natur.437..343G. doi:10.1038/nature04158. PMID 16163344.
- ^ a b c d e Giovannoni, S. J. (2005-08-19). “Genome Streamlining in a Cosmopolitan Oceanic Bacterium” (英語). Science 309 (5738): 1242–1245. doi:10.1126/science.1114057. ISSN 0036-8075.
- ^ “Pelagibacter ubique genome”. NCBI. 27 November 2012閲覧。
- ^ a b “Archived copy”. 2006年3月5日時点のオリジナルよりアーカイブ。2012年2月2日閲覧。, Gauthier, Nicholas; Zinman, Guy; D’Antonio, Matteo; Abraham, Michael. Comparative Microbial Genomics DTU course. 2005.
- ^ Meyer MM; Ames TD; Smith DP; Weinberg, Zasha; Schwalbach, Michael S; Giovannoni, Stephen J; Breaker, Ronald R (2009). “Identification of candidate structured RNAs in the marine organism 'Candidatus Pelagibacter ubique'”. BMC Genomics 10: 268. doi:10.1186/1471-2164-10-268. PMC 2704228. PMID 19531245.
- ^ Poiata E; Meyer MM; Ames TD; Breaker RR (November 2009). “A variant riboswitch aptamer class for S-adenosylmethionine common in marine bacteria”. RNA 15 (11): 2046–56. doi:10.1261/rna.1824209. PMC 2764483. PMID 19776155.
- ^ H. James Tripp; Michael S. Schwalbach; Michelle M. Meyer; Joshua B. Kitner; Ronald R. Breaker; Stephen J. Giovannoni (January 2009). “Unique glycine-activated riboswitch linked to glycine-serine auxotrophy in SAR11”. Environmental Microbiology 11 (1): 230–8. doi:10.1111/j.1462-2920.2008.01758.x. PMC 2621071. PMID 19125817.
- ^ Steindler Laura; Schwalbach Michael S.; Smith Daniel P.; Chan Francis; Giovannoni Stephen J. (2011). “Energy Starved Candidatus Pelagibacter Ubique Substitutes Light-Mediated ATP Production for Endogenous Carbon Respiration”. PLOS ONE 6 (5): 9999. Bibcode: 2011PLoSO...619725S. doi:10.1371/journal.pone.0019725. PMC 3090418. PMID 21573025.
- ^ Gregory R. Crane. “pelagus entry in Perseus Digital Library”. Perseus Digital Library Project. Tufts University. 22 May 2011閲覧。
- ^ Lapage, S.; Sneath, P.; Lessel, E.; Skerman, V.; Seeliger, H.; Clark, W. (1992). International Code of Nomenclature of Bacteria: Bacteriological Code, 1990 Revision. Washington, D.C.: ASM Press. PMID 21089234
