LIG1
LIG1またはDNAリガーゼI(英: DNA ligase 1)は、ヒトではLIG1遺伝子にコードされる酵素である。DNAリガーゼIは、DNA複製と修復に関与している既知の唯一の真核生物型DNAリガーゼであり、そのため最もよく研究されているリガーゼとなっている。
発見
[編集]DNA複製がDNA二本鎖の切断を介して行われることは知られていたが、その鎖をライゲーションして元に戻す酵素や、その作用機序は不明であった。Lehman、Gellert、Richardson、Hurwitzらの研究室の大きな貢献によって、1967年になってDNAリガーゼは発見された[5]。

リクルートと調節
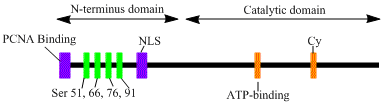
[編集]LIG1遺伝子は 120 kDa、919残基からなる、DNAリガーゼIと呼ばれる酵素をコードする。DNAリガーゼIはN末端の複製工場標的化配列(replication factory-targeting sequence、RFTS)、続いて核局在化配列、3つの機能的ドメインからなる[6]。3つの機能的ドメインはN末端側からDNA結合ドメイン(DBD)、触媒を行うヌクレオチジルトランスフェラーゼ(NTase)ドメイン、C末端のオリゴヌクレオチド/オリゴ糖結合(OB)ドメインである。N末端部分は触媒活性を持たず、細胞内での活性には必要ないが、RFTSを含んでおり複製工場(replication factory)と呼ばれるDNA複製部位へのリクルートに利用される。
DNAリガーゼIの活性化とリクルートには、翻訳後修飾が関係しているようである。N末端の4か所のセリン残基に対しリン酸化が行われ、Ser51、Ser76、Ser91のリン酸化はサイクリン依存性キナーゼ(CDK)によって、Ser66のリン酸化はカゼインキナーゼ2(CK2)によってそれぞれ行われる。Rossiらは、Ser66が脱リン酸化されているときにRFTSはPCNAと相互作用すると提唱しており[7]、Tomらによってin vitroでの確証が行われている[8]。どちらのデータもDNAリガーゼIのN末端領域がin vivoで核内での酵素機能を調節する役割を果たすことに対する妥当なエビデンス提供している[7][9]。さらに、触媒を行うC末端ドメインにはサイクリン結合モチーフ(Cyモチーフ)が同定されており、変異体解析からSer76とSer91のリン酸化に関与していることが示された。N末端の複数のセリンがCDKとCK2の基質となり、DNAリガーゼIが細胞周期のS期に複製工場へリクルートされた際にRFTSとPCNAとの相互作用を調節しているようである[6][7][10]。
機能と機構
[編集]DNAリガーゼIは、DNA複製と塩基除去修復過程で機能する[11]。
真核生物のDNAリガーゼIが触媒する反応は、化学的にはすべてのリガーゼと共通である。DNA複製と修復の双方で、DNAリガーゼIはエネルギー的に有利なライゲーション反応を行うために、アデノシン三リン酸(ATP)を利用する。DNA複製は、真核生物の細胞周期のS期の間に起こる。DNAリガーゼIは、DNAのラギング鎖でDNAポリメラーゼδによってRNAプライマーヌクレオチドがDNAヌクレオチドに置き換えられた後、非連続的なDNA合成によって形成された岡崎フラグメントの連結を担う。岡崎フラグメントのライゲーションが適切に行われずニックを含むDNAでは、容易に二本鎖切断が起こり、遺伝的変異が引き起こされる可能性がある。これらのフラグメントのライゲーションは、3つの段階を経て進行する。
- 酵素へのアデノシン一リン酸(AMP)基の付加(アデニリル化と呼ばれる)
- AMPのDNAへの転移
- ホスホジエステル結合の形成によってニックを閉じる(ニックシーリング)[9][12]

アデニリル化の際、ATPのαリン酸基は触媒リジン残基からの求核攻撃を受け、DNAリガーゼIの活性部位のリジンとAMPが共有結合した中間体と無機ピロリン酸が形成される。
AMPの転移段階では、DNAリガーゼIはDNAと結合してニック部分に位置し、ニックの5'-リン酸部位での反応を触媒する。ニックの5'-リン酸のアニオン性酸素が求核剤として機能し、リジンに共有結合しているAMPのαリン酸基を攻撃し、AMPがDNAに共有結合した中間体(DNA-AMP中間体)が形成される。
ホスホジエステル結合を形成するためには、DNA-AMP中間体は除去されなければならない。5'-リン酸基は上流の3'-OH基からの求核攻撃を受け、それによってホスホジエステル結合が形成される。この求核攻撃の間、AMP基は5'-リン酸基を脱離基として押し出し、ニックは閉じられてAMPは解離し、DNAライゲーションの1サイクルが完結する。
最適では無い条件下では、反応が完結する前にリガーゼがDNAから解離してしまう場合がある。例えばマグネシウム濃度が低い条件下では、ニックシーリングの過程が遅くなり、リガーゼはアデニリル化中間体を残してDNAから解離してしまう。こうした中間体はホスホジエステラーゼの助けを借りなければ修復できない。ホスホジエステラーゼのアプラタキシンは、こうした中断されたDNA中間体に作用してAMP-リン酸結合を加水分解し、リガーゼが反応する前の初期状態を回復することが示されている[13][14]。
損傷塩基修復における役割
[編集]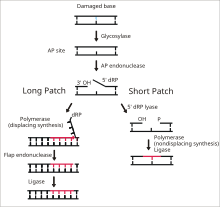
DNAリガーゼIは、塩基除去修復(BER)経路の最終段階で、一本鎖DNA切断のライゲーションを行う[15]。DNAの窒素含有塩基が活性酸素種、毒素、電離放射線などの環境の危険因子によって損傷することはよく起こる。BERは損傷した塩基の除去と置換を担う主要な修復経路である。リガーゼIはlong patch BER(LP-BER)経路に関与しており、一方リガーゼIIIはshort patch BER(single nucleotide BER、SN-BER)経路に関与している[16]。LP-BERは4つの段階を経て進行する。まず、DNAグリコシラーゼがN-グリコシド結合を切断して損傷塩基を解離させ、プリンまたはピリミジン塩基が存在しないAP部位が作り出される。次の段階では、APエンドヌクレアーゼがAP部位の5'末端側にニックを形成し、AP部位はデオキシリボースリン酸(dRP)残基となる。LP-BER経路ではその後、DNAポリメラーゼが5'から3'方向へ新たな塩基をいくつか合成し、5'末端にdRPが存在するDNAフラップが形成される。このフラップはフラップエンドヌクレアーゼによって切断される。この切断によってニックを含むDNA鎖が残され、DNAリガーゼIによる検知とライゲーションが行われる[15][16][17]。リガーゼIの作用は、LP-BER経路の他の酵素、特にAPエンドヌクレアーゼとDNAポリメラーゼによって促進される[17]。
臨床的意義
[編集]DNAリガーゼI欠乏を引き起こすLIG1の変異は、免疫不全やDNA損傷剤に対する感受性の増加につながる[11]。
DNAリガーゼI欠乏を示す患者の確定症例は1つだけ存在しており、遺伝性の変異アレルによるものである。この欠乏症の症状は発育不全と免疫不全である。患者由来の細胞株に基づいて作製されたマウスモデルでは、変異体リガーゼによってゲノム不安定性につながる複製エラーが生じることが確認された。特に、変異体マウスでは腫瘍形成の増加も示された[9]。
リガーゼIは、良性腫瘍細胞や正常細胞ではなく、増殖中の腫瘍細胞でアップレギュレーションされていることも判明している。さらに、これらの細胞でリガーゼIの発現を阻害すると細胞毒性効果が生じることが示されており、リガーゼI阻害剤の化学療法薬としての可能性が示唆される[18]。
DNAリガーゼIが反応を中断した際のアデニリル化DNAの除去を担うホスホジエステラーゼであるアプラタキシンの欠損は、神経変性と関係している。このことは、DNAはリガーゼのエラーを修正する他のバックアップ機構がなければ再び修復経路へ入ることができないことを示唆している[14]。
DNAの構造はよく解明され、また、その操作、修復、利用に必要な構成要素の多くが同定され、特徴づけられているため、病気を治療したり、がんと闘ったり、生物学的刺激に基づいて薬剤を放出したりする能力を持つナノスケールの装置の開発の検討が行われている。DNAリガーゼは、こうした装置に組み込まれる可能性が高い[19]。
出典
[編集]- ^ a b c GRCh38: Ensembl release 89: ENSG00000105486 - Ensembl, May 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000056394 - Ensembl, May 2017
- ^ Human PubMed Reference:
- ^ Mouse PubMed Reference:
- ^ “Insights into DNA Joining: I. Robert Lehman's work on DNA Ligase”. Journal of Biological Chemistry 282 (2): e1. (January 2007).
- ^ a b “Cell cycle-dependent phosphorylation of human DNA ligase I at the cyclin-dependent kinase sites”. J. Biol. Chem. 278 (39): 37761–7. (September 2003). doi:10.1074/jbc.M304462200. PMID 12851383.
- ^ a b c “The replication factory targeting sequence/PCNA-binding site is required in G(1) to control the phosphorylation status of DNA ligase I”. EMBO J. 18 (20): 5745–54. (October 1999). doi:10.1093/emboj/18.20.5745. PMC 1171641. PMID 10523317.
- ^ Tom, S.; Henricksen, L. A.; Park, M. S.; Bambara, R. A. (2001-07-06). “DNA ligase I and proliferating cell nuclear antigen form a functional complex”. The Journal of Biological Chemistry 276 (27): 24817–24825. doi:10.1074/jbc.M101673200. ISSN 0021-9258. PMID 11331287.
- ^ a b c “Eukaryotic DNA ligases: structural and functional insights”. Annu. Rev. Biochem. 77: 313–38. (2008). doi:10.1146/annurev.biochem.77.061306.123941. PMC 2933818. PMID 18518823.
- ^ “Activation of mammalian DNA ligase I through phosphorylation by casein kinase II”. EMBO J. 11 (8): 2925–33. (August 1992). doi:10.1002/j.1460-2075.1992.tb05362.x. PMC 556774. PMID 1639065.
- ^ a b “Entrez Gene: LIG1 ligase I, DNA, ATP-dependent”. 2020年5月6日閲覧。
- ^ “Chlorella virus DNA ligase: nick recognition and mutational analysis”. Nucleic Acids Res. 26 (2): 525–31. (January 1998). doi:10.1093/nar/26.2.525. PMC 147278. PMID 9421510.
- ^ “Kinetic mechanism of human DNA ligase I reveals magnesium-dependent changes in the rate-limiting step that compromise ligation efficiency”. J. Biol. Chem. 286 (26): 23054–62. (July 2011). doi:10.1074/jbc.M111.248831. PMC 3123073. PMID 21561855.
- ^ a b “Actions of aprataxin in multiple DNA repair pathways”. J. Biol. Chem. 282 (13): 9469–74. (March 2007). doi:10.1074/jbc.M611489200. PMID 17276982.
- ^ a b “Long-patch DNA repair synthesis during base excision repair in mammalian cells”. EMBO Rep. 4 (4): 363–7. (April 2003). doi:10.1038/sj.embor.embor796. PMC 1319152. PMID 12671676.
- ^ a b “Early steps in the DNA base excision/single-strand interruption repair pathway in mammalian cells”. Cell Res. 18 (1): 27–47. (January 2008). doi:10.1038/cr.2008.8. PMC 2692221. PMID 18166975.
- ^ a b “Long patch base excision repair proceeds via coordinated stimulation of the multienzyme DNA repair complex”. J. Biol. Chem. 284 (22): 15158–72. (May 2009). doi:10.1074/jbc.M109.000505. PMC 2685697. PMID 19329425.
- ^ “Elevated expression of DNA ligase I in human cancers”. Clin. Cancer Res. 7 (12): 4143–8. (December 2001). PMID 11751514.
- ^ Macdonald, Joanne. “Smart DNA: Programming the Molecule of Life for Work and Play [Preview]”. scientificamerican. 2013年2月22日閲覧。
関連文献
[編集]- “Targeting and association of proteins with functional domains in the nucleus: the insoluble solution.”. Int. Rev. Cytol.. International Review of Cytology 162B: 303–35. (1996). doi:10.1016/S0074-7696(08)62620-0. ISBN 9780123645661. PMID 8557490.
- “Structure and function of mammalian DNA ligases.”. Mutat. Res. 407 (1): 1–9. (1998). doi:10.1016/s0921-8777(97)00050-5. PMID 9539976.
- “[Paralysis following Dimer X radiculography]”. La Nouvelle Presse Médicale 5 (17): 1120–2. (1976). PMID 934827.
- “Growth retardation and immunodeficiency in a patient with mutations in the DNA ligase I gene.”. Lancet 339 (8808): 1508–9. (1992). doi:10.1016/0140-6736(92)91266-B. PMID 1351188.
- “Mutations in the DNA ligase I gene of an individual with immunodeficiencies and cellular hypersensitivity to DNA-damaging agents.”. Cell 69 (3): 495–503. (1992). doi:10.1016/0092-8674(92)90450-Q. PMID 1581963.
- “Assignment of the gene encoding DNA ligase I to human chromosome 19q13.2-13.3.”. Genomics 12 (1): 164–6. (1992). doi:10.1016/0888-7543(92)90422-O. PMID 1733856.
- “A wild-type DNA ligase I gene is expressed in Bloom's syndrome cells.”. Proc. Natl. Acad. Sci. U.S.A. 88 (17): 7615–9. (1991). doi:10.1073/pnas.88.17.7615. PMC 52352. PMID 1881902.
- “Mammalian DNA ligases. Biosynthesis and intracellular localization of DNA ligase I.”. J. Biol. Chem. 265 (21): 12618–22. (1990). PMID 2197279.
- “Human DNA ligase I cDNA: cloning and functional expression in Saccharomyces cerevisiae.”. Proc. Natl. Acad. Sci. U.S.A. 87 (17): 6679–83. (1990). doi:10.1073/pnas.87.17.6679. PMC 54600. PMID 2204063.
- “The N-terminal domain of human DNA ligase I contains the nuclear localization signal and directs the enzyme to sites of DNA replication.”. EMBO J. 14 (21): 5379–86. (1996). doi:10.1002/j.1460-2075.1995.tb00222.x. PMC 394647. PMID 7489727.
- “Oligo-capping: a simple method to replace the cap structure of eukaryotic mRNAs with oligoribonucleotides.”. Gene 138 (1–2): 171–4. (1994). doi:10.1016/0378-1119(94)90802-8. PMID 8125298.
- “Fluorescence in situ hybridization mapping of human chromosome 19: cytogenetic band location of 540 cosmids and 70 genes or DNA markers.”. Genomics 15 (1): 133–45. (1993). doi:10.1006/geno.1993.1021. PMID 8432525.
- “Isolation and characterization of the human MRE11 homologue.”. Genomics 29 (1): 80–6. (1996). doi:10.1006/geno.1995.1217. PMID 8530104.
- “DNA ligase I is required for fetal liver erythropoiesis but is not essential for mammalian cell viability.”. Nat. Genet. 13 (4): 489–91. (1996). doi:10.1038/ng0896-489. PMID 8696349.
- “Construction and characterization of a full length-enriched and a 5'-end-enriched cDNA library.”. Gene 200 (1–2): 149–56. (1997). doi:10.1016/S0378-1119(97)00411-3. PMID 9373149.
- “The replication factory targeting sequence/PCNA-binding site is required in G(1) to control the phosphorylation status of DNA ligase I.”. EMBO J. 18 (20): 5745–54. (1999). doi:10.1093/emboj/18.20.5745. PMC 1171641. PMID 10523317.
- “Reconstitution of proliferating cell nuclear antigen-dependent repair of apurinic/apyrimidinic sites with purified human proteins.”. J. Biol. Chem. 274 (47): 33703–8. (1999). doi:10.1074/jbc.274.47.33703. PMID 10559261.
- “DNA repair patch-mediated double strand DNA break formation in human cells.”. J. Biol. Chem. 275 (35): 27386–92. (2000). doi:10.1074/jbc.M003126200. PMID 10827190.






